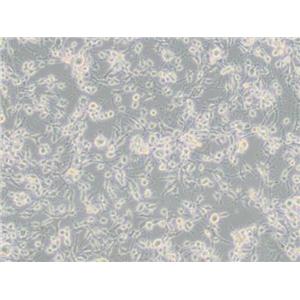
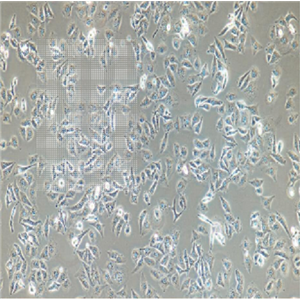
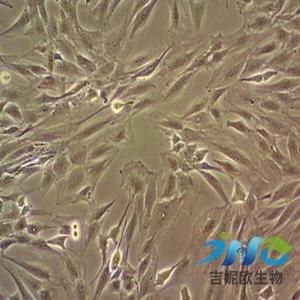
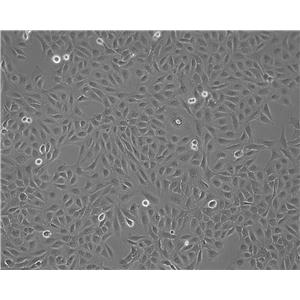
"MDA-MB-436人乳腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的应用:1)免疫组化研究2)RNA干扰研究3)药物作用研究4)慢病毒转染研究等其它应用。细胞系通常用于实验研究,如增殖、迁移、侵袭等。细胞系在多个领域的研究中被广泛应用,包括基础医学、临床试验、药物筛选和分子生物学研究。这些研究不仅在中国,也在日本、美国和欧洲等多个国家和地区进行。
换液周期:每周2-3次
CCD-841-CoTr Cells;背景说明:结肠癌;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MEF (CF-1)细胞、LS513细胞、HCoEpiC细胞
NR8383.1 Cells;背景说明:NR8383(正常大鼠,1983年8月3日)来源于肺灌洗时的正常大鼠肺泡巨噬细胞。细胞在gerbil肺细胞连续培养液存在下培养了大约8-9个月。随后,不再需要外源生长因子。通过有限稀释法从单个细胞克隆并亚克隆NR8383细胞,并三次用软琼脂亚克隆。细胞表现出巨噬细胞的特性,吞噬酵母多糖和铜绿,非特异性脂酶活性,Fc受体,氧化降解;分泌IL-1,TNFbeta和IL-6,可重复地响应外源生长因子。NR8383细胞响应博莱霉素,分泌TGFbeta前体。在博莱霉素刺激下,TGFbe;传代方法:1:2传代;生长特性:半贴壁生长;形态特性:巨噬细胞;相关产品有:NCIH520细胞、COC1/CDDP细胞、A2058细胞
HEK 293T/17 Cells;背景说明:胚肾;5型腺病毒及SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKML-28细胞、FHs 74 Int细胞、BHT-101细胞
MDA-MB-436人乳腺癌细胞代次低|培养基|送STR图谱
背景信息:该细胞源于一名43岁患有乳腺腺癌女性的胸腔积。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
公司细胞系主要引进ATCC、DSMZ、JCRB、KCLB、RIKEN、ECACC等细胞库,细胞系体外培养,它们会成长为单层细胞,附着或紧贴在培养瓶上,或悬浮在体外的溶液中,细胞系复苏周期短,公司细胞系状态良好,饱满,有光泽等优点。EDTA的作用:许多人不用胰酶,只用EDTA,或者用胰酶/EDTA联合作用。这里要明白,胰酶切割细胞外基质的一些负责粘连和附着的蛋白,而EDTA通过螯合Ca离子,作用于整联蛋白的活性,所以EDTA的作用更加温和。有的人在胰酶里添加一些EDTA,或者对付特别难消化的细胞,添加多一些EDTA,就是这个道理。一般不要试图延长消化时间(如果10min还消化不下来的话),而应该想其它办法。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
MDA-MB-436人乳腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
PL-12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MOLT-3细胞、NCIH747细胞、NIE-115细胞
RT4P Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:多角 ;相关产品有:Hep 3B2细胞、DMS153细胞、FRhK-4细胞
NCI-SNU-C2A Cells;背景说明:详见相关文献介绍;传代方法:每周两次换液;生长特性:松散附着、多单元的聚合;形态特性:上皮细胞样;相关产品有:SU 86.86细胞、TE4细胞、TE4细胞
SU.86 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:TCam-2细胞、IGROV1细胞、HPAF-I细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
贴壁细胞的消化方法介绍:1、胰酶。这是用得Zui多的。一般浓度在0.25-0.5%。作用时间根据细胞种类、作用温度等因素而变化很大,从几分钟到几十分钟不等。0.25%的胰酶作用于单层贴壁的细胞,在37度条件下,一般消化1-5分钟就足够了。终止是用血清。主要作用于细胞间。配制时不能用含、镁的平衡,否则影响活性。保存于-20度。2、胶原酶。这种方法比较少,一般是用原代培养时,从组织消化下细胞。这种方法作用温和,对细胞损伤较小,但是,价格也较贵。中止同样是用血清。3、EDA。用得也是非常多。一般浓度在0.02%左右。作用于细胞与间质,对细胞间也有一定作用。注意,它能显著影响pH值,而且在弱碱性条件下才易溶。因此,配制时应调节HAO碱度。它不能被终和。因此,消化下来的细胞要洗一遍。4、商品化的无酶消化。个人的使用经常觉得对细胞的损伤比较大,但是分离成单细胞悬的能力确实比较强。5、物理法。直接吹打或用细胞刮子将细胞刮下来。6、冷冻法。此方法仅能用于细胞传代时。无法使组织上的细胞脱落下来。本方法的原理,我想是因细胞冷冻后收缩,从而从培养瓶上脱落下来。YOU点是:对细胞损伤小,不需要中止或洗细胞,方便,不需要另外配制消化。别适用那些贴壁不是别紧,又别娇气的细胞。不足是细胞常成小片脱落。此种方法曾用于因用其它方法传代导致大量细胞死亡操作的间充质干细胞、DC细胞的培养,效果非常满意。具体过程是:1、用较多的4度的PBS 洗涤一遍细胞(以6孔板为例,加1.5ml/孔),2、再加0.5毫升4度的PBS,静置操作台上,很快细胞就小片脱落,3、轻轻吹打,细胞即完全脱落,4、按一定比例传代。
C643 Cells;背景说明:甲状腺未分化癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:VCaP细胞、CL1细胞、Hepa1-6细胞
3T3-442A Cells;背景说明:脂肪前体细胞;雄性;Swiss albino;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-MEL-MeWo细胞、PANC 327细胞、RKO-AS45-1细胞
SKLU01 Cells;背景说明:该细胞系源于一位60岁的白人女性患者的肺腺癌组织。;传代方法:1:2传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:WM 239细胞、MM-1S细胞、ATDC-5细胞
SUDHL4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:3T3-Swiss albino细胞、MPC-11细胞、GM0637细胞
MCCAR Cells;背景说明:B淋巴;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:TGHAVSMC细胞、U266 B1细胞、H1437细胞
KMS18 Cells;背景说明:浆细胞骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:NS20-Y细胞、UCLA NPA-87-1细胞、JHH-7细胞
NCIH526 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长 ;形态特性:上皮细胞;相关产品有:Panc8.13细胞、WERI Rb-1细胞、ATC241细胞
Hs870T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代;每周换液2-3次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:JG细胞、NCI-H748细胞、SK-MEL-1细胞
CORL279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MILE SVEN1细胞、SR细胞、U87 MG细胞
P3-NS1/1-Ag4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:NHLF细胞、NCI-H524细胞、Caco 2细胞
CF-PAC1 Cells;背景说明:详见相关文献介绍;传代方法:1:3-10传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCIH2452细胞、P30/Ohkubo细胞、NCC-IT细胞
PANC0504 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:ATN1细胞、HIT细胞、SK-MEL-24细胞
DHBE Cells;背景说明:支气管;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW900细胞、Suzhou Human Glioma-44细胞、Hep 3B2_1-7细胞
H3255_DA Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HCS2/8细胞、KPL-4细胞、OCI Ly3细胞
RDES Cells;背景说明:详见相关文献介绍;传代方法:1:8传代:每周换液2-3次;生长特性:贴壁和悬浮混合;形态特性:上皮样;相关产品有:RC-4B/C细胞、CD 18细胞、BT 549细胞
CD-18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:B16 melanoma F10细胞、NCIH1573细胞、RAEC细胞
LS-411 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H1693细胞、MC3T3-E1 Subclone 14细胞、OVCA433_Bast细胞
Abcam A-549 C1QBP KO Cells(提供STR鉴定图谱)
Abcam Raji TDP1 KO Cells(提供STR鉴定图谱)
BayGenomics ES cell line BGC099 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRS110 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA370 Cells(提供STR鉴定图谱)
CHEF/18 Cells(提供STR鉴定图谱)
DA01875 Cells(提供STR鉴定图谱)
DHMi005-A-5 Cells(提供STR鉴定图谱)
GM02831 Cells(提供STR鉴定图谱)
TGW-nu Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:神经元细胞;相关产品有:SKNEP细胞、OCI Ly10细胞、TE-85细胞
MDA-MB-436人乳腺癌细胞代次低|培养基|送STR图谱
HEL-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SCC 4细胞、Adeno-293细胞、H2052细胞
MF2059 Cells;背景说明:皮肤;T淋巴细胞瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:WM266mel细胞、HBL 100细胞、SU.86细胞
H128 Cells;背景说明:小细胞肺癌;胸腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Caco2BBe细胞、MBVP细胞、MGH-U3 (RN)细胞
HEK293T/17 Cells;背景说明:胚肾;5型腺病毒及SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKNSH细胞、University of Arizona Cell Culture-893细胞、SUM149PT细胞
C3H-10T1/2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MUTZ-3细胞、ATN1细胞、Hs 281.T细胞
5-2 Cells(提供STR鉴定图谱)
Vero 76 clone E-6 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SK HEP 01细胞、JEKO细胞、SKNSH细胞
RPMI-7666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T HEECs细胞、NTERA-2/D1细胞、Hs281T细胞
SHEP1 Cells;背景说明:神经母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKRC-39细胞、Panc-08.13细胞、Hs940T细胞
COLO320DM Cells;背景说明:该细胞可产生5-羟色胺、去甲、、ACTH和甲状旁腺素。角蛋白、波形蛋白弱阳性。培养条件: RPMI 1640 10%FBS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮+贴壁;形态特性:淋巴细胞;相关产品有:GC-1spg细胞、HFF细胞、HEL 92.1.7细胞
Rat Basophilic Leukemia-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Sp2/O-Ag14细胞、PG-LH7细胞、CAL-27细胞
UO-31 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Human Kidney-2细胞、PC2细胞、MC-3T3细胞
UO-31 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Human Kidney-2细胞、PC2细胞、MC-3T3细胞
H22-H8D8 Cells;背景说明:肝癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:CHL1细胞、NCI-H69C细胞、HS-294-T细胞
GM10439 Cells(提供STR鉴定图谱)
HAP1 CCDC6 (-) 3 Cells(提供STR鉴定图谱)
MZCRC1 Cells;背景说明:甲状腺髓样癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PNT-1A细胞、YD15细胞、HLF-a细胞
BE2-M17 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO680N细胞、CDC/EU.HMEC-1细胞、SW1222细胞
HLE-B3 Cells;背景说明:晶状体;Ad12-SV40转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:2PK-3细胞、HTSMC细胞、RPE-hTERT细胞
H-498 Cells;背景说明:结肠腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:NHRF细胞、SNU368细胞、COLO 320 DM细胞
HEC-151 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HepG2-luc细胞、H358细胞、T2 (174 x CEM.T2)细胞
TGW Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:神经元细胞;相关产品有:Clone Y-1细胞、ARO细胞、RCC-JF细胞
Calu3 Cells;背景说明:该细胞是从一名25岁的白人男性肺腺癌患者的胸水中分离建立的;该患者曾使用过环磷酰胺、博来霉素、阿霉素进行治疗。该细胞接种至裸鼠可成瘤;可作转染宿主。;传代方法:消化20分钟。1:2。5-6天长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:PY8119细胞、CV1细胞、TM3细胞
Human Hepatocyte Line 5 Cells;背景说明:肝;HPV16转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO206F细胞、NB.4细胞、Huh7.5细胞
HG03790 Cells(提供STR鉴定图谱)
IHL,AD036 Cells(提供STR鉴定图谱)
LSCC-RP12 Cells(提供STR鉴定图谱)
ND00355 Cells(提供STR鉴定图谱)
PC-R1 Cells(提供STR鉴定图谱)
Ubigene HeLa RHOT2 KO Cells(提供STR鉴定图谱)
XP39R Cells(提供STR鉴定图谱)
HAP1 TERF2IP (-) Cells(提供STR鉴定图谱)
H-1436 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:PNT1/A细胞、SUM-52-PE细胞、Hca-F细胞
MDA-MB436 Cells;背景说明:该细胞源于一名43岁患有乳腺腺癌女性的胸腔积液。;传代方法:1:2传代,每周换液2—3次;生长特性:贴壁生长;形态特性:多角形;相关产品有:LA-795细胞、X63Ag8.653细胞、Colon 38细胞
NCTC1469 Cells;背景说明:1952年建系,源于正常C3H/An小鼠的肝脏组织,表达H-2K抗原,鼠痘病毒阴性。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:淋巴母细胞;相关产品有:SkChA1细胞、RL952细胞、Madison 109细胞
QGY 7701 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI H23细胞、HOPC细胞、MS1细胞
SUM-159 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1341细胞、HS5细胞、JurkatE6-1细胞
SUM-159 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1341细胞、HS5细胞、JurkatE6-1细胞
CRFK Cells;背景说明:肾;自发永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TCC-PAN2细胞、JVM3细胞、NCI.H522细胞
HRA-19 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H441细胞、aTC1-6细胞、HL 60细胞
Virginia Mason Research Center-Renal Cancer Z Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MDA-PCa-2b细胞、HT-22细胞、293FT细胞
MSC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CALU1细胞、GT38细胞、ARPE-19细胞
SUM149-PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MHCC-LM3细胞、Vero E-6细胞、NOMO-1细胞
SUM 52PE Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OKa-C-1细胞、HT 1376细胞、NCI-H2198细胞
CACO2 Cells;背景说明:细胞株分离自一个原发性结肠癌。当细胞长满时,表现出典型的肠细胞分化的特征。Caco-2细胞表达维生素A酸结合蛋白I和视黄醇结合蛋白II,角蛋白阳性。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:PCI:SG231细胞、HFB细胞、GBCSD细胞
HGF-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PAN 02细胞、SNU-869细胞、JIMT-1细胞
Panc_03_27 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:A2780/Taxol细胞、Strain KB细胞、GP293细胞
Scid.adh.6D4 Cells(提供STR鉴定图谱)
SNU-878 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HR8348细胞、OCI-Ly 1细胞、NCI-SNU-878细胞
Hs 343.T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TN5B14细胞、HKC细胞、V79-1细胞
COLO678 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P30 OHK细胞、PANC403细胞、H9细胞
MDAPCa-2b Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:TOV-112D细胞、U-373MG细胞、HS-683细胞
HC11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MM6细胞、HEC-251细胞、NUGC4细胞
SuDHL 1 Cells;背景说明:间变性大细胞淋巴瘤;胸腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HSKMC细胞、BE2M17细胞、NCIH3255细胞
MDA-MB-436人乳腺癌细胞代次低|培养基|送STR图谱
Evsa T Cells;背景说明:乳腺癌;腹水转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SF-539细胞、U138细胞、IOSE 80细胞
HEI193 Cells;背景说明:神经鞘瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DLD1细胞、138MG细胞、UMNSAH/DF-1细胞
JROECL 19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BI-Mel细胞、KRC Y细胞、HBSMC细胞
P30OHK Cells;背景说明:详见相关文献介绍;传代方法:10^5 cells/60mm dish;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Rat-2细胞、H69/P细胞、SK-ES-1细胞
TE354.T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:S.B.细胞、KU 812F细胞、HKC细胞
H-EMC-SS Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:BSC-40细胞、PANC 813细胞、SK-MEL 28细胞
Hs1.Tes Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:OVCAR-3细胞、NH-6细胞、Jurkat clone A3细胞
EAC Cells;背景说明:艾氏腹水瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:NCIH1623细胞、Hs274T细胞、H-748细胞
BayGenomics ES cell line RRB168 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XE404 Cells(提供STR鉴定图谱)
Cx43KOM104-E6(-/-) Cells(提供STR鉴定图谱)
mHippoE-2 Cells(提供STR鉴定图谱)
Sq-1979-2 Cells(提供STR鉴定图谱)
LS180-SLC2A4-KO-c7 Cells(提供STR鉴定图谱)
" "PubMed=1961733; DOI=10.1073/pnas.88.23.10657; PMCID=PMC52989
Runnebaum I.B., Nagarajan M., Bowman M., Soto D., Sukumar S.
Mutations in p53 as potential molecular markers for human breast cancer.
Proc. Natl. Acad. Sci. U.S.A. 88:10657-10661(1991)
DOI=10.1016/B978-0-12-333530-2.50009-5
Leibovitz A.
Cell lines from human breast.
(In book chapter) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.161-184; Academic Press; New York; USA (1994)
PubMed=9288768
Ahmadian M., Wistuba I.I., Fong K.M., Behrens C., Kodagoda D.R., Saboorian M.H., Shay J.W., Tomlinson G.E., Blum J.L., Minna J.D., Gazdar A.F.
Analysis of the FHIT gene and FRA3B region in sporadic breast cancer, preneoplastic lesions, and familial breast cancer probands.
Cancer Res. 57:3664-3668(1997)
PubMed=9823299
Khan J., Simon R.M., Bittner M., Chen Y.-D., Leighton S.B., Pohida T., Smith P.D., Jiang Y., Gooden G.C., Trent J.M., Meltzer P.S.
Gene expression profiling of alveolar rhabdomyosarcoma with cDNA microarrays.
Cancer Res. 58:5009-5013(1998)
PubMed=10862037; DOI=10.1002/1098-2264(200007)28:3<308::AID-GCC9>3.0.CO;2-B
Kytola S., Rummukainen J., Nordgren A., Karhu R., Farnebo F., Isola J.J., Larsson C.
Chromosomal alterations in 15 breast cancer cell lines by comparative genomic hybridization and spectral karyotyping.
Genes Chromosomes Cancer 28:308-317(2000)
PubMed=10969801
Forozan F., Mahlamaki E.H., Monni O., Chen Y.-D., Veldman R., Jiang Y., Gooden G.C., Ethier S.P., Kallioniemi A.H., Kallioniemi O.-P.
Comparative genomic hybridization analysis of 38 breast cancer cell lines: a basis for interpreting complementary DNA microarray data.
Cancer Res. 60:4519-4525(2000)
PubMed=11343771; DOI=10.1016/S0165-4608(00)00387-3
Rummukainen J., Kytola S., Karhu R., Farnebo F., Larsson C., Isola J.J.
Aberrations of chromosome 8 in 16 breast cancer cell lines by comparative genomic hybridization, fluorescence in situ hybridization, and spectral karyotyping.
Cancer Genet. Cytogenet. 126:1-7(2001)
PubMed=15677628; DOI=10.1093/carcin/bgi032
Gorringe K.L., Chin S.-F., Pharoah P.D.P., Staines J.M., Oliveira C., Edwards P.A.W., Caldas C.
Evidence that both genetic instability and selection contribute to the accumulation of chromosome alterations in cancer.
Carcinogenesis 26:923-930(2005)
PubMed=16397213; DOI=10.1158/0008-5472.CAN-05-2853
Elstrodt F., Hollestelle A., Nagel J.H.A., Gorin M., Wasielewski M., van den Ouweland A.M.W., Merajver S.D., Ethier S.P., Schutte M.
BRCA1 mutation analysis of 41 human breast cancer cell lines reveals three new deleterious mutants.
Cancer Res. 66:41-45(2006)
PubMed=16541312; DOI=10.1007/s10549-006-9186-z
Wasielewski M., Elstrodt F., Klijn J.G.M., Berns E.M.J.J., Schutte M.
Thirteen new p53 gene mutants identified among 41 human breast cancer cell lines.
Breast Cancer Res. Treat. 99:97-101(2006)
PubMed=17157791; DOI=10.1016/j.ccr.2006.10.008; PMCID=PMC2730521
Neve R.M., Chin K., Fridlyand J., Yeh J., Baehner F.L., Fevr T., Clark L., Bayani N., Coppe J.-P., Tong F., Speed T., Spellman P.T., DeVries S., Lapuk A., Wang N.J., Kuo W.-L., Stilwell J.L., Pinkel D., Albertson D.G., Waldman F.M., McCormick F., Dickson R.B., Johnson M.D., Lippman M.E., Ethier S.P., Gazdar A.F., Gray J.W.
A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes.
Cancer Cell 10:515-527(2006)
PubMed=18516279; DOI=10.1016/j.molonc.2007.02.004; PMCID=PMC2391005
Kenny P.A., Lee G.Y., Myers C.A., Neve R.M., Semeiks J.R., Spellman P.T., Lorenz K., Lee E.H., Barcellos-Hoff M.H., Petersen O.W., Gray J.W., Bissell M.J.
The morphologies of breast cancer cell lines in three-dimensional assays correlate with their profiles of gene expression.
Mol. Oncol. 1:84-96(2007)
PubMed=19582160; DOI=10.1371/journal.pone.0006146; PMCID=PMC2702084
Kao J., Salari K., Bocanegra M., Choi Y.-L., Girard L., Gandhi J., Kwei K.A., Hernandez-Boussard T., Wang P., Gazdar A.F., Minna J.D., Pollack J.R.
Molecular profiling of breast cancer cell lines defines relevant tumor models and provides a resource for cancer gene discovery.
PLoS ONE 4:E6146-E6146(2009)
DOI=10.25904/1912/1434
Morrison B.J.
Breast cancer stem cells: tumourspheres and implications for therapy.
Thesis PhD (2010); Griffith University; Brisbane; Australia
PubMed=19593635; DOI=10.1007/s10549-009-0460-8
Hollestelle A., Nagel J.H.A., Smid M., Lam S., Elstrodt F., Wasielewski M., Ng S.S., French P.J., Peeters J.K., Rozendaal M.J., Riaz M., Koopman D.G., ten Hagen T.L.M., de Leeuw B.H.C.G.M., Zwarthoff E.C., Teunisse A.F.A.S., van der Spek P.J., Klijn J.G.M., Dinjens W.N.M., Ethier S.P., Clevers H.C., Jochemsen A.G., den Bakker M.A., Foekens J.A., Martens J.W.M., Schutte M.
Distinct gene mutation profiles among luminal-type and basal-type breast cancer cell lines.
Breast Cancer Res. Treat. 121:53-64(2010)
PubMed=21778573; DOI=10.3233/BD-2010-0307; PMCID=PMC3532890
Chavez K.J., Garimella S.V., Lipkowitz S.
Triple negative breast cancer cell lines: one tool in the search for better treatment of triple negative breast cancer.
Breast Dis. 32:35-48(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
PubMed=23151021; DOI=10.1186/1471-2164-13-619; PMCID=PMC3546428
Grigoriadis A., Mackay A., Noel E., Wu P.-J., Natrajan R., Frankum J., Reis-Filho J.S., Tutt A.
Molecular characterisation of cell line models for triple-negative breast cancers.
BMC Genomics 13:619.1-619.14(2012)
PubMed=23601657; DOI=10.1186/bcr3415; PMCID=PMC3672661
Riaz M., van Jaarsveld M.T.M., Hollestelle A., Prager-van der Smissen W.J.C., Heine A.A.J., Boersma A.W.M., Liu J.-J., Helmijr J.C.A., Ozturk B., Smid M., Wiemer E.A.C., Foekens J.A., Martens J.W.M.
miRNA expression profiling of 51 human breast cancer cell lines reveals subtype and driver mutation-specific miRNAs.
Breast Cancer Res. 15:R33.1-R33.17(2013)
PubMed=24094812; DOI=10.1016/j.ccr.2013.08.020; PMCID=PMC3931310
Timmerman L.A., Holton T., Yuneva M., Louie R.J., Padro M., Daemen A., Hu M., Chan D.A., Ethier S.P., van 't Veer L.J., Polyak K., McCormick F., Gray J.W.
Glutamine sensitivity analysis identifies the xCT antiporter as a common triple-negative breast tumor therapeutic target.
Cancer Cell 24:450-465(2013)
PubMed=24162158; DOI=10.1007/s10549-013-2743-3; PMCID=PMC3832776
Prat A., Karginova O., Parker J.S., Fan C., He X.-P., Bixby L.M., Harrell J.C., Roman E., Adamo B., Troester M.A., Perou C.M.
Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes.
Breast Cancer Res. Treat. 142:237-255(2013)
PubMed=24176112; DOI=10.1186/gb-2013-14-10-r110; PMCID=PMC3937590
Daemen A., Griffith O.L., Heiser L.M., Wang N.J., Enache O.M., Sanborn Z., Pepin F., Durinck S., Korkola J.E., Griffith M., Hur J.S., Huh N., Chung J., Cope L., Fackler M.J., Umbricht C.B., Sukumar S., Seth P., Sume V.P., Jakkula L.R., Lu Y.-L., Mills G.B., Cho R.J., Collisson E.A., van 't Veer L.J., Spellman P.T., Gray J.W.
Modeling precision treatment of breast cancer.
Genome Biol. 14:R110.1-R110.14(2013)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25892236; DOI=10.1016/j.celrep.2015.03.050; PMCID=PMC4425736
Lawrence R.T., Perez E.M., Hernandez D., Miller C.P., Haas K.M., Irie H.Y., Lee S.-I., Blau C.A., Villen J.
The proteomic landscape of triple-negative breast cancer.
Cell Rep. 11:630-644(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28287265; DOI=10.1021/acs.jproteome.6b00470; PMCID=PMC5557415
Yen T.-Y., Bowen S., Yen R., Piryatinska A., Macher B.A., Timpe L.C.
Glycoproteins in claudin-low breast cancer cell lines have a unique expression profile.
J. Proteome Res. 16:1391-1400(2017)
PubMed=28889351; DOI=10.1007/s10549-017-4496-x
Saunus J.M., Smart C.E., Kutasovic J.R., Johnston R.L., Kalita-de Croft P., Miranda M., Rozali E.N., Vargas A.C., Reid L.E., Lorsy E., Cocciardi S., Seidens T., McCart Reed A.E., Dalley A.J., Wockner L.F., Johnson J., Sarkar D., Askarian-Amiri M.E., Simpson P.T., Khanna K.K., Chenevix-Trench G., Al-Ejeh F., Lakhani S.R.
Multidimensional phenotyping of breast cancer cell lines to guide preclinical research.
Breast Cancer Res. Treat. 167:289-301(2018)
PubMed=29273624; DOI=10.1101/gr.226019.117; PMCID=PMC5793780
Franco H.L., Nagari A., Malladi V.S., Li W.-Q., Xi Y.-X., Richardson D., Allton K.L., Tanaka K., Li J., Murakami S., Keyomarsi K., Bedford M.T., Shi X.-B., Li W., Barton M.C., Dent S.Y.R., Kraus W.L.
Enhancer transcription reveals subtype-specific gene expression programs controlling breast cancer pathogenesis.
Genome Res. 28:159-170(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"