ATAC-seq简介
ATAC-seq(Assay for Transposase-Accessible Chromatin with highthroughput sequencing)是一种用于探究染色质开放区域的技术,该技术利用Tn5转座酶获得开放的染色质区段(open chromatin),通过高通量测序及生物信息学分析来挖掘相关基因及调控信息,以此探究生物学相关问题。ATAC-seq由Buenrostro于2013年首次提出[1],作为MNase-seq,FAIRE-seq和DNAse-seq的替代或补充方法。
ATAC-seq与MNase-seq、DNase-seq和FAIRE-seq比较而言,MNase-seq、DNase-seq即耗时费力与重复性差,虽然FAIRE-seq 不依赖酶,但其检测背景较高,测序信噪比低,甲醛交联时间不好把握等缺陷,大大限制其使用范围,ATAC-seq样本用量大幅减少,为研究珍稀及获取困难的样品提供可能。
ATAC-seq原理
ATAC seq技术就是利用了改造后的
Tn5转座酶,并将转座DNA设计为接头序列,这样便可以通过
“剪”和
“贴”的方式,直接将测序接头引入到开放的染色质中,进行捕获测序。
ATAC-seq服务流程
1、细胞悬液制备
2、细胞核纯化
3、Tn5酶切反应
4、文库构建
5、高通量测序
6、生信分析ATAC-seq应用
结合ATAC-seq优势以及研究热潮ATAC-seq有以下用途;当然进一步的开发也可能在不久的将来让其使用更加广泛。
1. 非生物逆境,病虫害,营养,激素等处理前后及动物疾病,转录活性差异。
2. 不同组织,器官转录活性差异,找到组织特异基因和启动子。
3. 利用ATAC-seq技术来研究A、B、D三个亚基因组的转录因子结合位点的差异,从而研究同源基因的表达调控差异。而六倍体与二倍体、四倍体等位基因间调控位点的比较。
4. 通过ATAC-seq定义的open chromatin区域 ,再结合motif 分析,识别哪种转录因子参与了基因表达调控(对于抗体质量不好的TF,尤其有效)
5. 而将ATAC-seq和RNA-seq进行整合研究,将会获得对生物体(动物或植物)中的转录调控机制;宏观分析细胞在该特定时空下整个基因组的调控网络。
ATAC-seq送样要求
| 样品类型 | 样品量 | 要求 | 样品处理 | 运输方式 |
| 细胞系 | >5×104个 | 细胞是完整的且为均匀的单细胞悬液; | 取样后直接速冻 | 干冰寄送(3d内) |
| 动物组织 | ≥1g | 组织新鲜无降解 |
| 植物组织 | ≥1g | 尽量选取新鲜幼嫩 |
| 真核微生物 | 需和老师协商 | 细胞核 |
ATAC-seq案例
研究人员选择胚胎发育的2-cell、4-cell、8-cell以及Blastocyst等时期利用ATAC-seq、RNA-seq发现了哺乳动物早期发育过程中染色体动态变化的特征以及可能的调控元件和转录因子,还揭示了在这个过程中染色质和转录调控元件不同于体细胞的特殊作用模式。
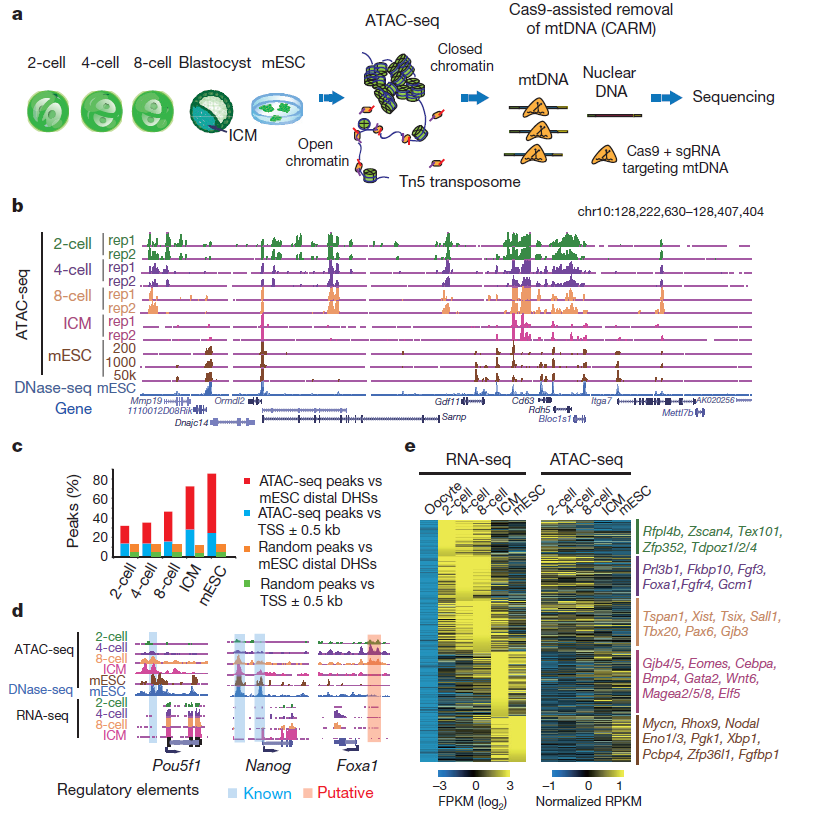
Wu, J., Huang, B., Chen, H., et al (2016). The landscape of accessible chromatin in mammalian preimplantation embryos. Nature, 534(7609), 652-657. doi: 10.1038/nature18606
资料下载:
武汉爱基百客ATAC-seq 产品手册.pdf
附件下载 (下载 5 次)