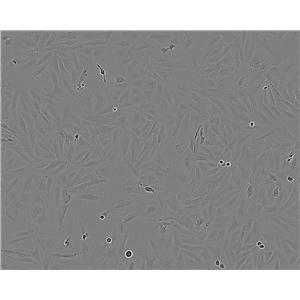
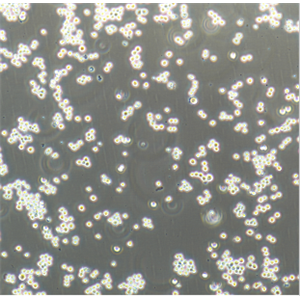
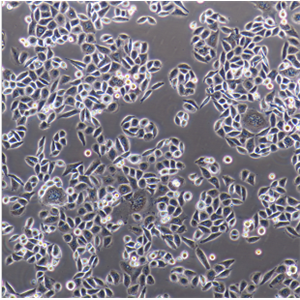
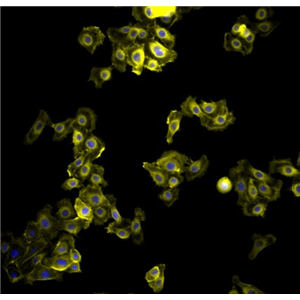
"Hep G2人肝癌细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
公司细胞系培养稳定、耐活、复苏好,提供人源、鼠源、兔源、猪源等不同组织如肺、气管、支气管、甲状腺、胰腺、垂体、肾上腺、扁桃体、胸腺、肾、膀胱、输尿管、前列腺、心脏、血管等细胞系。全程提供细胞系组织来源、生长特性、细胞形态、背景资料、培养条件、冻存条件、参考文献、运输方式等产品信息及技术服务。
换液周期:每周2-3次
Lewis-Lung Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH187细胞、CCRF细胞、HCC-1954细胞
MDAMB231 Cells;背景说明:MDA-MB-231来自患有转移乳腺腺癌的51岁女病人的胸水。在裸鼠和ALS处理的BALB/c小鼠中,它能形成低分化腺癌(III级)。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MUTZ-1细胞、PANC-08-13细胞、H128细胞
MHHCALL2 Cells;背景说明:急性B淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK-MEL-24细胞、Mouse Forestomach Carcinoma细胞、WM35细胞
背景信息:是一种人肝癌细胞系,是来自15岁男性白人的组织,该患者患有高度分化的肝细胞癌;Hep G2细胞形态为上皮细胞样,模式染色体数为55;Hep G2细胞在免疫抑制小鼠中不致瘤。
Hep G2人肝癌细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
Ocular Choroidal Melanoma-1 Cells;背景说明:葡萄膜黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:XuanWei Lung Cancer-05细胞、A375-SM细胞、CHP 212细胞
A3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SF-763细胞、X63-Ag8-653细胞、NPA87细胞
NS-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:MADB 106细胞、MLOY4细胞、KYSE-140细胞
2G7 [Mouse hybridoma against rat Hmgb1] Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
Hep G2人肝癌细胞全年复苏|已有STR图谱
形态特性:上皮细胞样
贴壁消化难题:1,先用PBS 把细胞洗两遍,使瓶内没有血清了,减少对胰酶的中和,然后用新配的0.25%的胰酶加入3ml左右,放在37度,然后可以在细胞有些消化下来时,拿着瓶口,运用手腕的力量轻轻震荡瓶内体,这样细胞很快就下来了,还不需要吹打,分散也均匀;2,成团、絮状:消化里加入eda可以减少细胞成团的现象,血清可以终止胰酶的作用,如果是进口血清的话也能终止eda的作用。用胰酶消化后胰酶可以倒掉,也可以不倒,直接加血清终止,如果消化中加入了eda的话,就要将消化倒干净,如果细胞贴壁要求不是很严格的话,一般不需要进行离心。鼻咽癌细胞的贴壁能力很强,用0.5%胰酶(含0.1%EDA)一般要消化12~15min。用PBS洗涤时要洗净残余的培养基,加入胰酶后在培养箱中消化(避免细胞室温下受损以及在此温度时胰酶活性Zui强)至细胞收缩变圆(可显微镜下观察)且有少许细胞脱落(有流下来的趋势),随后立即弃去胰酶(如果脱落的细胞很多且需要大量细胞实验,则不能弃去胰酶),加入培养基仔细吹打(不能用无血清培养基或者PBS替代,否则细胞聚集成团块或絮状)。一般我都离心一次弃去上清(去除残留的胰酶及漂浮的死细胞或细胞碎片);消化过度:马上用培养基中和,用吸管吹打细胞,收集全部的细胞到以无菌的离心管中800RPM 3分钟。弃上清,用全培重悬,换新的培养瓶继续培养,状态不HAO的细胞在培养的过程中会死亡脱落,在换的时候可以清除掉!
NRK Cells;背景说明:肾;自发永生;Osborne-Mendel;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DC2.4细胞、Sp2/O-Ag14细胞、NCTC-3960细胞
EoL-1 cell Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs746-T细胞、NGP细胞、143BTK-细胞
B16-BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C6661细胞、COLO 357细胞、HLEC-B3细胞
MALME 3M Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:A204细胞、HaCaT细胞、MDCK II细胞
MDA-MB-468-RED Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM346细胞、Medical Research Council cell strain-5细胞、Me Wo细胞
HUASMC Cells;背景说明:脐动脉平滑肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HC11细胞、SKLU01细胞、hTERT-HME1细胞
NSI/1-Ag4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:NCI-H2066细胞、Nthy-ori 3.1细胞、alpha TC1 clone 6细胞
ZR75_1 Cells;背景说明:该细胞产生高水平的黏液素MUC-1 mRNA,低水平的MUC-2 mRNA,但不表达MUC-3基因;表达雌激素受体。;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AML193细胞、CCC-ESF-1细胞、RPMI 2650细胞
SW 839 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:EL 4细胞、TT细胞、FU-97细胞
SUDHL-8 Cells;背景说明:弥漫大B淋巴瘤;腹腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK-MEL-1细胞、OCI-Ly18细胞、LS123细胞
CEM/0 Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCI-H1734细胞、GM01232E细胞、NTera-2D1细胞
SCC15 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H1092细胞、NCI-H1694细胞、Colo699细胞
BIU87 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H2591细胞、NCI-H358细胞、HSAEC1-KT细胞
U-251 MG Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:AML-EOL-1细胞、AMO1细胞、PC615.3细胞
HO-8910PM Cells;背景说明:高转移卵巢癌 Cells;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:3T6细胞、TYKnu细胞、T98G细胞
BL1339 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:L363细胞、OVISE细胞、H-358细胞
RenCa Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NIE 115细胞、SVOG细胞、MT-2细胞
35841 Cells(提供STR鉴定图谱)
Abcam HeLa UBE3B KO 1 Cells(提供STR鉴定图谱)
AIDHC-NMC2 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRJ220 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XK172 Cells(提供STR鉴定图谱)
C40210 Cells(提供STR鉴定图谱)
DA00280 Cells(提供STR鉴定图谱)
DA06234 Cells(提供STR鉴定图谱)
GM00800 Cells(提供STR鉴定图谱)
H-1355 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:CCC-HIE-2细胞、BERH-2细胞、UCLA NPA871细胞
IPEC-1 Cells;背景说明:小肠;上皮细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P3 (Jiyoye)细胞、CCRF/CEM/0细胞、SKNO1细胞
H82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:FRTL-5细胞、H596细胞、OCM-1细胞
NCI-H220 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEC-1A细胞、WM 239细胞、MUTZ1细胞
BTI-Tn5B14 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs 281.T细胞、CNE2细胞、LNCaP-ATCC细胞
Tca8113 Cells;背景说明:舌鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs 695T细胞、SUDHL-5细胞、Tu-212细胞
BV173 Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RGC-5细胞、RBL.2H3细胞、HS-445细胞
Hs 746.T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SK_N_BE2C细胞、U87 MG细胞、Pt K2 (NBL-5)细胞
Hep G2人肝癌细胞全年复苏|已有STR图谱
NCI-H1417 Cells;背景说明:小细胞肺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KTCTL-140细胞、SNU-620细胞、HuT78细胞
SNU638 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NIH:OVCAR3细胞、SP2-0-Ag14细胞、AD293细胞
HR8348 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:DHL10细胞、LS174T细胞、DMS153细胞
U-87 MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LAC细胞、L-6TG细胞、ST 486细胞
OVCAR432 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LU-65M细胞、Malme-3M细胞、H-2030细胞
KYSE 50 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HeLa S3细胞、HM1900细胞、Huh7.5.1细胞
RS4;11 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:KYSE 150 KYSE150 Kyse150 KY150
细胞、Colo741细胞、D10G41细胞
GM21952 Cells(提供STR鉴定图谱)
HAP1 MAVS (-) 2 Cells(提供STR鉴定图谱)
NCI-H446 Cells;背景说明:该细胞是1982年由CarneyD和GazdarAF等从一位小细胞肺癌患者的胸腔积液中建立的。细胞的原始形态并不具有小细胞肺癌特征。这个细胞株是小细胞肺癌的生化和形态学上的变种,表达神经元特有的烯醇酶和脑型肌酸激酶同工酶;左旋多巴脱羧酶、蚕素、抗利尿激素、催产素或胃泌激素释放肽未达到可检测水平。与正常细胞相比,该细胞c-mycDNA序列扩增约20倍,RNA增加15倍。最初传代培养基用含有5%FBS的RPMI1640,另外添加10nM化可的松、0.005mg/ml胰岛素、0.01mg/ml转铁;传代方法:1:2传代;生长特性:贴壁/悬浮生长,混合;形态特性:上皮样;相关产品有:WM 451-Lu细胞、TALL1细胞、H-226细胞
SaOS Cells;背景说明:该细胞是FoghJ和TrempeG分离和鉴定的众多人类肿瘤细胞系中的一种;该细胞来自一位11岁的白人女性的骨肉瘤组织。患者经过放疗以及甲喋呤、阿霉素、长春新碱、环磷酰胺和aramycin-C等多种药物治疗。该细胞在免疫抑制小鼠中不致瘤,细胞表达表皮生长因子EGF受体、转化生长因子β(1型和2型)受体。;传代方法:1:2-1:4传代;每周1-2次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:EA.Hy926细胞、RPMI #8226细胞、CWR22R-V1细胞
MLA144 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCA7细胞、LA-4 [Mouse lung adenoma]细胞、Scott and White No. 13细胞
B/C3T3 Cells;背景说明:胚胎;成纤维;自发永生;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1672细胞、HMC细胞、HNE3细胞
AN3 CA Cells;背景说明:AN3CA细胞建系于1964年。它衍生于子宫内膜癌患者淋巴结转移组织,具有癌细胞的基本特性,能在体外长期传代培养,接种实验动物产生明显肿瘤。但细胞的生物学特性及超微结构尚未深入研究,仅发现该细胞系促黑激素合成为阴性。细胞常用于人子宫内膜癌细胞生物学及其相关特性研究。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BV-2细胞、Ishikawa细胞、COLO 679细胞
HCC-9724 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LN299细胞、L-6 myoblast细胞、697细胞
SCC9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H2135细胞、CCD-841-CoTr细胞、NIH:OVCAR-10细胞
NCI-H220 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEC-1A细胞、WM 239细胞、MUTZ1细胞
HPSI0616i-mifg_2 Cells(提供STR鉴定图谱)
KB-C2.5 Cells(提供STR鉴定图谱)
MES19 Cells(提供STR鉴定图谱)
NI-1.9 Cells(提供STR鉴定图谱)
Raji-hPD-L1 Cells(提供STR鉴定图谱)
Ubigene A-549 FKBP5 KO Cells(提供STR鉴定图谱)
UP4-33 Cells(提供STR鉴定图谱)
HCT116-MFSD9-KO-c12 Cells(提供STR鉴定图谱)
3T6 Swiss Albino Cells;背景说明:胚胎;成纤维;自发永生;雄性;Swiss albino;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:D283-MED细胞、NCI H23细胞、TI-73细胞
PLA 802 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MCF-7ADR细胞、Fukuoka University-Malignant mixed Mullerian Tumor-1细胞、MGH-UI细胞
MPC-5 Cells;背景说明:肾足细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-SNU-886细胞、NCIH1703细胞、MOLM-14细胞
JROECL19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:LC1/Sq细胞、U118细胞、BT483细胞
Mc Ardle 7777 Cells;背景说明:肝癌;雌性;Buffalo;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PLC-PRF-5细胞、MBMEC细胞、SUDHL-6细胞
Mc Ardle 7777 Cells;背景说明:肝癌;雌性;Buffalo;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PLC-PRF-5细胞、MBMEC细胞、SUDHL-6细胞
NR8383 Cells;背景说明:NR8383(正常大鼠,1983年8月3日)来源于肺灌洗时的正常大鼠肺泡巨噬细胞。细胞在gerbil肺细胞连续培养液存在下培养了大约8-9个月。随后,不再需要外源生长因子。通过有限稀释法从单个细胞克隆并亚克隆NR8383细胞,并三次用软琼脂亚克隆。细胞表现出巨噬细胞的特性,吞噬酵母多糖和铜绿,非特异性脂酶活性,Fc受体,氧化降解;分泌IL-1,TNFbeta和IL-6,可重复地响应外源生长因子。NR8383细胞响应博莱霉素,分泌TGFbeta前体。在博莱霉素刺激下,TGFbe;传代方法:1:2传代;生长特性:半贴壁生长;形态特性:巨噬细胞;相关产品有:HAPI细胞、WM 451-Lu细胞、HEK293细胞
MHH-CALL2 Cells;背景说明:急性B淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:D-283MED细胞、W133细胞、BE(2)-C细胞
LK2 Cells;背景说明:肺鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H3396细胞、PaTu 8988 S细胞、PC-3M 2B4细胞
Fetal Rhesus Kidney-4 Cells;背景说明:胚胎;肾;自发永生;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IGR-OV1细胞、HOS TE85细胞、LAN-6细胞
Mc Coy Cells;背景说明:成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FHs74 Int细胞、526细胞、KASUMI1细胞
MB 157 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:UPCI:SCC090细胞、Z-138细胞、Hepatoma 22细胞
T-47D Cells;背景说明:浸润性导管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:149 PT细胞、HT115细胞、BCaP-37细胞
IMR32 Cells;背景说明:该细胞是1967年4月由NicholsWW,LeeJ和DwightS建立,来源于一名13月龄白人男婴腹部肿块,临床诊断为神经母细胞瘤,伴有极少部位的类器官样分化。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:存在两种细胞类型,小的神经母细胞样细胞和大的透明成纤维样细胞;相关产品有:Tu686细胞、TE8细胞、266 Bl细胞
RAW264 Cells;背景说明:单核巨噬细胞白血病;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TYK-nu细胞、CD18细胞、HIEC6细胞
ST91/MHT34 Cells(提供STR鉴定图谱)
TKB-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Human Embryo Lung-1细胞、AMC-HN-8细胞、KYSE270细胞
GM00346B Cells;背景说明:皮下结缔组织;自发永生;雄性;C3H/An;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ReNcell CX细胞、RCC10 RGB细胞、TR-146细胞
HDQ-P1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCT.116细胞、A-673细胞、HIEC细胞
SK-N-FI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Tb 1 Lu细胞、P3/X63/Ag8细胞、MCF 10A细胞
RPMI 1846 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AMC-HN8细胞、CMT64细胞、TE85细胞
HaCaT Cells;背景说明:该细胞源自一位62岁患有黑色素瘤男性的病灶外围正常皮肤,角蛋白、角化细胞交联外膜蛋白、中间丝相关蛋白阳性。;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HO-1-N-1细胞、Be Wo细胞、MGSMC细胞
L132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW 579细胞、UCLA SO M14细胞、SK-LMS-1细胞
H510A Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;每周换液2次。;生长特性:混合生长;形态特性:上皮细胞样;相关产品有:DHL-5细胞、PLB 985细胞、COLO-741细胞
Hep G2人肝癌细胞全年复苏|已有STR图谱
NCI-322 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H-2171细胞、H226细胞、NCI-H460细胞
Wien133 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CHL-CL-11细胞、Hs 683.T细胞、L-M(TK-)细胞
CFSC-2G Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H2405细胞、CAL12T细胞、TE-7细胞
NCIH1781 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Jijoye细胞、beta-TC6细胞、bEnd.3[BEND3]细胞
HIEC Cells;背景说明:肠;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HSC(Human Schwann)细胞、SKML-28细胞、HEK-A细胞
Hi5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEK293-A细胞、HEK 293细胞、RT-BM-1细胞
BayGenomics ES cell line RRR214 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHD181 Cells(提供STR鉴定图谱)
HT16 Cells(提供STR鉴定图谱)
PCRP-HES1-1A10 Cells(提供STR鉴定图谱)
ENU-T-1 Cells(提供STR鉴定图谱)
HPS2559 Cells(提供STR鉴定图谱)
" "Patent=US4393133
Knowles B.B., Aden D.P.
Human hepatoma derived cell line, process for preparation thereof, and uses therefor.
Patent number US4393133, 12-Jul-1983
PubMed=2439335; DOI=10.1111/j.1432-1033.1987.tb11497.x
Vincent C., Marceau M., Blangarin P., Bouic P., Madjar J.-J., Revillard J.-P.
Purification of alpha 1-microglobulin produced by human hepatoma cell lines. Biochemical characterization and comparison with alpha 1-microglobulin synthesized by human hepatocytes.
Eur. J. Biochem. 165:699-704(1987)
PubMed=8224613; DOI=10.1096/fasebj.7.14.8224613
Puisieux A., Galvin K., Troalen F., Bressac B., Marcais C., Galun E., Ponchel F., Yakicier C., Ji J.-W., Ozturk M.
Retinoblastoma and p53 tumor suppressor genes in human hepatoma cell lines.
FASEB J. 7:1407-1413(1993)
PubMed=8384076; DOI=10.1016/0165-4608(93)90227-D
Chen H.-L., Chiu T.-S., Chen P.-J., Chen D.-S.
Cytogenetic studies on human liver cancer cell lines.
Cancer Genet. Cytogenet. 65:161-166(1993)
PubMed=8389256; DOI=10.1093/carcin/14.5.987
Hsu I.-C., Tokiwa T., Bennett W.P., Metcalf R.A., Welsh J.A., Sun T.-T., Harris C.C.
p53 gene mutation and integrated hepatitis B viral DNA sequences in human liver cancer cell lines.
Carcinogenesis 14:987-992(1993)
PubMed=7972006; DOI=10.1073/pnas.91.23.11045; PMCID=PMC45163
Okamoto A., Demetrick D.J., Spillare E.A., Hagiwara K., Hussain S.P., Bennett W.P., Forrester K., Gerwin B.I., Serrano M., Beach D.H., Harris C.C.
Mutations and altered expression of p16INK4 in human cancer.
Proc. Natl. Acad. Sci. U.S.A. 91:11045-11049(1994)
PubMed=8050184; DOI=10.1111/j.1365-2249.1994.tb06089.x; PMCID=PMC1534706
Wadee A.A., Paterson A., Coplan K.A., Reddy S.G.
HLA expression in hepatocellular carcinoma cell lines.
Clin. Exp. Immunol. 97:328-333(1994)
PubMed=8835345; DOI=10.1002/(SICI)1096-9071(199602)48:2<133::aid-jmv3>3.0.CO;2-A
Tsuboi S., Nagamori S., Miyazaki M., Mihara K., Fukaya K.-i., Teruya K., Kosaka T., Tsuji T., Namba M.
Persistence of hepatitis C virus RNA in established human hepatocellular carcinoma cell lines.
J. Med. Virol. 48:133-140(1996)
DOI=10.11418/jtca1981.16.3_173
Mihara K., Miyazaki M., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ohashi R., Namba M.
The p53 gene status and other cellular characteristics of human cell lines maintained in our laboratory.
Tissue Cult. Res. Commun. 16:173-178(1997)
PubMed=9178645; DOI=10.1006/cimm.1997.1108
Nakao M., Sata M., Saitsu H., Yutani S., Kawamoto M., Kojiro M., Itoh K.
CD4+ hepatic cancer-specific cytotoxic T lymphocytes in patients with hepatocellular carcinoma.
Cell. Immunol. 177:176-181(1997)
PubMed=9359923; DOI=10.18926/AMO/30789
Mihara K., Miyazaki M., Kondo T., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ishioka C., Namba M.
Yeast functional assay of the p53 gene status in human cell lines maintained in our laboratory.
Acta Med. Okayama 51:261-265(1997)
PubMed=11050057; DOI=10.1053/jhep.2000.19349
Wong N., Lai P.B.-S., Pang E., Leung T.W.-T., Lau J.W.-Y., Johnson P.J.
A comprehensive karyotypic study on human hepatocellular carcinoma by spectral karyotyping.
Hepatology 32:1060-1068(2000)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=11981770; DOI=10.1053/jhep.2002.32668
Clemens D.L., Forman A., Jerrells T.R., Sorrell M.F., Tuma D.J.
Relationship between acetaldehyde levels and cell survival in ethanol-metabolizing hepatoma cells.
Hepatology 35:1196-1204(2002)
PubMed=12029633; DOI=10.1053/jhep.2002.33683
Yasui K., Arii S., Zhao C., Imoto I., Ueda M., Nagai H., Emi M., Inazawa J.
TFDP1, CUL4A, and CDC16 identified as targets for amplification at 13q34 in hepatocellular carcinomas.
Hepatology 35:1476-1484(2002)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
DOI=10.1385/CP:1:3-4:313
Pang R.T.-K., Poon T.C.-W., Wong N., Lai P.B.-S., Wong N.L.-Y., Chan C.M.-L., Yu J.W.S., Chan A.T.-C., Sung J.J.-Y.
Comparison of protein expression patterns between hepatocellular carcinoma cell lines and a hepatoblastoma cell line.
Clin. Proteomics 1:313-331(2004)
PubMed=14980111
Zhai B.-J., Wu F., Shao Z.-Y., Hu K., Zhao C.-L., Wang Z.-B.
Establishment of human hepatocellular carcinoma multidrug-resistance cell line (HepG2/Adm) and study apoptosis induced by low-frequency pulse ultrasound exposure.
Zhonghua Gan Zang Bing Za Zhi 12:95-98(2004)
PubMed=15767549; DOI=10.1158/1535-7163.MCT-04-0234
Nakatsu N., Yoshida Y., Yamazaki K., Nakamura T., Dan S., Fukui Y., Yamori T.
Chemosensitivity profile of cancer cell lines and identification of genes determining chemosensitivity by an integrated bioinformatical approach using cDNA arrays.
Mol. Cancer Ther. 4:399-412(2005)
PubMed=16181800; DOI=10.1016/j.biocel.2005.07.010
Donohue T.M., Osna N.A., Clemens D.L.
Recombinant Hep G2 cells that express alcohol dehydrogenase and cytochrome P450 2E1 as a model of ethanol-elicited cytotoxicity.
Int. J. Biochem. Cell Biol. 38:92-101(2006)
PubMed=16935386; DOI=10.1016/j.jhep.2006.05.019
Sun D.-X., Nassal M.
Stable HepG2- and Huh7-based human hepatoma cell lines for efficient regulated expression of infectious hepatitis B virus.
J. Hepatol. 45:636-645(2006)
PubMed=17254797; DOI=10.1016/j.biologicals.2006.10.001
Azari S., Ahmadi N., Jeddi-Tehrani M., Shokri F.
Profiling and authentication of human cell lines using short tandem repeat (STR) loci: report from the National Cell Bank of Iran.
Biologicals 35:195-202(2007)
PubMed=19215227; DOI=10.1111/j.1349-7006.2009.01082.x; PMCID=PMC11158180
Kuwahara Y., Li L., Baba T., Nakagawa H., Shimura T., Yamamoto Y., Ohkubo Y., Fukumoto M.
Clinically relevant radioresistant cells efficiently repair DNA double-strand breaks induced by X-rays.
Cancer Sci. 100:747-752(2009)
PubMed=19751877; DOI=10.1016/j.humpath.2009.07.003
Lopez-Terrada D.H., Cheung S.-W., Finegold M.J., Knowles B.B.
Hep G2 is a hepatoblastoma-derived cell line.
Hum. Pathol. 40:1512-1515(2009)
PubMed=20069059; DOI=10.1155/2010/437143; PMCID=PMC2801507
Srisomsap C., Sawangareetrakul P., Subhasitanont P., Chokchaichamnankit D., Chiablaem K., Bhudhisawasdi V., Wongkham S., Svasti J.
Proteomic studies of cholangiocarcinoma and hepatocellular carcinoma cell secretomes.
J. Biomed. Biotechnol. 2010:437143.1-437143.18(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20228232; DOI=10.1124/dmd.109.031831; PMCID=PMC2879958
Hart S.N., Li Y., Nakamoto K., Subileau E.-A., Steen D., Zhong X.-B.
A comparison of whole genome gene expression profiles of HepaRG cells and HepG2 cells to primary human hepatocytes and human liver tissues.
Drug Metab. Dispos. 38:988-994(2010)
PubMed=20937217; DOI=10.1170/149
Di Masi A., Viganotti M., Antoccia A., Magrelli A., Salvatore M., Azzalin G., Tosto F., Lorenzetti S., Maranghi F., Mantovani A., Macino G., Tanzarella C., Taruscio D.
Characterization of HuH6, Hep3B, HepG2 and HLE liver cancer cell lines by WNT/beta-catenin pathway, microRNA expression and protein expression profile.
Cell. Mol. Biol. 56:OL1299-OL1317(2010)
PubMed=21269460; DOI=10.1186/1752-0509-5-17; PMCID=PMC3039570
Burkard T.R., Planyavsky M., Kaupe I., Breitwieser F.P., Burckstummer T., Bennett K.L., Superti-Furga G., Colinge J.
Initial characterization of the human central proteome.
BMC Syst. Biol. 5:17.1-17.13(2011)
PubMed=22278370; DOI=10.1074/mcp.M111.014050; PMCID=PMC3316730
Geiger T., Wehner A., Schaab C., Cox J., Mann M.
Comparative proteomic analysis of eleven common cell lines reveals ubiquitous but varying expression of most proteins.
Mol. Cell. Proteomics 11:M111.014050-M111.014050(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23325432; DOI=10.1101/gr.147942.112; PMCID=PMC3589544
Varley K.E., Gertz J., Bowling K.M., Parker S.L., Reddy T.E., Pauli-Behn F., Cross M.K., Williams B.A., Stamatoyannopoulos J.A., Crawford G.E., Absher D.M., Wold B.J., Myers R.M.
Dynamic DNA methylation across diverse human cell lines and tissues.
Genome Res. 23:555-567(2013)
PubMed=23505090; DOI=10.1002/hep.26402
Wang K., Lim H.Y., Shi S., Lee J., Deng S.-B., Xie T., Zhu Z., Wang Y.-L., Pocalyko D., Yang W.J., Rejto P.A., Mao M., Park C.-K., Xu J.-C.
Genomic landscape of copy number aberrations enables the identification of oncogenic drivers in hepatocellular carcinoma.
Hepatology 58:706-717(2013)
PubMed=23887712; DOI=10.1038/ncomms3218; PMCID=PMC3731665
Nault J.-C., Mallet M., Pilati C., Calderaro J., Bioulac-Sage P., Laurent C., Laurent A., Cherqui D., Balabaud C., Zucman-Rossi J.
High frequency of telomerase reverse-transcriptase promoter somatic mutations in hepatocellular carcinoma and preneoplastic lesions.
Nat. Commun. 4:2218.1-2218.7(2013)
PubMed=24116068; DOI=10.1371/journal.pone.0075692; PMCID=PMC3792989
Weiskirchen R., Weimer J., Meurer S.K., Kron A., Seipel B., Vater I., Arnold N., Siebert R., Xu L.-M., Friedman S.L., Bergmann C.
Genetic characteristics of the human hepatic stellate cell line LX-2.
PLoS ONE 8:E75692-E75692(2013)
PubMed=24618588; DOI=10.1371/journal.pone.0091433; PMCID=PMC3950186
Chernobrovkin A.L., Zubarev R.A.
Detection of viral proteins in human cells lines by xeno-proteomics: elimination of the last valid excuse for not testing every cellular proteome dataset for viral proteins.
PLoS ONE 9:E91433-E91433(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25574106; DOI=10.3748/wjg.v21.i1.311; PMCID=PMC4284350
Cevik D., Yildiz G., Ozturk M.
Common telomerase reverse transcriptase promoter mutations in hepatocellular carcinomas from different geographical locations.
World J. Gastroenterol. 21:311-317(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26160117; DOI=10.1093/toxsci/kfv136; PMCID=PMC4583060
Sison-Young R.L.C., Mitsa D., Jenkins R.E., Mottram D., Alexandre E., Richert L., Aerts H., Weaver R.J., Jones R.P., Johann E., Hewitt P.G., Ingelman-Sundberg M., Goldring C.E.P., Kitteringham N.R., Park B.K.
Comparative proteomic characterization of 4 human liver-derived single cell culture models reveals significant variation in the capacity for drug disposition, bioactivation, and detoxication.
Toxicol. Sci. 147:412-424(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26825538; DOI=10.1016/j.jprot.2016.01.016
Wisniewski J.R., Vildhede A., Noren A., Artursson P.
In-depth quantitative analysis and comparison of the human hepatocyte and hepatoma cell line HepG2 proteomes.
J. Proteomics 136:234-247(2016)
PubMed=27027780; DOI=10.1007/s10565-016-9316-2
Wu Y., Geng X.-C., Wang J.-F., Miao Y.-F., Lu Y.-L., Li B.
The HepaRG cell line, a superior in vitro model to L-02, HepG2 and hiHeps cell lines for assessing drug-induced liver injury.
Cell Biol. Toxicol. 32:37-59(2016)
PubMed=27329724; DOI=10.18632/oncotarget.10161; PMCID=PMC5216950
Watari K., Nishitani A., Shibata T., Noda M., Kawahara A., Akiba J., Murakami Y., Yano H., Kuwano M., Ono M.
Phosphorylation of mTOR Ser2481 is a key target limiting the efficacy of rapalogs for treating hepatocellular carcinoma.
Oncotarget 7:47403-47417(2016)
PubMed=27470094; DOI=10.1016/j.chroma.2016.07.042
Liu Z.-Y., Wang F.-J., Chen J., Zhou Y., Zou H.-F.
Modulating the selectivity of affinity absorbents to multi-phosphopeptides by a competitive substitution strategy.
J. Chromatogr. A 1461:35-41(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29610054; DOI=10.1016/j.dmpk.2018.03.003; PMCID=PMC6309175
Shi J., Wang X.-W., Lyu L.-Y., Jiang H., Zhu H.-J.
Comparison of protein expression between human livers and the hepatic cell lines HepG2, Hep3B, and Huh7 using SWATH and MRM-HR proteomics: Focusing on drug-metabolizing enzymes.
Drug Metab. Pharmacokinet. 33:133-140(2018)
PubMed=29660373; DOI=10.1016/j.bbagen.2018.04.012
Touat-Hamici Z., Bulteau A.-L., Bianga J., Jean-Jacques H., Szpunar J., Lobinski R., Chavatte L.
Selenium-regulated hierarchy of human selenoproteome in cancerous and immortalized cells lines.
Biochim. Biophys. Acta 1862:2493-2505(2018)
PubMed=30629668; DOI=10.1371/journal.pone.0210404; PMCID=PMC6328144
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30864654; DOI=10.1093/nar/gkz169; PMCID=PMC6486628
Zhou B., Ho S.S., Greer S.U., Spies N., Bell J.M., Zhang X.-L., Zhu X.-W., Arthur J.G., Byeon S., Pattni R., Saha I., Huang Y.-L., Song G., Perrin D., Wong W.H., Ji H.P., Abyzov A., Urban A.E.
Haplotype-resolved and integrated genome analysis of the cancer cell line HepG2.
Nucleic Acids Res. 47:3846-3861(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31063779; DOI=10.1053/j.gastro.2019.05.001
Caruso S., Calatayud A.-L., Pilet J., La Bella T., Rekik S., Imbeaud S., Letouze E., Meunier L., Bayard Q., Rohr-Udilova N., Peneau C., Grasl-Kraupp B., de Koning L., Ouine B., Bioulac-Sage P., Couchy G., Calderaro J., Nault J.-C., Zucman-Rossi J., Rebouissou S.
Analysis of liver cancer cell lines identifies agents with likely efficacy against hepatocellular carcinoma and markers of response.
Gastroenterology 157:760-776(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31378681; DOI=10.1016/j.ccell.2019.07.001; PMCID=PMC7505724
Qiu Z.-X., Li H., Zhang Z.-T., Zhu Z.-F., He S., Wang X.-J., Wang P.-C., Qin J.-J., Zhuang L.-P., Wang W., Xie F.-B., Gu Y., Zou K.-K., Li C., Li C., Wang C.-H., Cen J., Chen X.-T., Shu Y.-J., Zhang Z., Sun L.-L., Min L.-H., Fu Y., Huang X.-W., Lv H., Zhou H., Ji Y., Zhang Z.-G., Meng Z.-Q., Shi X.-L., Zhang H.-B., Li Y.-X., Hui L.-J.
A pharmacogenomic landscape in human liver cancers.
Cancer Cell 36:179-193.e11(2019)
PubMed=31395879; DOI=10.1038/s41467-019-11415-2; PMCID=PMC6687785
Yu K., Chen B., Aran D., Charalel J., Yau C., Wolf D.M., van 't Veer L.J., Butte A.J., Goldstein T., Sirota M.
Comprehensive transcriptomic analysis of cell lines as models of primary tumors across 22 tumor types.
Nat. Commun. 10:3574.1-3574.11(2019)
PubMed=31561354; DOI=10.3233/CH-199226; PMCID=PMC6918903
Schulz C., Kammerer S., Kupper J.-H.
NADPH-cytochrome P450 reductase expression and enzymatic activity in primary-like human hepatocytes and HepG2 cells for in vitro biotransformation studies.
Clin. Hemorheol. Microcirc. 73:249-260(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)"