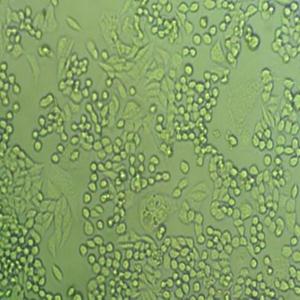
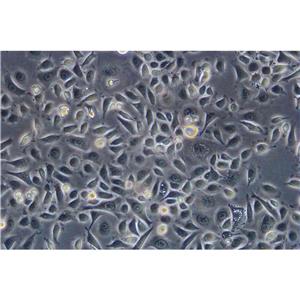
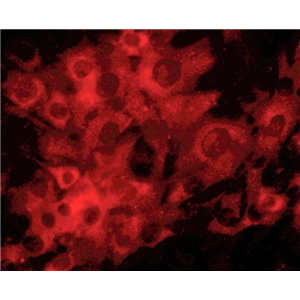
"DU 145人前列腺癌细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
换液周期:每周2-3次
RBMVEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:DoTc2 4510细胞、G401细胞、IPLB-SF-21-AE细胞
Ly7 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CBRH-7919细胞、H-748细胞、MD Anderson-Metastatic Breast-453细胞
PL45 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:4T1细胞、NHDF细胞、Centre Antoine Lacassagne-12T细胞
背景信息:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。
DU 145人前列腺癌细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
细胞培养应用于分子生物学、细胞生物学、遗传学、免疫学、肿瘤学及病毒学等领域,细胞培养是指将细胞从动物或植物体内取出,然后在适宜的人工环境中生长的过程。细胞可以在培养前直接从组织中取出并通过酶或机械方法进行解离,也可以来源于已建立的细胞系。传代培养是指当细胞生长至高密度时,将其分殖至新的培养瓶中,以维持其生长和增殖。贴壁细胞是指在培养基表面附着生长的细胞,悬浮细胞是指在培养基中悬浮生长的细胞,不依附于培养皿表面。半贴壁半悬浮细胞同时具备贴壁细胞和悬浮细胞的特点,通常在培养基中部分附着生长,部分悬浮于培养基上。
WPMY-1 Cells;背景说明:肌成纤维基质细胞株,WPMY-1,与RWPE-1cells(ATCCCRL-11609)一样,来源于同一张成人前列腺组织切片的周围区域的基质细胞。通过一个pRSTV质料结构,用SV40大T抗原对基质细胞进行永生化。WPMY-1细胞,与RWPE-1细胞及其它上皮细胞衍生株一样,属于来源于同一个前列腺的一系列细胞株。由于它们来源于同一个前列腺的周围区域,WPMY-1细胞株对于研究分泌和基质与上皮细胞相互作用尤其有用。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成肌细胞;相关产品有:8226细胞、K7M2-WT细胞、VM Cub 1细胞
DU_145 Cells;背景说明:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,酸性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:ETCC-007细胞、FT-293细胞、RCC 7860细胞
COLO.205 Cells;背景说明:该细胞系是1957年由T.U.Sample等从患有结肠癌的70岁男性白人的腹水中分离的。该病人在取腹水样品前已用5-尿嘧啶治疗4~6周。角蛋白免疫过氧化物酶染色阳性;产生CEA、IL10。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:PC3细胞、RCM1细胞、NCI-1155细胞
B16-F10.9.KG1 Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
DU 145人前列腺癌细胞全年复苏|已有STR图谱
形态特性:上皮细胞样
细胞传代培养实验:体外培养的原代细胞或细胞株要在体外持续地培养就必须传代,以便获得稳定的细胞株或得到大量的同种细胞,并维持细胞种的延续。培养的细胞形成单层汇合以后,由于密度过大生存空间不足而引起营养枯竭,将培养的细胞分散,从容器中取出,以1:2或1:3以上的比率转移到另外的容器中进行培养,即为传代培养;细胞“一代”指从细胞接种到分离再培养的一段期间,与细胞世代或倍增不同。在一代中,细胞培增3~6次。细胞传代后,一般经过三个阶段:游离期、指数增生期和停止期。常用细胞分裂指数表示细胞增殖的旺盛程度,即细胞群的分裂相数/100个细胞。一般细胞分裂指数介于0.2%~0.5%,肿瘤细胞可达3~5%;细胞接种2~3天分裂增殖旺盛,是活力ZuiHAO时期,称指数增生期(对数生长期),适宜进行各种试验。实验步骤:1.将长成的培养细胞从二氧化碳培养箱中取出,在超净工作台中倒掉瓶内的培养,加入少许消化。(以面盖住细胞为宜),静置5~10分钟。2.在倒置镜下观察被消化的细胞,如果细胞变圆,相互之间不再连接成片,这时应立即在超净台中将消化倒掉,加入3~5ml新鲜培养,吹打,制成细胞悬。3.将细胞悬吸出2ml左右,加到另一个培养瓶中并向每个瓶中分别加3ml左右培养,盖HAO瓶塞,送回二氧化碳培养箱中,继续进行培养。一般情况,传代后的细胞在2小时左右就能附着在培养瓶壁上,2~4天就可在瓶内形成单层,需要再次进行传代。
MRC-5 Cells;背景说明:MRC-5细胞系来自14周龄男性胎儿的正常肺组织,该细胞老化前能传代42~46个倍增时间。;传代方法:1:2-1:5传代;每周1-2次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Huh7.5细胞、SW 780细胞、ST2细胞
MES-23.5 Cells;背景说明:多巴胺能神经元 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P3J.HR1K细胞、TMD-8细胞、RT4细胞
EAC-E2G8 Cells;背景说明:腹水瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:OVCAR3细胞、TM-3细胞、A9 (Hamprecht)细胞
MDA-MB 361 Cells;背景说明:该细胞源自40岁女性乳腺癌的脑转移组织。;传代方法: 1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:Roswell Park Memorial Institute 6666细胞、HUASMC细胞、SF-126细胞
A549 ATCC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ECC1细胞、NCI-H2023细胞、NCIH2122细胞
WI-38 Cells;背景说明:LeonardHayflick建系;有限传代细胞系;寿命为50±10代(倍增时间24h);来自妊娠3个月的正常胚胎肺组织。该细胞系是第一个用于人制备的人二倍体细胞;培养基中添加TNFα可以加快细胞生长。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:RD细胞、LMTK-细胞、OCI-AML3细胞
SACCLM Cells;背景说明:涎腺腺样囊性癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK 293细胞、HCT/Taxo1细胞、Det. 562细胞
MODE-K Cells;背景说明:小肠;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RC-4B细胞、GM03190A细胞、H-69细胞
KTA7 Cells;背景说明:肺腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NuTu-19细胞、M07e细胞、QGY7701细胞
SK-N-BE(2)C Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA-MB157细胞、NCI-H64细胞、J-111细胞
A101D Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SNU-C2B细胞、NCI-A549细胞、B6Tert-1细胞
A498 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-196细胞、C2-C12细胞、OSC19细胞
HCC941122 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1963细胞、Blotchy fibroblast-11细胞、H-1793细胞
SUIT2 Cells;背景说明:胰腺管癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:rRMECs细胞、PG-4(S+L-)细胞、CHO Lec1细胞
CL1-0 Cells;背景说明:肺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-231细胞、NCIADR.RES细胞、PK(15)细胞
LMTK- Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ECV细胞、MUM2B细胞、MOLM-13细胞
NHRF Cells;背景说明:肾;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:D-341 Med细胞、MB 157细胞、IOSE 29细胞
Abcam HEK293T FKBP10 KO Cells(提供STR鉴定图谱)
AG10200 Cells(提供STR鉴定图谱)
BayGenomics ES cell line NPX499 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XC686 Cells(提供STR鉴定图谱)
BT121 Cells(提供STR鉴定图谱)
CPA-Yang3 Cells(提供STR鉴定图谱)
DA04092 Cells(提供STR鉴定图谱)
FAFU&CU-Px-2 Cells(提供STR鉴定图谱)
GM07816 Cells(提供STR鉴定图谱)
SU 86.86 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:UWB1.289细胞、HEK-293 c18细胞、Hs840_T细胞
H8 Cells;背景说明:宫颈;上皮细胞;永生化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU-354细胞、HCC-78细胞、Jurkat Clone E6-1细胞
NTERA-2 Cells;背景说明:畸胎瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TCMK1细胞、FD-LSC-1细胞、NCI-H378细胞
LNCaP FGC Cells;背景说明:人前列腺癌细胞LNCaP克隆FGC是从一位50岁白人男性(血型B+)的左锁骨淋巴结针刺活检中分离,该患者经确诊为前列腺癌转移。 这株细胞对5-α-二睾酮(生长调节子和酸性脂酶产物)有响应。这株细胞并不形成一致的单层,而是形成集落,在传代时可以用滴管反复吹吸打碎。它们仅仅轻轻地吸附在基底上,不形成汇合,很快使培养基变酸。生长很慢。传代后48小时内不应扰动。当培养瓶封包后,多数细胞从培养瓶底分离,悬浮在培养基中。收到后,在通常培养单层细胞的条件下培养24到48小时,以合细胞再贴壁。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:H446细胞、SMC-1细胞、H1092细胞
Scott and White No. 13 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H735细胞、MCF-7ADR细胞、NCIH740细胞
SKLU1 Cells;背景说明:该细胞系源于一位60岁的白人女性患者的肺腺癌组织。;传代方法:1:2传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BTI-Tn-5B1-4细胞、H2085细胞、CNE2细胞
TCCSUP Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:DiFi细胞、FOX-NY细胞、KYSE-70细胞
NCIH146 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:6传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:NCI-H64细胞、A375SM细胞、293-F细胞
DU 145人前列腺癌细胞全年复苏|已有STR图谱
Alpha Mouse Liver 12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:KTC-1细胞、COV-362细胞、EAhy 926细胞
NIH:OVCAR4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ly8细胞、CHL-1细胞、DK-MG细胞
LC-2 ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C17.2细胞、IMR-32细胞、SMA 560细胞
Stanford University-Diffuse Histiocytic Lymphoma-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:MDA-MB361细胞、SW-837细胞、Cloudman M3细胞
Human Lung Microvascular Endothelial Cell line-5a Cells;背景说明:肺微血管;内皮细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HS940细胞、S.B.细胞、Ca761细胞
GP293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KU 812细胞、CL-11细胞、BMSC/hBMSCs细胞
HCT GEO Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ISHI细胞、Rainbow Trout Embryo细胞、T_T_细胞
GM24173 Cells(提供STR鉴定图谱)
HAP1 NFATC1 (-) 2 Cells(提供STR鉴定图谱)
CEM clone 7 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NKM-1细胞、BTI-TN5B1-4细胞、SW-620细胞
NCI-H460 Cells;背景说明:该细胞1982年由A.F.Gazdar建系,源自一位患有大细胞肺癌的男性的胸腔积液。该细胞角蛋白、波形蛋白阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SK Mel28细胞、NCI-H1793细胞、SW 480细胞
MDA-MB361 Cells;背景说明:该细胞源自40岁女性乳腺癌的脑转移组织。;传代方法: 1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:DMS-53细胞、LA4细胞、Mouse Podocyte Clone-5细胞
EOC20 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COV-434细胞、AgC11x3A细胞、Madin-Darby Canine Kidney细胞
H1417 Cells;背景说明:小细胞肺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jiyoye细胞、GM03671C细胞、GM2131细胞
HSAS3 Cells;背景说明:皮肤;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PE/CA-PJ-34细胞、Buffalo Rat Liver-3A细胞、HUTU80细胞
ROS17/28 Cells;背景说明:骨肉瘤;ACI 9935;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Immortalized Human Hepatocytes细胞、MC57G细胞、IGROV-1细胞
EFO-27 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PLA-802细胞、NCIH1092细胞、PNT1A细胞
HQ02897 Cells(提供STR鉴定图谱)
KK-5 Cells(提供STR鉴定图谱)
MKT-BR Cells(提供STR鉴定图谱)
NRC-12 Cells(提供STR鉴定图谱)
RD/VCR-F3 Cells(提供STR鉴定图谱)
Ubigene HeLa CNTF KO Cells(提供STR鉴定图谱)
WTC-mEGFP-TJP1-cl20 Cells(提供STR鉴定图谱)
HG02222 Cells(提供STR鉴定图谱)
HT-144 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RMC-1细胞、SU-DH-L5细胞、Line 522细胞
COLO 678 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OVCA-432细胞、NCIH295R细胞、Panc-1-P细胞
4T1 Cells;背景说明:4T1是从410.4瘤株中未经诱变筛得的6-鸟嘌噙抗性细胞株。当注射到BALB/c小鼠中时,4T1自发产生高转移肿瘤,可转移到肺,肝,淋巴结和大脑,同时在注射部位形成始发灶。诱导转移时不需要摘除始发灶。4T1细胞在BALB/c小鼠中的生长与转移特性与人体中的乳腺癌十分相近。这种肿瘤是人VI期乳腺癌的动物模型。4T1-诱导的肿瘤在手术后及未手术情况下转移的动力学相近,可以用作手术后及未手术模型。跟其他肿瘤模型相比,由于4T1的抗6-鸟嘌噙特性,微小的转移细胞团(少到仅仅1个)也可以在许多远端器官中检测到。没必要数淋巴结或称重器官。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BSC40细胞、T173细胞、SK_HEP1细胞
SV40-HCEC Cells;背景说明:角膜上皮细胞;Ad-SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IOSE-Mar细胞、Capan 2细胞、Nthy ori 3.1细胞
MDAMB-231 Cells;背景说明:MDA-MB-231来自患有转移乳腺腺癌的51岁女病人的胸水。在裸鼠和ALS处理的BALB/c小鼠中,它能形成低分化腺癌(III级)。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KTCTL140细胞、SUM 149PT细胞、PC3-M细胞
MDAMB-231 Cells;背景说明:MDA-MB-231来自患有转移乳腺腺癌的51岁女病人的胸水。在裸鼠和ALS处理的BALB/c小鼠中,它能形成低分化腺癌(III级)。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KTCTL140细胞、SUM 149PT细胞、PC3-M细胞
HCC1599 Cells;背景说明:详见相关文献介绍;传代方法:每3-4天换液;生长特性:悬浮生长;形态特性:上皮样;相关产品有:ARO 81-1细胞、ChaGo-K-1细胞、HCC95细胞
H2195 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-SNU-449细胞、COLO679细胞、NCI H716细胞
Panc08.13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:PC-3M-1E8细胞、MEL-526细胞、NIH3T3-L1细胞
Hs 940.T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:CoC1细胞、TE10细胞、MEF (CF-1)细胞
MDAMB175VII Cells;背景说明:该细胞源自一位54岁患有乳腺导管癌白人女性的胸腔积液。;传代方法:1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:HCV-29细胞、CT-26细胞、WM451细胞
MDA-MB-134 VI Cells;背景说明:该细胞1973年由R. Cailleau建系,源自74岁乳腺导管癌女性患者的胸腔积液,细胞生长缓慢,松散贴壁,生长过程中会脱落到培养基,不会汇合,过表达FGF受体;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:G292细胞、Centre Antoine Lacassagne-33细胞、Plaepi 34细胞
WC00044 Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK 293 EBNA细胞、Stanford University-Diffuse Histiocytic Lymphoma-16细胞、MDA-175细胞
NCI-460 Cells;背景说明:该细胞1982年由A.F.Gazdar建系,源自一位患有大细胞肺癌的男性的胸腔积液。该细胞角蛋白、波形蛋白阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CAL12T细胞、GFP-Olig2细胞、MKN-7细胞
HCC1588 Cells;背景说明:肺鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-211细胞、MDA-kb2细胞、H-3255细胞
STBCi048-A Cells(提供STR鉴定图谱)
OC-316 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ZR75_1细胞、HT-115细胞、alpha-TC1.6细胞
NCI-SNU-761 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:293细胞、KM12细胞、SNU-182细胞
NB-9 Cells;背景说明:详见相关文献介绍;传代方法:1:10 1:50每2 - 3周;每周换液2-3次。;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:LYR细胞、HEM细胞、HEK-AD 293细胞
Mouse Podocyte Clone-5 Cells;背景说明:肾足细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DSL-6A/C1细胞、HCEC-12细胞、U-251细胞
VM Cub 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:INS1E细胞、HLEpiC细胞、22Rv1细胞
COV 434 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:HEK/EBNA细胞、SKNBE2C细胞、HT1197细胞
SNU739 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CF-PAC1细胞、149 PT细胞、SNU-484细胞
U2-OS Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:bEnd3细胞、SK-RC-52细胞、OAW-42细胞
DU 145人前列腺癌细胞全年复苏|已有STR图谱
NMC-G1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MB157细胞、HLCL9B10细胞、PanC1细胞
SW-1573 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDBK (NBL-1)细胞、X63.Ag8.653细胞、SNU182细胞
Hos TE-85 Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MS1细胞、293-FT细胞、OC-316细胞
H-35 Reuber Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:COLO 394细胞、BJAB细胞、TE-15细胞
RIN m5F Cells;背景说明:胰岛β细胞瘤;雄性;NEDH;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KHYG细胞、R 1610细胞、U138-MG细胞
Panc 4.03 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MuM-2C细胞、CFPAC细胞、Hs729T细胞
BayGenomics ES cell line RRI148 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XH649 Cells(提供STR鉴定图谱)
F1-3G8-1 Cells(提供STR鉴定图谱)
NCI-HPN-F24 Mam Cells(提供STR鉴定图谱)
utr-1 Cells(提供STR鉴定图谱)
MCB3901 Cells(提供STR鉴定图谱)
" "PubMed=7017212; DOI=10.1093/jnci/66.6.1003
Pollack M.S., Heagney S.D., Livingston P.O., Fogh J.
HLA-A, B, C and DR alloantigen expression on forty-six cultured human tumor cell lines.
J. Natl. Cancer Inst. 66:1003-1012(1981)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2744886; DOI=10.1002/ijc.2910440128
Hartley-Asp B., Billstrom A., Kruse E.
Identification by C-banding of two human prostate tumour cell lines, 1013L and DU 145.
Int. J. Cancer 44:161-164(1989)
PubMed=1873816
Isaacs W.B., Carter B.S., Ewing C.M.
Wild-type p53 suppresses growth of human prostate cancer cells containing mutant p53 alleles.
Cancer Res. 51:4716-4720(1991)
PubMed=1574572; DOI=10.2307/3578273
Dunphy E.J., Beckett M.A., Thompson L.H., Weichselbaum R.R.
Expression of the polymorphic human DNA repair gene XRCC1 does not correlate with radiosensitivity in the cells of human head and neck tumor cell lines.
Radiat. Res. 130:166-170(1992)
PubMed=8104329; DOI=10.1002/pros.2990230206
Carroll A.G., Voeller H.J., Sugars L., Gelmann E.P.
p53 oncogene mutations in three human prostate cancer cell lines.
Prostate 23:123-134(1993)
PubMed=8510267; DOI=10.1016/S0022-5347(17)35458-7
Effert P.J., McCoy R.H., Walther P.J., Liu E.T.-B.
p53 gene alterations in human prostate carcinoma.
J. Urol. 150:257-261(1993)
PubMed=9018337; DOI=10.1002/(SICI)1097-0045(19970101)30:1<58::aid-pros9>3.0.CO;2-H
Webber M.M., Bello D., Quader S.T.A.
Immortalized and tumorigenic adult human prostatic epithelial cell lines: characteristics and applications Part 2. Tumorigenic cell lines.
Prostate 30:58-64(1997)
PubMed=9214606; DOI=10.1093/carcin/18.6.1225
Webber M.M., Bello D., Kleinman H.K., Hoffman M.P.
Acinar differentiation by non-malignant immortalized human prostatic epithelial cells and its loss by malignant cells.
Carcinogenesis 18:1225-1231(1997)
PubMed=9460501; DOI=10.1016/S0165-4608(97)00060-5
Nupponen N.N., Hyytinen E.-R., Kallioniemi A.H., Visakorpi T.
Genetic alterations in prostate cancer cell lines detected by comparative genomic hybridization.
Cancer Genet. Cytogenet. 101:53-57(1998)
PubMed=10702678; DOI=10.1159/000015432
Pan Y., Kytola S., Farnebo F., Wang N., Lui W.-O., Nupponen N.N., Isola J.J., Visakorpi T., Bergerheim U.S.R., Larsson C.
Characterization of chromosomal abnormalities in prostate cancer cell lines by spectral karyotyping.
Cytogenet. Cell Genet. 87:225-232(1999)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=10972993; DOI=10.1002/1098-2744(200008)28:4<236::aid-mc6>3.0.CO;2-H
Rauh-Adelmann C., Lau K.-M., Sabeti N., Long J.P., Mok S.C., Ho S.-M.
Altered expression of BRCA1, BRCA2, and a newly identified BRCA2 exon 12 deletion variant in malignant human ovarian, prostate, and breast cancer cell lines.
Mol. Carcinog. 28:236-246(2000)
PubMed=11135431; DOI=10.1002/1098-2264(2000)9999:9999<::aid-gcc1076>3.0.CO;2-E
Aurich-Costa J., Vannier A., Gregoire E., Nowak F., Cherif D.
IPM-FISH, a new M-FISH approach using IRS-PCR painting probes: application to the analysis of seven human prostate cell lines.
Genes Chromosomes Cancer 30:143-160(2001)
PubMed=11172901; DOI=10.1016/S0165-4608(00)00339-3
Strefford J.C., Lillington D.M., Young B.D., Oliver R.T.D.
The use of multicolor fluorescence technologies in the characterization of prostate carcinoma cell lines: a comparison of multiplex fluorescence in situ hybridization and spectral karyotyping data.
Cancer Genet. Cytogenet. 124:112-121(2001)
PubMed=11280753
Urasaki Y., Laco G.S., Pourquier P., Takebayashi Y., Kohlhagen G., Gioffre C., Zhang H.-L., Chatterjee D., Pantazis P., Pommier Y.
Characterization of a novel topoisomerase I mutation from a camptothecin-resistant human prostate cancer cell line.
Cancer Res. 61:1964-1969(2001)
PubMed=11304728; DOI=10.1002/pros.1045
van Bokhoven A., Varella-Garcia M., Korch C.T., Hessels D., Miller G.J.
Widely used prostate carcinoma cell lines share common origins.
Prostate 47:36-51(2001)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=12606952; DOI=10.1038/sj.onc.1206247
Clark J., Edwards S., Feber A., Flohr P., John M., Giddings I., Crossland S., Stratton M.R., Wooster R., Campbell C., Cooper C.S.
Genome-wide screening for complete genetic loss in prostate cancer by comparative hybridization onto cDNA microarrays.
Oncogene 22:1247-1252(2003)
PubMed=12615715
Reinhold W.C., Kouros-Mehr H., Kohn K.W., Maunakea A.K., Lababidi S., Roschke A.V., Stover K., Alexander J., Pantazis P., Miller L., Liu E.T.-B., Kirsch I.R., Urasaki Y., Pommier Y., Weinstein J.N.
Apoptotic susceptibility of cancer cells selected for camptothecin resistance: gene expression profiling, functional analysis, and molecular interaction mapping.
Cancer Res. 63:1000-1011(2003)
PubMed=12725112; DOI=10.1385/1-59259-372-0:21
Russell P.J., Kingsley E.A.
Human prostate cancer cell lines.
Methods Mol. Med. 81:21-39(2003)
PubMed=14518029; DOI=10.1002/pros.10290
van Bokhoven A., Varella-Garcia M., Korch C.T., Johannes W.U., Smith E.E., Miller H.L., Nordeen S.K., Miller G.J., Lucia M.S.
Molecular characterization of human prostate carcinoma cell lines.
Prostate 57:205-225(2003)
CLPUB00698
van Bokhoven A.
Models for prostate cancer. Molecular characterization and critical appraisal of human prostate carcinoma cell lines.
Thesis PhD (2004); Katholieke Universiteit Nijmegen; Nijmegen; Netherlands
PubMed=15486987; DOI=10.1002/pros.20158
Zhao H.-J., Kim Y., Wang P., Lapointe J., Tibshirani R., Pollack J.R., Brooks J.D.
Genome-wide characterization of gene expression variations and DNA copy number changes in prostate cancer cell lines.
Prostate 63:187-197(2005)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=17254797; DOI=10.1016/j.biologicals.2006.10.001
Azari S., Ahmadi N., Jeddi-Tehrani M., Shokri F.
Profiling and authentication of human cell lines using short tandem repeat (STR) loci: report from the National Cell Bank of Iran.
Biologicals 35:195-202(2007)
PubMed=17440963; DOI=10.1002/pros.20581
Takeda M., Mizokami A., Mamiya K., Li Y.-Q., Zhang J., Keller E.T., Namiki M.
The establishment of two paclitaxel-resistant prostate cancer cell lines and the mechanisms of paclitaxel resistance with two cell lines.
Prostate 67:955-967(2007)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22275356; DOI=10.1074/jbc.M111.302547; PMCID=PMC3322861
Lynch T.P., Ferrer C.M., Jackson S.R., Shahriari K.S., Vosseller K., Reginato M.J.
Critical role of O-linked beta-N-acetylglucosamine transferase in prostate cancer invasion, angiogenesis, and metastasis.
J. Biol. Chem. 287:11070-11081(2012)
PubMed=22336246; DOI=10.1016/j.bmc.2012.01.017
Kong D.-X., Yamori T.
JFCR39, a panel of 39 human cancer cell lines, and its application in the discovery and development of anticancer drugs.
Bioorg. Med. Chem. 20:1947-1951(2012)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=23671654; DOI=10.1371/journal.pone.0063056; PMCID=PMC3646030
Lu Y.-H., Soong T.D., Elemento O.
A novel approach for characterizing microsatellite instability in cancer cells.
PLoS ONE 8:E63056-E63056(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=23957452; DOI=10.3109/01913123.2013.814739
Gilloteaux J., Eze N., Jamison J.M., McGuire K., Summers J.L.
A rare, human prostate oncocyte cell originates from the prostatic carcinoma (DU145) cell line.
Ultrastruct. Pathol. 37:440-448(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26972028; DOI=10.1016/j.jprot.2016.03.008
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Identification of glycosylphosphatidylinositol-anchored proteins and omega-sites using TiO2-based affinity purification followed by hydrogen fluoride treatment.
J. Proteomics 139:77-83(2016)
PubMed=27141528; DOI=10.1016/j.dib.2016.04.001; PMCID=PMC4838930
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Data for identification of GPI-anchored peptides and omega-sites in cancer cell lines.
Data Brief 7:1302-1305(2016)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=30244336; DOI=10.1007/s00345-018-2501-6
Samli H., Samli M., Vatansever B., Ardicli S., Aztopal N., Dincel D., Sahin A., Balci F.
Paclitaxel resistance and the role of miRNAs in prostate cancer cell lines.
World J. Urol. 37:1117-1126(2019)
PubMed=30629668; DOI=10.1371/journal.pone.0210404; PMCID=PMC6328144
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30787054; DOI=10.1158/1055-9965.EPI-18-1132; PMCID=PMC6548687
Hooker S.E. Jr., Woods-Burnham L., Bathina M., Lloyd S., Gorjala P., Mitra R., Nonn L., Kimbro K.S., Kittles R.A.
Genetic ancestry analysis reveals misclassification of commonly used cancer cell lines.
Cancer Epidemiol. Biomarkers Prev. 28:1003-1009(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31395879; DOI=10.1038/s41467-019-11415-2; PMCID=PMC6687785
Yu K., Chen B., Aran D., Charalel J., Yau C., Wolf D.M., van 't Veer L.J., Butte A.J., Goldstein T., Sirota M.
Comprehensive transcriptomic analysis of cell lines as models of primary tumors across 22 tumor types.
Nat. Commun. 10:3574.1-3574.11(2019)"