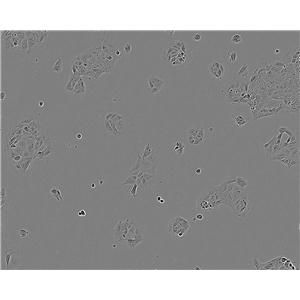
"Caki-1人肾透明细胞癌皮肤转移细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
公司细胞系形态漂亮、增殖倍数高、纯度高、功能性强,细胞培养就跟养孩子一个样。养孩子要喂奶,养细胞要加补液,都需要在前期补充足够的营养,初始状态的细胞或刚刚复苏的细胞还要适量加入血清或细胞因子来帮助它们的存活增殖,如果营养物质缺乏,细胞就会不生长甚至死亡。养孩子要从小培养学习,养细胞也得培养宝宝顺利生下来,你会经常抚摸他,给他看各种颜色,刺激他的五感。细胞也是一样,分离后的细胞需要使用特定的细胞因子进行活化、增殖。另外加入因子的种类、因子的浓度、加入时间、加入顺序都会影响细胞最终的结果。养孩子最怕孩子生病,养细胞最怕被污染,平时你会仔细观察宝宝是否呕吐、是否突然哭闹,猜测宝宝是否生病了。对于细胞,我们也需要时刻进行观察的,假如培养液浑浊(污染了),则需要换液后加抗生素;假如细胞增殖不明显,形态变差,则可能是因为营养不足了,对贴壁细胞可以消化后重新用新的培养基接种并加倍加入细胞因子含量;对悬浮细胞增殖能力不强的,则不着急补液,只是先补加血清、细胞因子看是否可以好转。培养时还得全程在无菌的环境,一个小小的偏差,细胞就会死亡。
换液周期:每周2-3次
NCIH2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:74Int细胞、HPAFII细胞、Homo sapiens No. 578, breast cells细胞
COC1/CDDP Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:UCLA RO-81A-1细胞、IFRS1细胞、DHL-6细胞
NCI-460 Cells;背景说明:该细胞1982年由A.F.Gazdar建系,源自一位患有大细胞肺癌的男性的胸腔积液。该细胞角蛋白、波形蛋白阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CAL12T细胞、GFP-Olig2细胞、MKN-7细胞
背景信息:该细胞超微结构中包含许多微绒毛、少许微丝、许多小线粒体、发达的高尔基休和内质网、许多脂滴和多层体、次级溶酶体,没有发现病毒颗粒。
Caki-1人肾透明细胞癌皮肤转移细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
ATCC细胞库(American Type Culture Colection),该中心一直致力于细胞分类、鉴定和保藏工作。ATCC是全球最大的生物资源保藏中心,ATCC通过行业标准产品、服务和创新解决方案支持全球学术、政府、生物技术、制药、食品、农业和工业领域的科学进步。ATCC提供的服务和定制解决方案包括细胞和微生物培养、鉴定、生物衍生物的开发和生产、性能测试和生物资源保藏服务。美国国家标准协会(ANSI)认可了ATCC标准开发组织,并制定了标准协议,以确保生物材料的可靠性和可重复性。ATCC的使命是为了获取、鉴定、保存、开发、标准化和分发生物资源和生物信息,以提高和应用生物科学知识。
WEHI-3B Cells;背景说明:白血病;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Okayama University Medical School-23细胞、H1385细胞、GA-10-Clone-4细胞
MDA330 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Pt K2细胞、PG [Human lung carcinoma]细胞、ROS-17/2.8细胞
HUT28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:COLO 320 HSR细胞、KS-1 [Human glioblastoma]细胞、QSG7701细胞
802C11 Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
Caki-1人肾透明细胞癌皮肤转移细胞全年复苏|已有STR图谱
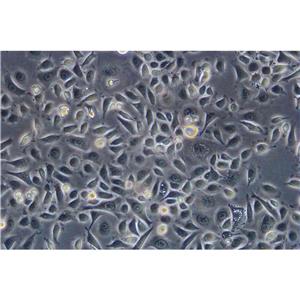
形态特性:上皮细胞样
细胞冻存复苏材料与方法步骤:常用的细胞冷冻贮存器为贮存器,规格有35L和50L两种。使用时要注意以下几点:(1)一般两周需充一次,至少一个月充一次。温度达-196℃,使用时注意勿让溅到皮肤上,以免引起冻伤。(2)容器为双层结构,中间为真空层,瓶口有双层焊接处,应防止焊接部裂开。(3)在装入时,要注意缓慢小心,并用厚纸卷筒或制漏斗作引导,使直达瓶底,如有专用灌注装置则更HAO。若为初次使用,加时更要缓慢,以免温度骤降而使容器损坏。细胞冻存时常备的材料有:0.25%胰蛋白酶,含10%~20%的血清培养,DMSO(分析纯)或无色新鲜甘油(121°C蒸气GAO压消毒),2mL安瓿(或专用细胞冻存管)、吸管、离心管、喷灯、纱布袋(或冻存管架)等。主要操作步骤为:(1)选择处于对数生长期的细胞,在冻存前一天ZuiHAO换。将多个培养瓶中的细胞培养 去掉,用0.25%胰蛋白酶消化。适时去掉胰蛋白酶,加入少量新培养。用吸管吸取培养反复吹打瓶壁上的细胞,使其成为均匀分散的细胞悬。悬浮生产细胞则不要消化处理。然后将细胞收集于离心管中离心(1000r/min,10分钟)。(2)去上清,加入含20%小牛血清的完全培养基,于4℃预冷15分钟后,逐滴加入已无菌的DMSO或甘油,用吸管轻轻吹打使细胞均匀,细胞浓度为3×106~1×107/mL之间。(3)将上述细胞分装于安瓿或专用冷冻塑料管中,安瓿装1~1.5mL在火焰喷灯上封口,封口处要完全封闭,圆滑无勾。冷冻管要将盖子盖紧,并标记HAO细胞名称和冻存日期,同时作HAO登记(日期、细胞种类及代次、冻存支数)。(4)将装HAO细胞的安瓿或冻存管装入沙布袋内;置于容器颈口处存放过夜,次日转入中。采用控制降温速度的方法也可采用下列步骤:先将安瓿置入4℃冰箱中2~3小时,再移至冰箱冷冻室内3~4小时,再吊入容器颈气态部分存放2小时,Zui后沉入中。细胞冻存在中可以长期保存,但为妥善起见,冻存半年后,ZuiHAO取出一只安瓿细胞复苏培养,观察生长情况,然后再继续冻存。
MEF (CF-1) Cells;背景说明:胚胎;成纤维细胞;CF-1;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HPDE6c7细胞、RMG-I细胞、1301细胞
NCI-H2195 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Menschliche Und Tierische Zellkulture-3细胞、HEK 293 EBNA细胞、VMCUB1细胞
UCLA-SO-M21 Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NK92MI细胞、STO细胞、MV-3细胞
HTR8 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:JHH2细胞、Farage OL细胞、HDQP1细胞
AG06814-M Cells;背景说明:LeonardHayflick建系;有限传代细胞系;寿命为50±10代(倍增时间24h);来自妊娠3个月的正常胚胎肺组织。该细胞系是第一个用于人制备的人二倍体细胞;培养基中添加TNFα可以加快细胞生长。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:J774 A.1细胞、SNU-761细胞、Walker/LLC-WRC256细胞
ZR75_1 Cells;背景说明:该细胞产生高水平的黏液素MUC-1 mRNA,低水平的MUC-2 mRNA,但不表达MUC-3基因;表达雌激素受体。;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AML193细胞、CCC-ESF-1细胞、RPMI 2650细胞
PK(15) Cells;背景说明:PK-15细胞建系于1955(Stice,E)。是PK-1a细胞的克隆系。该细胞系可用于多种病毒的增值及特性研究。另外,电镜观察发现,PK-15细胞内有C-型病毒颗粒存在,是研究C-型病毒的材料。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:SW403细胞、T/G HA-VSMC细胞、BGC823细胞
SK N SH Cells;背景说明:SK-N-SH细胞系由J.L.Bieder建系,它与SK-N-MC所不同的是倍增时间较长且多巴胺-β-羟基酶水平较高。 SK-N-SH在细胞介导的细胞毒性试验中用作靶细胞系。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:上皮细胞样;相关产品有:MDCC MSB1细胞、Hopkins-92细胞、H-1341细胞
OCI-AML2 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:COLO320DM细胞、Panc02.03细胞、HNE1细胞
SKM-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HGMC细胞、GM00637H细胞、L cells细胞
U-118-MG Cells;背景说明:注意: 据报道来自不同个体的胶质母细胞瘤细胞株U-118 MG (HTB-15) 和 U-138 MG (HTB-16)有着一致的VNTR和相近的STR模式。 U-118 MG 和 U-138 MG细胞遗传学上很相似并有至少六个衍生标记染色体。 这是1966年至1969年间J. Ponten和同事从恶性神经胶质瘤中构建的细胞株中的一株(其它包括ATCC HTB-14和 ATCC HTB-16 and ATCC HTB-17)。 1987年用BM-Cycline培养6周去除了支原体污染。 ;传代方法: 消化3-5分钟。1:2传代。3天内可长满。;生长特性:贴壁生长;形态特性:混合型;相关产品有:KP4细胞、Hs611T细胞、NB-9细胞
SW900 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H510细胞、HCA7细胞、BxPC-3细胞
CCD-1095Sk Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SCC 9细胞、hFOB 1.19细胞、HCO细胞
JIMT1 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKMel-28细胞、CL40细胞、Calu-6细胞
Panc-8_13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HUV-EC-C细胞、KMST6细胞、NCIH838细胞
WI 38 Cells;背景说明:LeonardHayflick建系;有限传代细胞系;寿命为50±10代(倍增时间24h);来自妊娠3个月的正常胚胎肺组织。该细胞系是第一个用于人制备的人二倍体细胞;培养基中添加TNFα可以加快细胞生长。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:GM-346细胞、MOLT-4细胞、Okayama University Medical School-27细胞
B16BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEI193细胞、Rat Basophilic Leukemia-1细胞、MSF细胞
346F1 Cells(提供STR鉴定图谱)
Abcam HeLa TRIM7 KO Cells(提供STR鉴定图谱)
AIBNi011-A Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRJ156 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XK142 Cells(提供STR鉴定图谱)
C3H/HeN-emb Cells(提供STR鉴定图谱)
DA00260 Cells(提供STR鉴定图谱)
E3GS2.4 Cells(提供STR鉴定图谱)
GM04693 Cells(提供STR鉴定图谱)
HBE 135-E6/E7 Cells;背景说明:支气管上皮细胞;HPV16转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FHL-124细胞、SCC 9细胞、HPMC细胞
T-98 Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKml2细胞、MKN-28细胞、MDCK Type II细胞
ACC-M Cells;背景说明:涎腺腺样囊性癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MCF10-A细胞、NFS60细胞、HN13细胞
KP-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:ES-2细胞、IR983F细胞、P-19细胞
JG Cells;背景说明:肾小球旁 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SCC9细胞、High5细胞、MFE-296细胞
786-O RCC Cells;背景说明:该细胞源自一位原发性肾透明细胞癌患者。该细胞有微绒毛和桥粒,能在软琼脂上生长。此细胞生成一种PTH(甲状旁腺素)样的多肽,与乳癌和肺癌中生成的肽相似,其N端序列与PTH相似,具有PTH样活性,分子量为6000D。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SMA 560细胞、Murine Carcinoma-38细胞、VK2/E6E7细胞
UMRC2 Cells;背景说明:肾透明细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:32D/cl3细胞、Malme3M细胞、HBZY-1细胞
NCIH889 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮细胞;相关产品有:LS180细胞、SK-HEP-1细胞、FHL 124细胞
Caki-1人肾透明细胞癌皮肤转移细胞全年复苏|已有STR图谱
M1 Cells;背景说明:髓系白血病;SL;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:VM-CUB-1细胞、COLO-320细胞、MDA-MB-435S细胞
KPNRTBM1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:32D clone3细胞、H 9细胞、CP-70细胞
NCI-H2141 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:KTA-7细胞、Ca9-22细胞、BIC-1细胞
H1563 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:GM00346细胞、T98细胞、MT2细胞
EFM-192B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH548细胞、CEK细胞、Hs 578T细胞
EFM-192B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH548细胞、CEK细胞、Hs 578T细胞
OCI-Ly 10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:IEC 18细胞、HLE B-3细胞、WILL-2细胞
GM20272 Cells(提供STR鉴定图谱)
HAP1 ITGA3 (-) 1 Cells(提供STR鉴定图谱)
NCIH2085 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Hs840T细胞、Mino细胞、IEC6细胞
DLM8 Cells;背景说明:Dunn's骨肉瘤;雌性;C3H;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TFH细胞、U-343细胞、CC-LP-1细胞
SW-579 Cells;背景说明:在裸鼠中成瘤(产生三级恶性纺锤状巨细胞瘤)。 ;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:OCI/AML3细胞、M-G63细胞、MAVER细胞
HSC3 Cells;背景说明:详见相关文献介绍;传代方法:1 x 10^5 cells/10cm dish;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Panc3_27细胞、NCI-H498细胞、HuTu-80细胞
HT115 Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OCI-AML-5细胞、SNU5细胞、SKMEL3细胞
RIN-m Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MOLM-13细胞、DMS 53细胞、BMU-S1细胞
HFTF Cells;背景说明:胚胎;眼;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HeLa细胞、AX-Mel细胞、NCIH1048细胞
HGF-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PAN 02细胞、SNU-869细胞、JIMT-1细胞
HPS1205 Cells(提供STR鉴定图谱)
JS2 TLR4-/- T399I Cells(提供STR鉴定图谱)
MDCC-UD14 Cells(提供STR鉴定图谱)
ND42506 Cells(提供STR鉴定图谱)
Psi-CRIP-MFGmIL-10 Cells(提供STR鉴定图谱)
Ubigene HeLa NCOR2 KO Cells(提供STR鉴定图谱)
XP13OS Cells(提供STR鉴定图谱)
HG01955 Cells(提供STR鉴定图谱)
U87-MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1385细胞、C3H10T1-2细胞、C33-A细胞
EB-3 Cells;背景说明:该细胞源自一名3岁患有Burkitt's淋巴瘤的黑人男孩的B淋巴细胞,EBNA阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:SW 1783细胞、SK-NM-C细胞、Y3-Ag 1.2.3细胞
NUGC-3 Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-735细胞、MOLT 16细胞、HTR-8/SV neo细胞
Hs294 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:混合星状和多边形;相关产品有:HIT细胞、COLO 201细胞、MDA-MB435细胞
MPP89 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HNEpC细胞、MBdSMC细胞、KS-SLK细胞
Human Fetal Thymocyte-8810 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC-1569细胞、H-69细胞、KATO III细胞
SK-Hep1 Cells;背景说明:SK-HEP-1细胞系已被鉴定为内皮来源。该细胞系为异倍体女性人(XX),染色体在亚三倍体范围内。在裸鼠中,它能形成与肝癌相一致的大细胞癌;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hepatoma 22细胞、YH细胞、Mink Lung细胞
TCC Sup Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Hs 695.T细胞、OCI-Ly01细胞、OCILY10细胞
RBL.2H3 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:BT20细胞、HCC 70细胞、GM346细胞
CCC-HIE-2 Cells;背景说明:胚胎;肠粘膜;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK293EBNA细胞、293 H细胞、Gejiu Lung Carcinoma-82细胞
HuH-28 Cells;背景说明:肝内胆管癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MCCAR细胞、Hs934T细胞、Jurkat, Clone E6-1细胞
Human podocyte Cells;背景说明:肾;足 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L(TK-)细胞、ECC12细胞、Daoy细胞
UC-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCFB(HCF)细胞、NCIH650细胞、DH-82细胞
293FT Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:Panc327细胞、BC3 H1细胞、C4-2 B细胞
HIT.T15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:M3 Clone M-3细胞、H7721细胞、Capan 1细胞
SNEC-MI Cells(提供STR鉴定图谱)
Alexander Cells;背景说明:该细胞系分泌乙肝病毒表面抗原(HBsAg)。 此细胞系原先被支原体污染,后用BM-cycline去除支原体;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:IPLB-SF-21-AE细胞、95D细胞、Sp2/0-Ag-14细胞
Blotchy fibroblast-11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Mevo细胞、OCI-AML-3细胞、UMC11细胞
BC-009 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MCF-7细胞、L-540细胞、Pt-K1细胞
NCl-H157 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:BC-027细胞、HITT15细胞、MDA-134细胞
TU 686 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NF639细胞、KOPN8细胞、N-Tera-2细胞
CHOK1 Cells;背景说明:1957年,PuckTT从成年中国仓鼠卵巢的活检组织建立了CHO细胞,CHO-K1是CHO的一个亚克隆。CHO-K1的生长需要脯酸。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SKRC20细胞、R 1610细胞、KHYG-1细胞
DAN-G Cells;背景说明:胰腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:aNK细胞、MC 3T3-E1细胞、Ramos-2G6-4C10细胞
Glioma 261 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:A431细胞、KYSE450细胞、WM 115细胞
Caki-1人肾透明细胞癌皮肤转移细胞全年复苏|已有STR图谱
3T3NIH Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LX-2细胞、HCC1395细胞、OV1-P细胞
RH-35 Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-2452细胞、Hs578Bst细胞、H-1436细胞
GM04680 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:PL11细胞、Hs940-T细胞、NCI-H1048细胞
HuLEC-5a Cells;背景说明:肺微血管;内皮细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:AG06814-M细胞、Karpas 422细胞、Hs 742.T细胞
NCIH345 Cells;背景说明:小细胞肺癌;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA-MB-175-VII细胞、T-ALL-1细胞、RH30SJ细胞
HCC1428 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:贴壁生长,偶尔上皮细胞液泡的形成;形态特性:上皮细胞样;相关产品有:CEM/C1细胞、MIO-M1细胞、WEHI3B细胞
BayGenomics ES cell line CSG330 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRU178 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC707 Cells(提供STR鉴定图谱)
KC-16 Cells(提供STR鉴定图谱)
PCRP-ZNF134-3E11 Cells(提供STR鉴定图谱)
MT-W9/PL Cells(提供STR鉴定图谱)
" "PubMed=571047
Fogh J.
Cultivation, characterization, and identification of human tumor cells with emphasis on kidney, testis, and bladder tumors.
Natl. Cancer Inst. Monogr. 49:5-9(1978)
PubMed=6244232
Williams R.D.
Human urologic cancer cell lines.
Invest. Urol. 17:359-363(1980)
PubMed=6935474; DOI=10.1093/jnci/66.2.239
Wright W.C., Daniels W.P., Fogh J.
Distinction of seventy-one cultured human tumor cell lines by polymorphic enzyme analysis.
J. Natl. Cancer Inst. 66:239-247(1981)
PubMed=7017212; DOI=10.1093/jnci/66.6.1003
Pollack M.S., Heagney S.D., Livingston P.O., Fogh J.
HLA-A, B, C and DR alloantigen expression on forty-six cultured human tumor cell lines.
J. Natl. Cancer Inst. 66:1003-1012(1981)
PubMed=7459858
Rousset M., Zweibaum A., Fogh J.
Presence of glycogen and growth-related variations in 58 cultured human tumor cell lines of various tissue origins.
Cancer Res. 41:1165-1170(1981)
PubMed=6582512; DOI=10.1073/pnas.81.2.568; PMCID=PMC344720
Mattes M.J., Cordon-Cardo C., Lewis J.L. Jr., Old L.J., Lloyd K.O.
Cell surface antigens of human ovarian and endometrial carcinoma defined by mouse monoclonal antibodies.
Proc. Natl. Acad. Sci. U.S.A. 81:568-572(1984)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2041050; DOI=10.1093/jnci/83.11.757
Monks A., Scudiero D.A., Skehan P., Shoemaker R.H., Paull K.D., Vistica D.T., Hose C.D., Langley J., Cronise P., Vaigro-Wolff A., Gray-Goodrich M., Campbell H., Mayo J.G., Boyd M.R.
Feasibility of a high-flux anticancer drug screen using a diverse panel of cultured human tumor cell lines.
J. Natl. Cancer Inst. 83:757-766(1991)
PubMed=7591954; DOI=10.1111/j.1349-7006.1995.tb03087.x; PMCID=PMC5920923
Kinoshita H., Yamada H., Ogawa O., Kakehi Y., Osaka M., Nakamura E., Mishina M., Habuchi T., Takahashi R., Sugiyama T., Yoshida O.
Contribution of chromosome 9p21-22 deletion to the progression of human renal cell carcinoma.
Jpn. J. Cancer Res. 86:795-799(1995)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::aid-mc5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=10723130; DOI=10.1038/sj.onc.1203449
Alimov A., Kost-Alimova M., Liu J., Li C.-D., Bergerheim U.S.R., Imreh S., Klein G., Zabarovsky E.R.
Combined LOH/CGH analysis proves the existence of interstitial 3p deletions in renal cell carcinoma.
Oncogene 19:1392-1399(2000)
PubMed=10929426; DOI=10.1007/s002400000103
Shintaku I., Kawagoe N., Yutani S., Hoshi S., Orikasa S., Yoshizumi O., Itoh K.
Expression of the SART1 tumor rejection antigen in renal cell carcinoma.
Urol. Res. 28:178-184(2000)
PubMed=11146448; DOI=10.1002/1097-0215(200002)9999:9999<::aid-ijc1034>3.0.CO;2-S
Kondo K.-i., Yao M., Kobayashi K., Ota S., Yoshida M., Kaneko S., Baba M., Sakai N., Kishida T., Kawakami S., Uemura H., Nagashima Y., Nakatani Y., Hosaka M.
PTEN/MMAC1/TEP1 mutations in human primary renal-cell carcinomas and renal carcinoma cell lines.
Int. J. Cancer 91:219-224(2001)
PubMed=15604581; DOI=10.1159/000081597
Ikemoto S., Sugimura K., Yoshida N., Kuratsukuri K., Wada S., Nakatani T.
Comparative antitumor activity of 5-fluorouracil and 5'-deoxy-5-fluorouridine in combination with interferon-alpha in renal cell carcinoma cell lines.
Urol. Int. 73:348-353(2004)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=17212712; DOI=10.1111/j.1399-0039.2006.00725.x
Komohara Y., Suekane S., Noguchi M., Matsuoka K., Yamada A., Itoh K.
Expression of APOBEC3G in kidney cells.
Tissue Antigens 69:95-98(2007)
PubMed=17804913; DOI=10.1159/000107804
Glube N., Giessl A., Wolfrum U., Langguth P.
Caki-1 cells represent an in vitro model system for studying the human proximal tubule epithelium.
Nephron Exp. Nephrol. 107:e47-e56(2007)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=22949125; DOI=10.1002/ijc.27822
Pawlowski R., Muhl S.M., Sulser T., Krek W., Moch H., Schraml P.
Loss of PBRM1 expression is associated with renal cell carcinoma progression.
Int. J. Cancer 132:E11-E17(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24477694; DOI=10.1007/s00432-014-1593-7
Tanaka T., Torigoe T., Hirohashi Y., Sato E., Honma I., Kitamura H., Masumori N., Tsukamoto T., Sato N.
Hypoxia-inducible factor (HIF)-independent expression mechanism and novel function of HIF prolyl hydroxylase-3 in renal cell carcinoma.
J. Cancer Res. Clin. Oncol. 140:503-513(2014)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26972028; DOI=10.1016/j.jprot.2016.03.008
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Identification of glycosylphosphatidylinositol-anchored proteins and omega-sites using TiO2-based affinity purification followed by hydrogen fluoride treatment.
J. Proteomics 139:77-83(2016)
PubMed=27141528; DOI=10.1016/j.dib.2016.04.001; PMCID=PMC4838930
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Data for identification of GPI-anchored peptides and omega-sites in cancer cell lines.
Data Brief 7:1302-1305(2016)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=27993170; DOI=10.1186/s12943-016-0565-8; PMCID=PMC5168717
Brodaczewska K.K., Szczylik C., Fiedorowicz M., Porta C., Czarnecka A.M.
Choosing the right cell line for renal cell cancer research.
Mol. Cancer 15:83.1-83.15(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28489074; DOI=10.1038/ncomms15165; PMCID=PMC5436135
Sinha R., Winer A.G., Chevinsky M.S., Jakubowski C., Chen Y.-B., Dong Y.-Y., Tickoo S.K., Reuter V.E., Russo P., Coleman J.A., Sander C., Hsieh J.J.-D., Hakimi A.A.
Analysis of renal cancer cell lines from two major resources enables genomics-guided cell line selection.
Nat. Commun. 8:15165.1-15165.10(2017)
PubMed=30260228; DOI=10.1021/acs.jproteome.8b00538
Knott M.E., Manzi M., Zabalegui N., Salazar M.O., Puricelli L.I., Monge M.E.
Metabolic footprinting of a clear cell renal cell carcinoma in vitro model for human kidney cancer detection.
J. Proteome Res. 17:3877-3888(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"
![Caki-1[人肾透明细胞癌细胞]](https://img.chemicalbook.com/SupplyImg/2019-08-28/Large/201908280906063948758.jpg)