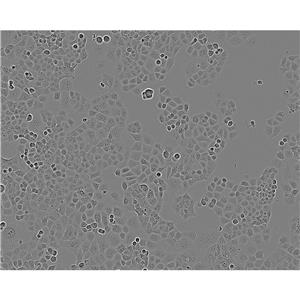
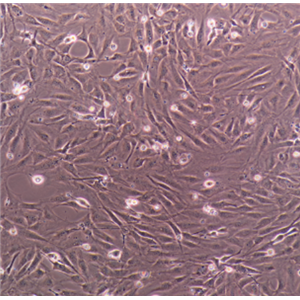
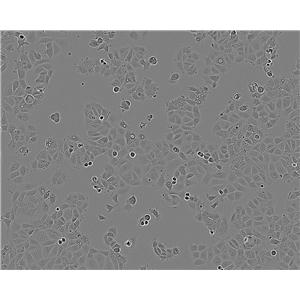
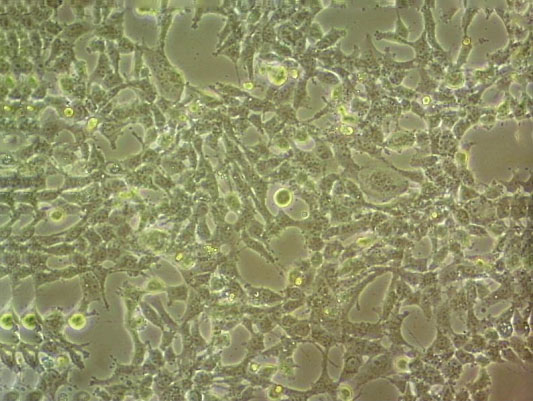
"MIA PaCa-2人胰腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
当T25瓶复苏细胞收到货时,请观察好细胞状态后,将T25细胞瓶壁进行75%酒精消毒,将T25瓶置于37度培养箱放置2-4h,以便稳定细胞状态,当细胞密度达80%-90%,即可进行首次传代培养;干冰运输的细胞冻存管收到货后,需立即转入液氮保存或直接进行复苏(第三天换液并检查复苏细胞密度,以便进行下一步)。 能够在实验室条件下进行大量培养和繁殖。这种细胞系在分子生物学和生物技术研究中十分常用。
换液周期:每周2-3次
TCCPAN2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HLEC细胞、QGP-1细胞、OP9细胞
SU-DHL1 Cells;背景说明:间变性大细胞淋巴瘤;胸腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs695细胞、HEC1B细胞、Mono-Mac-6细胞
A101D Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SNU-C2B细胞、NCI-A549细胞、B6Tert-1细胞
MIA PaCa-2人胰腺癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
公司细胞系主要引进ATCC、DSMZ、JCRB、KCLB、RIKEN、ECACC等细胞库,细胞系体外培养,它们会成长为单层细胞,附着或紧贴在培养瓶上,或悬浮在体外的溶液中,细胞系复苏周期短,公司细胞系状态良好,饱满,有光泽等优点。EDTA的作用:许多人不用胰酶,只用EDTA,或者用胰酶/EDTA联合作用。这里要明白,胰酶切割细胞外基质的一些负责粘连和附着的蛋白,而EDTA通过螯合Ca离子,作用于整联蛋白的活性,所以EDTA的作用更加温和。有的人在胰酶里添加一些EDTA,或者对付特别难消化的细胞,添加多一些EDTA,就是这个道理。一般不要试图延长消化时间(如果10min还消化不下来的话),而应该想其它办法。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
MIA PaCa-2人胰腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
C6661 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:MOLP-8细胞、THLE-3细胞、NS1细胞
SK-RC 52 Cells;背景说明:肾癌;纵隔膜转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BLO11细胞、McCoy细胞、Vertebral Cancer of the Prostate细胞
Jurkat (clone E6-1) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW756细胞、CEM-CCRF (CAMR)细胞、MOLT-16细胞
K7M2 wt Cells;背景说明:骨肉瘤;肺转移;雌性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Caov-4细胞、J-774A.1细胞、Jeko1细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞培养无菌操作步骤和注意事项:1.打开无菌工作台及净化室的紫外灯,消毒半小时以上。2.进入净化室前先关闭紫外灯,打开超净台风机,等待30min以上,以排尽臭氧。3.穿HAO隔离衣,戴HAO口罩,帽子。4.风淋2分钟。5.0.1%水泡手,擦干。6.点燃酒精灯;超净台内应避免放入过多的物品;使用的吸管,滴管,试管,培养瓶等均事先灭菌。7.打开各类瓶盖前先过火,以固定灰尘;打开的瓶口、试管口过火焰,镊子使用前应经火焰烧灼。8.水平式风机的超净台,应使瓶口斜置,应尽量避免瓶口敞开直立。9.同一根吸管或滴管不应连续用于几个不同的细胞系;吸取培养基的吸管应离开培养瓶或试管口0.5cm,避免伸入培养瓶口或试管口;以防止细胞系的互相混杂污染。10.漏在培养瓶上或台上的体,立即用酒精棉球擦净。11.操作完毕后恢复工作台面。
RGC5 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HeLa.S3细胞、L- cell细胞、SW839细胞
ID8 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:IPI-2I细胞、RA 1细胞、FHs 74 Int细胞
624 MEL Cells;背景说明:黑色素瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNUC1细胞、CCD-841CoN细胞、COLO-684细胞
NCIH548 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO 679细胞、HGC27细胞、NCI-H838细胞
Plaepi 34 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MDA PCa 2b细胞、NBL_S细胞、CA922细胞
H2141 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:Reuber H-35细胞、LP-1细胞、NCI-Hut 125细胞
Panc 3.27 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:J774细胞、P3JHR-1细胞、HS852.T细胞
GM03573A Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Panc 5.04细胞、NOMO1细胞、LLC-PK1细胞
LO2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:IOSE 29细胞、NR 8383细胞、Hs 695T细胞
143B Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代;每周换液2-3次;生长特性:贴壁生长;形态特性:混合型;相关产品有:JVM2细胞、Hs-695-T细胞、V 79-4细胞
RPMI 7951 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:X63.Ag8.653细胞、BEAS 2B细胞、SU-DHL-4细胞
Y1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CaSki细胞、28SC细胞、LS-123细胞
HPBALL Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HBL100细胞、FDCP1细胞、3T3-F442A细胞
Be-Wo Cells;背景说明:取自人绒癌脑转移组织,在仓鼠颊囊移植传代8年。利用移植瘤组织进行体外培养,建立细胞系。利用不同传代方法建立了不同亚系,JEG-3是其衍生克隆。该细胞可以产生雌激素、孕激素、雌酮、雌二醇、雌三醇、hCG、胎盘催乳素、角蛋白。;传代方法:1:3传代,3-4天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BC-022细胞、SIRC细胞、3T3(A31)细胞
PF-382 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:HuT-102细胞、Hs888 Lu细胞、SNU-398细胞
Medical University of Graz-Chordoma 1 Cells;背景说明:骶骨脊索瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEH2细胞、CA46细胞、B95-8细胞
Centre Antoine Lacassagne-51 Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LWnt-3A细胞、HT 1197.T细胞、SRA01/04细胞
Abcam HEK293T RRAGA KO Cells(提供STR鉴定图谱)
AG12502 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRB168 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XE404 Cells(提供STR鉴定图谱)
BY00332 Cells(提供STR鉴定图谱)
CS97iHD180-n1 Cells(提供STR鉴定图谱)
DA04456 Cells(提供STR鉴定图谱)
DD3420 Cells(提供STR鉴定图谱)
GM02408 Cells(提供STR鉴定图谱)
hEM15A Cells;背景说明:子宫内膜间质细胞;永生化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H2591细胞、U-251_MG细胞、M-14细胞
MIA PaCa-2人胰腺癌细胞代次低|培养基|送STR图谱
LX-2 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MDA-MB-435S细胞、OVCA433_Bast细胞、GM04678细胞
HL7702 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HPAFII细胞、MCAEC细胞、H-1373细胞
FAT Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Dunn LM8细胞、IPLB-SF 21AE细胞、H1650_CO细胞
8305C Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MKN28细胞、NCI-H719细胞、SK-MEL2细胞
SKML-28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:CT26.CL25细胞、TE32细胞、PG13细胞
4C11 [Mouse hybridoma against human FcRn] Cells(提供STR鉴定图谱)
OVMANA Cells;背景说明:详见相关文献介绍;传代方法:5 x 10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SK-MEL-5细胞、LIXC-002细胞、PA317细胞
KP-N-NS Cells;背景说明:源自脑转移灶的肾上腺神经母细胞瘤。;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CV 1细胞、NTERA-2cl.D1细胞、OVCAR420细胞
MonoMac6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:DMS53细胞、SUM190细胞、NCI-H810细胞
ATDC-5 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:KMST-6细胞、OVCAR-3细胞、MBMEC细胞
IMR-90 Cells;背景说明:W.W. Nichols及其同事从一位16周女婴的肺中取材,建立了人二倍体成纤维细胞株IMR-90。分裂潜能,病毒感受性和其它性质都得到了充分研究,因而这株细胞可以作为WI-38或其它标准人肺细胞株的替代株。有报道称这株细胞在表现出衰老前可倍增58次。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:LC1细胞、WILL2细胞、MIN6细胞
CP-70 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MHCC97-H细胞、SNU-5细胞、UMC-11细胞
MRMT-1 Cells;背景说明:乳腺癌;SD;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HOEC细胞、SNU-638细胞、CORL88细胞
Saos2 Cells;背景说明:该细胞是FoghJ和TrempeG分离和鉴定的众多人类肿瘤细胞系中的一种;该细胞来自一位11岁的白人女性的骨肉瘤组织。患者经过放疗以及甲喋呤、阿霉素、长春新碱、环磷酰胺和aramycin-C等多种药物治疗。该细胞在免疫抑制小鼠中不致瘤,细胞表达表皮生长因子EGF受体、转化生长因子β(1型和2型)受体。;传代方法:1:2-1:4传代;每周1-2次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:BHK21细胞、CL MC/9细胞、RH-30细胞
GM22565 Cells(提供STR鉴定图谱)
HAP1 MFSD8 (-) 2 Cells(提供STR鉴定图谱)
HT(H9) Cells;背景说明:H9细胞是HUT78(ATCCTIB161)的克隆系(Callo,RC,etal)。细胞表面带有CD3、CD4标记。研究表明,该细胞系对人体免疫缺陷病毒(HIV-1)敏感,可用于检测、分离和增殖HIV-1,也可用于其它人类Tcell病毒的研究。;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Jurkat E6-1细胞、PLA-801C细胞、CG-4细胞
CCD 1112SK Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OVCAR-432细胞、OV1-P细胞、H-1650细胞
H2073 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:253J B-V细胞、CSQT-2细胞、McA-RH8994细胞
SUPT-1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:RIN14B细胞、Hs852T细胞、Cates 1B细胞
UCD-MLA-144 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW-900细胞、L132细胞、SK MEL 5细胞
SK-ES1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:5传代;每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:HS-5细胞、PSN1细胞、PFSK1细胞
MARC 145 Cells;背景说明:胚肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CoCL2细胞、CP70细胞、T-T细胞
HCE-T Cells;背景说明:角膜上皮细胞;Ad-SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Panc-8_13细胞、CBRH-7919细胞、HTh-74细胞
HPSI1116i-aimh_5 Cells(提供STR鉴定图谱)
KCB 88002 Cells(提供STR鉴定图谱)
MG1.19 Cells(提供STR鉴定图谱)
NIMHi009-A Cells(提供STR鉴定图谱)
RAW 264.7 Rab29 KO Cells(提供STR鉴定图谱)
Ubigene HEK293 WDR24 KO Cells(提供STR鉴定图谱)
WMUi023-A Cells(提供STR鉴定图谱)
HeLa S3 ATF4 KO Cells(提供STR鉴定图谱)
H322 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:P3-x63-Ag8 653细胞、Jiyoye细胞、293F细胞
WERI-Rb 1 Cells;背景说明:WERI-Rb-I细胞株是1974年R.M. McFall 和 T.W. Sery建立的两株人眼癌细胞系中的一株。 细胞能在Difco Bacto-Agar中存活但不形成克隆。 扫描电镜显示在表面囊泡,板状伪足和微绒毛在数量上和频率上的改变。 细胞分化研究,肿瘤治疗的动物模型和生化评价都涉及这株细胞。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:圆形细胞聚集成葡萄状;相关产品有:C4-2细胞、RK13细胞、Y3-Ag1,2,3细胞
CD18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:LUDLU 1细胞、MARC-145细胞、HCC0827细胞
NCI-H165 Cells;背景说明:非小细胞肺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H69C细胞、MC-4细胞、HPF细胞
NCI-H716 Cells;背景说明:从一位经5-尿嘧啶治疗的患者腹水中得到的细胞建立了这个细胞株。 与其它结直肠癌细胞系不同,这株细胞有多巴脱羧酶,细胞质中有核心致密的内分泌型颗粒。 这株细胞不表达TAG-72 或CA19-9抗原,也不生成癌胚抗原(CEA);传代方法:1:3—1:6传代,每周换液2—3次;生长特性:悬浮生长,聚团,少数贴壁;形态特性:上皮细胞样;相关产品有:GM 637细胞、NT2-D1细胞、PK13细胞
NCI-H716 Cells;背景说明:从一位经5-尿嘧啶治疗的患者腹水中得到的细胞建立了这个细胞株。 与其它结直肠癌细胞系不同,这株细胞有多巴脱羧酶,细胞质中有核心致密的内分泌型颗粒。 这株细胞不表达TAG-72 或CA19-9抗原,也不生成癌胚抗原(CEA);传代方法:1:3—1:6传代,每周换液2—3次;生长特性:悬浮生长,聚团,少数贴壁;形态特性:上皮细胞样;相关产品有:GM 637细胞、NT2-D1细胞、PK13细胞
NW-MEL-38 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LL/2(LLc1)细胞、D324 Med细胞、NCIH1944细胞
NCI H69 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:悬浮生长,聚团;形态特性:聚团悬浮;相关产品有:SU.86.86细胞、CAL51细胞、SNU-387细胞
H-1755 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCIH2342细胞、COLO-680N细胞、OCI-AML-3细胞
ATN1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NOR 10细胞、GM-215细胞、H-727细胞
NCIH820 Cells;背景说明:乳头状肺腺癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HBE细胞、SNU-668细胞、SKBr3细胞
NCIH929 Cells;背景说明:详见相关文献介绍;传代方法:保持细胞密度在5×105—1×106 cells/ml之间,每周换液2—3次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:U-251MG细胞、HuCCT-1细胞、MARC145细胞
MC3T3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3-NS1/1-Ag4-1细胞、Panc 02细胞、COLO 201细胞
MeWo Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:OCI-Ly3细胞、HBVSMC细胞、OPM-2细胞
SCC-9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Ls-174-T细胞、TU 686细胞、A2780/CP70细胞
STAN116i-148C4 Cells(提供STR鉴定图谱)
HLE Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PLC细胞、D10G41细胞、NB4细胞
WM115mel Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Panc203细胞、HTori-3.1细胞、UCLA RO-81A-1细胞
BBE Cells;背景说明:详见相关文献介绍;传代方法:1:6—1:10传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Kupffer细胞、Y1细胞、HuCCT-1细胞
CCC-HEH-2 Cells;背景说明:心肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-GT-4细胞、Tn5 B1-4细胞、MESSADX5细胞
QSG-7701 Cells;背景说明:该细胞系来自35岁女性的肝癌癌旁组织。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SW 626细胞、Y3-Ag1.2.3细胞、RPTC细胞
C666-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:AM细胞、WI-38细胞、AU-Mel细胞
MIA PaCa-2人胰腺癌细胞代次低|培养基|送STR图谱
L-540 Cells;背景说明:霍奇金淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:FRO 81-2细胞、NCI-H2405细胞、RL-95-2细胞
MDA-MB-231-GFP Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EJ细胞、LuCL4细胞、BTI-Tn-5B1-4细胞
SLMT1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L 428细胞、SK-N-F1细胞、HEC-1-B细胞
WB F344 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:T-HSC细胞、HOP62细胞、P3-X63-Ag8.653细胞
NHEK Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MDA 435细胞、KP-2细胞、H-1048细胞
EA hy 926 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:GM00637B细胞、NCIH1155细胞、Sp 2817细胞
NCIH28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Human Pancreatic Duct Epithelial细胞、NTera 2/cl.D1细胞、MX1细胞
HS-695T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:VP 267细胞、KRC-Y细胞、FRTL-5细胞
BayGenomics ES cell line RRF397 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG611 Cells(提供STR鉴定图谱)
EJ-6-2-Bam-6a Cells(提供STR鉴定图谱)
mSG-PAC1 Cells(提供STR鉴定图谱)
TRP 7.4 Cells(提供STR鉴定图谱)
Ma-31 Cells(提供STR鉴定图谱)
" "PubMed=7809022; DOI=10.1097/00006676-199409000-00018
Sumi S., Beauchamp R.D., Townsend C.M. Jr., Pour P.M., Ishizuka J., Thompson J.C.
Lovastatin inhibits pancreatic cancer growth regardless of RAS mutation.
Pancreas 9:657-661(1994)
PubMed=7961102; DOI=10.1111/j.1349-7006.1994.tb02898.x; PMCID=PMC5919355
Suwa H., Yoshimura T., Yamaguchi N., Kanehira K., Manabe T., Imamura M., Hiai H., Fukumoto M.
K-ras and p53 alterations in genomic DNA and transcripts of human pancreatic adenocarcinoma cell lines.
Jpn. J. Cancer Res. 85:1005-1014(1994)
PubMed=8026879; DOI=10.1002/ijc.2910580207
Berrozpe G., Schaeffer J., Peinado M.A., Real F.X., Perucho M.
Comparative analysis of mutations in the p53 and K-ras genes in pancreatic cancer.
Int. J. Cancer 58:185-191(1994)
PubMed=8194712; DOI=10.1016/0016-5085(94)90422-7
Simon B., Weinel R., Hohne M., Watz J., Schmidt J., Kortner G., Arnold R.
Frequent alterations of the tumor suppressor genes p53 and DCC in human pancreatic carcinoma.
Gastroenterology 106:1645-1651(1994)
PubMed=21607521; DOI=10.3892/or.1.6.1223
Iguchi H., Morita R., Yasuda D., Takayanagi R., Ikeda Y., Takada Y., Shimazoe T., Nawata H., Kono A.
Alterations of the p53 tumor-suppressor gene and ki-ras oncogene in human pancreatic cancer-derived cell-lines with different metastatic potential.
Oncol. Rep. 1:1223-1227(1994)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::AID-MC5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=10027410; DOI=10.1016/S0002-9440(10)65298-4; PMCID=PMC1850008
Ghadimi B.M., Schrock E., Walker R.L., Wangsa D., Jauho A., Meltzer P.S., Ried T.
Specific chromosomal aberrations and amplification of the AIB1 nuclear receptor coactivator gene in pancreatic carcinomas.
Am. J. Pathol. 154:525-536(1999)
PubMed=11115575; DOI=10.3892/or.8.1.89
Sun C.-L., Yamato T., Furukawa T., Ohnishi Y., Kijima H., Horii A.
Characterization of the mutations of the K-ras, p53, p16, and SMAD4 genes in 15 human pancreatic cancer cell lines.
Oncol. Rep. 8:89-92(2001)
PubMed=11169957; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1014>3.0.CO;2-U
Wallrapp C., Hahnel S., Boeck W., Soder A., Mincheva A., Lichter P., Leder G., Gansauge F., Sorio C., Scarpa A., Gress T.M.
Loss of the Y chromosome is a frequent chromosomal imbalance in pancreatic cancer and allows differentiation to chronic pancreatitis.
Int. J. Cancer 91:340-344(2001)
PubMed=11169959; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1049>3.0.CO;2-C
Sirivatanauksorn V., Sirivatanauksorn Y., Gorman P.A., Davidson J.M., Sheer D., Moore P.S., Scarpa A., Edwards P.A.W., Lemoine N.R.
Non-random chromosomal rearrangements in pancreatic cancer cell lines identified by spectral karyotyping.
Int. J. Cancer 91:350-358(2001)
PubMed=11787853; DOI=10.1007/s004280100474
Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=12692724; DOI=10.1007/s00428-003-0784-4
Sipos B., Moser S., Kalthoff H., Torok V., Lohr J.-M., Kloppel G.
A comprehensive characterization of pancreatic ductal carcinoma cell lines: towards the establishment of an in vitro research platform.
Virchows Arch. 442:444-452(2003)
PubMed=14695172
Iacobuzio-Donahue C.A., Ashfaq R., Maitra A., Adsay N.V., Shen-Ong G.L.-C., Berg K., Hollingsworth M.A., Cameron J.L., Yeo C.J., Kern S.E., Goggins M.G., Hruban R.H.
Highly expressed genes in pancreatic ductal adenocarcinomas: a comprehensive characterization and comparison of the transcription profiles obtained from three major technologies.
Cancer Res. 63:8614-8622(2003)
PubMed=15770730; DOI=10.3748/wjg.v11.i10.1521; PMCID=PMC4305696
Ma J.-H., Patrut E., Schmidt J., Knaebel H.-P., Buchler M.W., Marten A.
Synergistic effects of interferon-alpha in combination with chemoradiation on human pancreatic adenocarcinoma.
World J. Gastroenterol. 11:1521-1528(2005)
PubMed=16912165; DOI=10.1158/0008-5472.CAN-06-0721
Calhoun E.S., Hucl T., Gallmeier E., West K.M., Arking D.E., Maitra A., Iacobuzio-Donahue C.A., Chakravarti A., Hruban R.H., Kern S.E.
Identifying allelic loss and homozygous deletions in pancreatic cancer without matched normals using high-density single-nucleotide polymorphism arrays.
Cancer Res. 66:7920-7928(2006)
PubMed=17254797; DOI=10.1016/j.biologicals.2006.10.001
Azari S., Ahmadi N., Jeddi-Tehrani M., Shokri F.
Profiling and authentication of human cell lines using short tandem repeat (STR) loci: report from the National Cell Bank of Iran.
Biologicals 35:195-202(2007)
PubMed=18298655; DOI=10.1111/j.1582-4934.2008.00289.x; PMCID=PMC3828895
Pilarsky C., Ammerpohl O., Sipos B., Dahl E., Hartmann A., Wellmann A., Braunschweig T., Lohr J.-M., Jesenofsky R., Friess H., Wente M.N., Kristiansen G., Jahnke B., Denz A., Ruckert F., Schackert H.K., Kloppel G., Kalthoff H., Saeger H.-D., Grutzmann R.
Activation of Wnt signalling in stroma from pancreatic cancer identified by gene expression profiling.
J. Cell. Mol. Med. 12:2823-2835(2008)
PubMed=18380791; DOI=10.1111/j.1349-7006.2008.00779.x; PMCID=PMC11158928
Suzuki A., Shibata T., Shimada Y., Murakami Y., Horii A., Shiratori K., Hirohashi S., Inazawa J., Imoto I.
Identification of SMURF1 as a possible target for 7q21.3-22.1 amplification detected in a pancreatic cancer cell line by in-house array-based comparative genomic hybridization.
Cancer Sci. 99:986-994(2008)
PubMed=18575732; DOI=10.3892/or.20.1.155
Kawaoka T., Oka M., Takashima M., Ueno T., Yamamoto K., Yahara N., Yoshino S., Hazama S.
Adoptive immunotherapy for pancreatic cancer: cytotoxic T lymphocytes stimulated by the MUC1-expressing human pancreatic cancer cell line YPK-1.
Oncol. Rep. 20:155-163(2008)
CLPUB00416
Oberlin L.
Treatment of pancreatic carcinoma cell lines in vitro and vivo with a monoclonal antibody against the transferrin receptor.
Thesis VMD (2009); Justus-Liebig-Universitat Giessen; Giessen; Germany
PubMed=19077451; DOI=10.1159/000178871
Harada T., Chelala C., Crnogorac-Jurcevic T., Lemoine N.R.
Genome-wide analysis of pancreatic cancer using microarray-based techniques.
Pancreatology 9:13-24(2009)
PubMed=19188929; DOI=10.1038/tpj.2008.20
Guo J., Anderson M.G., Tapang P., Palma J.P., Rodriguez L.E., Niquette A.L., Li J.-L., Bouska J.J., Wang G., Semizarov D., Albert D.H., Donawho C.K., Glaser K.B., Shah O.J.
Identification of genes that confer tumor cell resistance to the aurora B kinase inhibitor, AZD1152.
Pharmacogenomics J. 9:90-102(2009)
PubMed=20037478; DOI=10.4161/cbt.8.21.9685; PMCID=PMC2824894
Kent O.A., Mullendore M.E., Wentzel E.A., Lopez-Romero P., Tan A.-C., Alvarez H., West K.M., Ochs M.F., Hidalgo M., Arking D.E., Maitra A., Mendell J.T.
A resource for analysis of microRNA expression and function in pancreatic ductal adenocarcinoma cells.
Cancer Biol. Ther. 8:2013-2024(2009)
PubMed=19904223; DOI=10.1097/MPA.0b013e3181bc44dd; PMCID=PMC2835986
Xiao J., Lee W.-N.P., Zhao Y.-C., Cao R., Go V.L.-W., Recker R.R., Wang Q., Xiao G.G.-S.
Profiling pancreatic cancer-secreted proteome using 15N amino acids and serum-free media.
Pancreas 39:e17-e23(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20418756; DOI=10.1097/MPA.0b013e3181c15963; PMCID=PMC2860631
Deer E.L., Gonzalez-Hernandez J., Coursen J.D., Shea J.E., Ngatia J.G., Scaife C.L., Firpo M.A., Mulvihill S.J.
Phenotype and genotype of pancreatic cancer cell lines.
Pancreas 39:425-435(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
DOI=10.4172/2324-9293.1000104
Wagenhauser M.U., Ruckert F., Niedergethmann M., Grutzmann R., Saeger H.-D.
Distribution of characteristic mutations in native ductal adenocarcinoma of the pancreas and pancreatic cancer cell lines.
Cell Biol. Res. Ther. 2:1000104.1-1000104.5(2013)
PubMed=23386380; DOI=10.1002/jcb.24506
Bose B., Shenoy P.S.
Non insulin producing cell line, MIA PaCa-2 is rendered insulin producing in vitro via mesenchymal epithelial transition.
J. Cell. Biochem. 114:1642-1652(2013)
PubMed=25167228; DOI=10.1038/bjc.2014.475; PMCID=PMC4453732
Hamidi H., Lu M., Chau K., Anderson L., Fejzo M.S., Ginther C., Linnartz R., Zubel A., Slamon D.J., Finn R.S.
KRAS mutational subtype and copy number predict in vitro response of human pancreatic cancer cell lines to MEK inhibition.
Br. J. Cancer 111:1788-1801(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25485960; DOI=10.4161/15384047.2014.986967; PMCID=PMC4623403
Samulitis B.K., Pond K.W., Pond E., Cress A.E., Patel H., Wisner L., Patel C., Dorr R.T., Landowski T.H.
Gemcitabine resistant pancreatic cancer cell lines acquire an invasive phenotype with collateral hypersensitivity to histone deacetylase inhibitors.
Cancer Biol. Ther. 16:43-51(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26884312; DOI=10.1038/srep21648; PMCID=PMC4756684
Gradiz R., Silva H.C., Carvalho L., Botelho M.F., Mota-Pinto A.
MIA PaCa-2 and PANC-1 -- pancreas ductal adenocarcinoma cell lines with neuroendocrine differentiation and somatostatin receptors.
Sci. Rep. 6:21648-21648(2016)
PubMed=27067801; DOI=10.1186/s12885-016-2297-y; PMCID=PMC4828819
Miura K., Kimura K., Amano R., Yamazoe S., Ohira G., Murata A., Nishio K., Hasegawa T., Yashiro M., Nakata B., Ohira M., Hirakawa K.
Establishment and characterization of new cell lines of anaplastic pancreatic cancer, which is a rare malignancy: OCUP-A1 and OCUP-A2.
BMC Cancer 16:268.1-268.13(2016)
PubMed=27259358; DOI=10.1074/mcp.M116.058313; PMCID=PMC4974343
Humphrey E.S., Su S.-P., Nagrial A.M., Hochgrafe F., Pajic M., Lehrbach G.M., Parton R.G., Yap A.S., Horvath L.G., Chang D.K., Biankin A.V., Wu J.-M., Daly R.J.
Resolution of novel pancreatic ductal adenocarcinoma subtypes by global phosphotyrosine profiling.
Mol. Cell. Proteomics 15:2671-2685(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31037374; DOI=10.1007/s00216-019-01814-1
Lagies S., Schlimpert M., Braun L.M., Kather M., Plagge J., Erbes T., Wittel U.A., Kammerer B.
Unraveling altered RNA metabolism in pancreatic cancer cells by liquid-chromatography coupling to ion mobility mass spectrometry.
Anal. Bioanal. Chem. 411:6319-6328(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)"