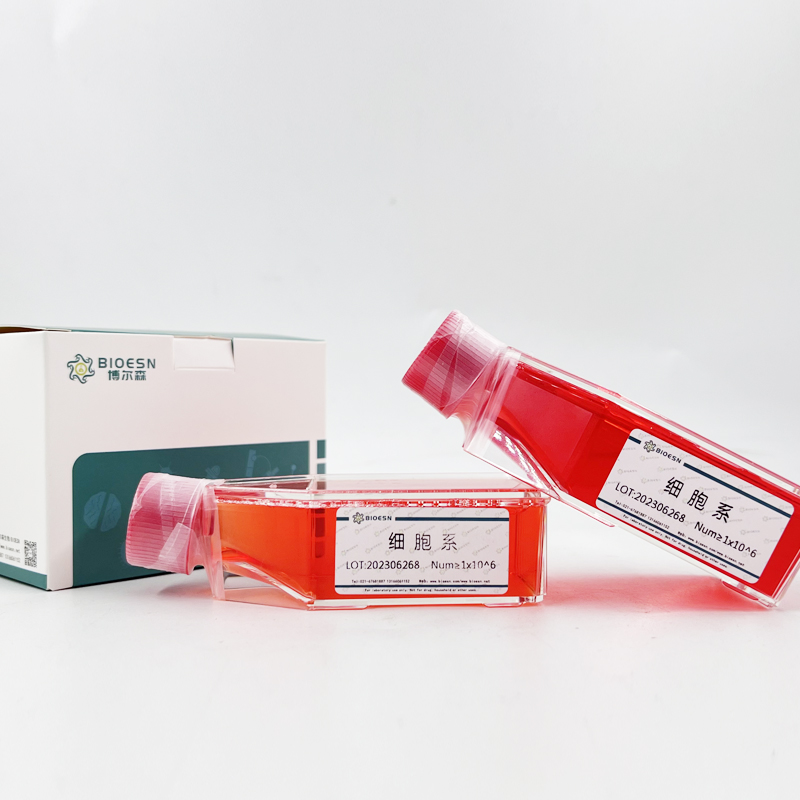
"U251人胶质瘤细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
L-M (TK-) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Sp2/0细胞、SKGT2细胞、SKG IIIa细胞
GM03570E Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SU-DHL-2细胞、WM-239A细胞、Lu-165细胞
H1563 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:GM00346细胞、T98细胞、MT2细胞
U251人胶质瘤细胞代次低|培养基|送STR图谱
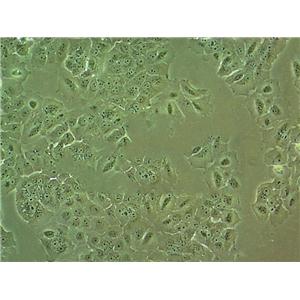
背景信息:最初是从一名患有恶性胶质瘤的患者的脑组织中分离并培养的。是一种人胶质瘤细胞系,表达多种胶质瘤标志蛋白,如胶质纤维酸性蛋白(GFAP)、巢蛋白(Nestin)、神经细胞黏附分子(NCAM)和SALL4。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞(adherent cells):该类细胞的生长必须有可以贴附的支持物表面,细胞依靠自身分泌的或培养基中提供的贴附因子才能在该表面上生长,繁殖;当细胞在该表面生长后,一般形成两种形态,即成纤维样细胞性或上皮样细胞;其生长过程分为游离期、贴壁期、潜伏期、对数期、平台期和衰退期。悬浮细胞(suspension cell):不贴附于生长物,细胞呈圆形,呈单个细胞或者细小细胞团,悬浮细胞生长空间大,传代方便,能够大量增殖。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
U251人胶质瘤细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
IPLB-SF 21 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HITT15细胞、RIN Cl-5F细胞、CLL-CII细胞
MEG01 Cells;背景说明:MEG-01细胞株源自一位CML患者成巨核细胞转换期的骨髓细胞。细胞质因子VIII和表面球蛋白IIb/IIIa,-Schiff(PAS)反应,α醋酸酯酶和酸性酶阳性。髓过氧化物酶,α酯酶,化醋酸AS-D酯酶和碱性酶阴性。用单克隆抗体BA-1(抗B细胞,粒性白细胞),HPL-3(抗球蛋白IIb/IIIa)和20.3(抗单核细胞,血小板)染色成阳性。其他淋巴和骨髓类抗体成阴性。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:OVCAR4细胞、Hs706T细胞、Murine Long bone Osteocyte-Y4细胞
Large Cell Lung Cancer-103H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:WPMY-1细胞、A2780CP细胞、NCI-SNU-423细胞
McARH7777 Cells;背景说明:肝癌;雌性;Buffalo;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIADR.RES细胞、MRCV细胞、MASMC细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:成纤维细胞样
正确的细胞复苏需知事项:细胞冻存HAO了,接下来要注意什么问题呢?没错,就是记得到时间了,拿出来复苏。那么,细胞复苏的过程中又有哪些该注意的事项呢?细胞活力和形态检查的作用何在?活力检查——千万不要使用不健康的细胞,可能有污染(真菌、支原体等),如果发现有污染毫不犹豫的丢弃!形态检查——检查细胞的固有形态和生长行为。冻存细胞:补充新的培养——在您开始冻存细胞的前一天补充新的培养。在细胞长至70%单层时收获细胞,计数活细胞数,用冻存调整细胞密度~5 x106 s/ml (根据不同的细胞类型调整);冻存——用冻存洗细胞并用冻存重悬细胞,有不同类型的冻存,根据细胞类型选择Zui合适的冻存(常用的冻存成分有):5-10% DMSO——注意确保DMSO不含有其他的毒性物质;5-15%甘油;如果细胞在无血清培养基内生长,应在50%条件培养基内(细胞在无血清培养基内生长24小时)内冻存和复苏。在冻存管上标记HAO细胞类型,日期,冻存人等信息,并保证每冻存管不超过1.5ml。放入罐之前记录冻存管的数量和位置。以Zui快的速度转移冻存管知罐内,因此,此步骤ZuiHAO使用干冰,或者把冻存管浸入装有的小盒内。此外还要注意,在冻存管上没有足够的空间记录细胞的详细信息,做HAO记录是非常非常重要的!还有一个Zui重要的,一定要在异地的罐内保存同样的一份细胞,以免其中的一个罐出现问题!细胞正确的复苏方式和正确的冻存方式同样重要,熟记以下要点:当从罐内取出细胞时,有可能会出现冻存管破裂的情况,使用保护面罩和防护服十分必要;其实,细胞复苏只是一个简单的实验,不过这其中却不可避免有一些需要注意的细节,不然,也不一定会尽如人意。例如说,人身健康方面:一定要记得做HAO防冻工作,戴上护目镜;尽量降低DMSO对细胞的损伤等等。
Chang Cells Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Z138细胞、G-361细胞、KYSE450细胞
BC-028 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FHC细胞、H446细胞、IAR20细胞
NCIH196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:YES 2细胞、AZ521细胞、ZR75-1细胞
HIT T15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SuDHL 8细胞、DH82细胞、TCMK-1细胞
SKNFI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Acanthosis Nigricans 3rd attempt-CArcinoma细胞、NCI-H1155细胞、WBF344细胞
CC-LP-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC-2279细胞、TE-11细胞、HEC1B细胞
P3-NS1/1-Ag4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:NHLF细胞、NCI-H524细胞、Caco 2细胞
KU1919 Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MC9细胞、NMC-G1细胞、Panc 04.03细胞
BEND Cells;背景说明:子宫内膜;上皮细胞;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SJ-Rh 30细胞、HBMEC细胞、MOLP-2细胞
T2 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HCC-38细胞、Mel-624细胞、Hs-695-T细胞
DLD1 Cells;背景说明:DLD-1是1977-1979年间D.L.Dexter和同事分离的两株结直肠腺癌细胞株中的一株。在ATCC和其它地方进行的DNAfingerprinting和染色体组型分析表明这株细胞与HCT-15(CCL-225)相似,说明这两者是来自同一个人的不同克隆。他们的遗传起源可通过DNAfingerprinting证实,但染色体组型分析显示它们缺乏染色体标记一致改变或数目上一致改变。细胞的CSAp阴性(CSAp-)。DLD-1细胞的p53抗原表达呈阳性(p53抗原产生了一个C->;传代方法:消化5分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:AgC11x3A细胞、Roswell Park Memorial Institute 1846细胞、Mahlavu细胞
UMNSAH-DF1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:PC10细胞、BE(2)-M17细胞、CAL-12T细胞
GAK Cells;背景说明:详见相关文献介绍;传代方法:8.4 x 10^4 cells / sq.cm.;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:RSC-364细胞、C-28I2细胞、SVEC4-10细胞
GCT0404 Cells;背景说明:骨巨细胞瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HS766T细胞、Roswell Park Memorial Institute 1788细胞、SL1细胞
UCH1 Cells;背景说明:骶骨脊索瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HBZY1细胞、H-650细胞、P3J HR1-K细胞
Tu686 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:UCLA-RO 81细胞、SUM102细胞、RPMI8402细胞
C127 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:M 1细胞、PTK 1细胞、TE6细胞
18b Cells(提供STR鉴定图谱)
Abcam HeLa LIF KO Cells(提供STR鉴定图谱)
AG23105 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRF552 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG757 Cells(提供STR鉴定图谱)
C0814 Cells(提供STR鉴定图谱)
CW60418 Cells(提供STR鉴定图谱)
DA05667 Cells(提供STR鉴定图谱)
GL15 Cells(提供STR鉴定图谱)
J-774A.1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CEM-CCRF细胞、OCI-Ly 19细胞、PC12(高分化)细胞
U251人胶质瘤细胞代次低|培养基|送STR图谱
WEHI 3 Cells;背景说明:白血病;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI-H292细胞、Hs27F细胞、Clone 15 HL-60细胞
HAEC Cells;背景说明:主动脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Baby Hamster Kidney 21细胞、A-72细胞、DU4475细胞
RPTEC/TERT 1 Cells;背景说明:肾;近端小管上皮;HGNC-TERT转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RFL6细胞、NCIH1568细胞、Psi2 DAP细胞
BE(2)-C Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H209细胞、MESSA/Dx5细胞、COLO679细胞
NCI-H1437 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周换液2次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H2141细胞、MV 3细胞、KBM-7/Hap8细胞
211F3 Cells(提供STR鉴定图谱)
SKM1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:QBC-939细胞、KBM-5细胞、NCIH647细胞
PC 61-5-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM637细胞、NIH-3T3细胞、BNL.CL2细胞
KMST-6 Cells;背景说明:胚成纤维细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DF-1细胞、TK+/- (clone 3.7.2C)细胞、NCIH441细胞
NCI-H711 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3/X63/Ag8细胞、BEL-7405细胞、EoL-1-cell细胞
H4IIE Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-1975细胞、M109细胞、C4I细胞
SW1088 Cells;背景说明:星形胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P3X63NS1细胞、SUDHL-2细胞、SW-1573细胞
HPAF2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:EB-2细胞、NCI-H1373细胞、NCIH358细胞
HBL1 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U2-OS细胞、VSMC细胞、D324细胞
h1RPE7 Cells(提供STR鉴定图谱)
HAP1 PUSL1 (-) 2 Cells(提供STR鉴定图谱)
32D clone3 Cells;背景说明:骨髓淋巴瘤;C3H/HeJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A9细胞、4T1-A细胞、PANC-02-03细胞
HCT116 Cells;背景说明:结肠腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Rat podocyte细胞、MKN 45细胞、SK-N-BE(2)C细胞
MMVECs Cells;背景说明:心肌微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MFE 280细胞、MOLT16细胞、JIMT1细胞
Hx147 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:343 MG细胞、BJAB细胞、COLO 394细胞
MOLT3 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI-H660细胞、P3 (Jiyoye)细胞、293 c18细胞
KASUMI6 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成髓细胞;相关产品有:NCIH1435细胞、OCI/AML-3细胞、RMG1细胞
C-4-I Cells;背景说明:宫颈鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Capan2细胞、beta-TC6细胞、Jijoye细胞
DBTRG-05MG Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:EBTr细胞、HB611细胞、NCI-SNU-216细胞
HuH7-SLC22A9-KO-c5 Cells(提供STR鉴定图谱)
L1210/TZR Cells(提供STR鉴定图谱)
MSY00350IN Cells(提供STR鉴定图谱)
OKT4 Cells(提供STR鉴定图谱)
Royan C29 Cells(提供STR鉴定图谱)
UAB-iPSC-PKD2-cKO NP/null #1 Cells(提供STR鉴定图谱)
UMGi140-A Cells(提供STR鉴定图谱)
HG02061 Cells(提供STR鉴定图谱)
Mel RM Cells;背景说明:黑色素瘤;神经节转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MFC细胞、HEK-293-F细胞、H2135细胞
RPMI6666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCRF/CEM-C7细胞、MPP89细胞、DH 82细胞
H22 Cells;背景说明:1952年,前苏联医学科学院肿瘤研究所以C3HA小鼠诱发的H22肝癌实体瘤的瘤细胞悬液,昆明种小鼠皮下移植后,转腹水瘤。经检测,该瘤株在Km小鼠、615小鼠、C57BL/6小鼠、BALB/C小鼠体内可以形成实体瘤和腹水瘤。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:KYSE 70细胞、Y3Ag1.2.3细胞、Glioma 261细胞
H-1755 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCIH2342细胞、COLO-680N细胞、OCI-AML-3细胞
HLF-a Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TW01细胞、MDAMB330细胞、COLO.205细胞
HLF-a Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TW01细胞、MDAMB330细胞、COLO.205细胞
SBC-3 Cells;背景说明:小细胞肺癌;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCC4006细胞、NCIH716细胞、RM-1细胞
GC1-SPG Cells;背景说明:精原细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SaOS-2细胞、SUM149-PT细胞、H-1048细胞
CT26WT Cells;背景说明:CT26细胞是被N-亚硝基-N-甲基脲烷(NNMU)诱导得到的未分化的小鼠结肠癌细胞,该细胞的一个克隆形成的细胞系被命名为CT26.WT。CT26.WT被逆转录病毒载体LXSN稳定转化形成了一个致死性的亚克隆CT26.CL25,这一病毒载体含有lacZ基因、编码肿瘤相关抗原(TAA)和beta半乳糖苷酶。CT26.WT和CT26.CL25细胞在小鼠中生长速度和致死率都很相似,不同的是CT26.CL25细胞可以表达肿瘤相关抗原和beta半乳糖苷酶,因此这两株细胞可以联合用于免疫治疗和宿主免疫反应的研究。 ;传代方法:1:4-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RAW-264.7细胞、hRMECs细胞、P3JHR-1细胞
SKG IIIa Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HS578T细胞、NIH:OVCAR-3细胞、WEHI 3细胞
HITT15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CNE-1细胞、Lu-65细胞、HR-1细胞
HLEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:50.B1细胞、HEK293A细胞、KE-39细胞
CCRF-CEM/S Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Panc05.04细胞、PANC0403细胞、MLOY4细胞
NCIH2196 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCT/Taxo1细胞、CAOV4细胞、RKO_AS45细胞
231-luc Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BTT739细胞、HCC-2185细胞、RPE1-hTERT细胞
t-HUE4 Cells(提供STR鉴定图谱)
Mink Lung Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1915细胞、HCC-44细胞、LNCaP clone FGC细胞
NCIH196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:YES 2细胞、AZ521细胞、ZR75-1细胞
MZCRC1 Cells;背景说明:甲状腺髓样癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PNT-1A细胞、YD15细胞、HLF-a细胞
H-1522 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KNS-42细胞、CAL-78细胞、Hs 766.T细胞
U-251 MG Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:AML-EOL-1细胞、AMO1细胞、PC615.3细胞
B35 Cells;背景说明:神经母细胞瘤;BDIX;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HT-3细胞、NCI-H1688细胞、MeT-5A细胞
U251人胶质瘤细胞代次低|培养基|送STR图谱
OCI-Ly18 Cells;背景说明:弥漫大B细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HCC9724细胞、C-643细胞、LoVo细胞
NCIH1694 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:GC1-SPG细胞、S3-HeLa细胞、Hs 636 T细胞
SO-RB50 Cells;背景说明:视网膜母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U118-MG细胞、NP69SV40T细胞、H-660细胞
4T1 Cells;背景说明:4T1是从410.4瘤株中未经诱变筛得的6-鸟嘌噙抗性细胞株。当注射到BALB/c小鼠中时,4T1自发产生高转移肿瘤,可转移到肺,肝,淋巴结和大脑,同时在注射部位形成始发灶。诱导转移时不需要摘除始发灶。4T1细胞在BALB/c小鼠中的生长与转移特性与人体中的乳腺癌十分相近。这种肿瘤是人VI期乳腺癌的动物模型。4T1-诱导的肿瘤在手术后及未手术情况下转移的动力学相近,可以用作手术后及未手术模型。跟其他肿瘤模型相比,由于4T1的抗6-鸟嘌噙特性,微小的转移细胞团(少到仅仅1个)也可以在许多远端器官中检测到。没必要数淋巴结或称重器官。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BSC40细胞、8402细胞、Giant Cell Tumor细胞
GM01232E Cells;背景说明:1971年1月,该细胞由ReidTW及其同事从病人右眼切除的肿瘤进行原代培养建立而成,此病人有很强的视网膜母细胞瘤的母系家族遗传性。该细胞的超微结构,如核膜内折、三层膜结构、大的被膜小泡、环孔板、微管、中心粒、基粒等都与原始肿瘤相似。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形,成簇生长;相关产品有:P3.times.63 Ag8.653细胞、FL-83B细胞、NCTC-929细胞
A431 Cells;背景说明:该细胞源自一位患有皮肤鳞状细胞癌的85岁女性,是GiardDJ等人建立的一系列细胞株中的一株。该细胞在免疫抑制小鼠体内可成瘤,在琼脂上培养可形成克隆;是一个超三倍体人细胞株。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HS688AT细胞、HEC-1-A细胞、DH 82细胞
RAW-264.7 Cells;背景说明:单核巨噬细胞白血病;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MHHCALL2细胞、SF-763细胞、MFM223细胞
P3-X63-Ag8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:DHL4细胞、H661细胞、SKRC-20细胞
BayGenomics ES cell line RRR472 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA004 Cells(提供STR鉴定图谱)
HyCyte B16-F10-Luc Cells(提供STR鉴定图谱)
PCRP-JAZF1-1C2 Cells(提供STR鉴定图谱)
GFP2 Cells(提供STR鉴定图谱)
HPSI0115i-iuad_3 Cells(提供STR鉴定图谱)
" "PubMed=608846; DOI=10.1111/j.1601-5223.1978.tb01267.x
Mark J., Westermark B., Ponten J., Hugosson R.
Banding patterns in human glioma cell lines.
Hereditas 87:243-260(1977)
PubMed=833871; DOI=10.1093/jnci/58.2.209
Fogh J., Wright W.C., Loveless J.D.
Absence of HeLa cell contamination in 169 cell lines derived from human tumors.
J. Natl. Cancer Inst. 58:209-214(1977)
PubMed=77569; DOI=10.1111/j.1399-0039.1978.tb01259.x
Espmark J.A., Ahlqvist-Roth L., Sarne L., Persson A.
Tissue typing of cells in culture. III. HLA antigens of established human cell lines. Attempts at typing by the mixed hemadsorption technique.
Tissue Antigens 11:279-286(1978)
PubMed=450131; DOI=10.1038/279797a0
Day R.S. 3rd, Ziolkowski C.H.J.
Human brain tumour cell strains with deficient host-cell reactivation of N-methyl-N'-nitro-N-nitrosoguanidine-damaged adenovirus 5.
Nature 279:797-799(1979)
PubMed=6260907; DOI=10.1097/00005072-198105000-00001
Bigner D.D., Bigner S.H., Ponten J., Westermark B., Mahaley M.S. Jr., Ruoslahti E., Herschman H.R., Eng L.F., Wikstrand C.J.
Heterogeneity of genotypic and phenotypic characteristics of fifteen permanent cell lines derived from human gliomas.
J. Neuropathol. Exp. Neurol. 40:201-229(1981)
PubMed=6582512; DOI=10.1073/pnas.81.2.568; PMCID=PMC344720
Mattes M.J., Cordon-Cardo C., Lewis J.L. Jr., Old L.J., Lloyd K.O.
Cell surface antigens of human ovarian and endometrial carcinoma defined by mouse monoclonal antibodies.
Proc. Natl. Acad. Sci. U.S.A. 81:568-572(1984)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=3675803
Bigner S.H., Bjerkvig R., Laerum O.D., Muhlbaier L.H., Bigner D.D.
DNA content and chromosomes in permanent cultured cell lines derived from malignant human gliomas.
Anal. Quant. Cytol. Histol. 9:435-444(1987)
PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2041050; DOI=10.1093/jnci/83.11.757
Monks A., Scudiero D.A., Skehan P., Shoemaker R.H., Paull K.D., Vistica D.T., Hose C.D., Langley J., Cronise P., Vaigro-Wolff A., Gray-Goodrich M., Campbell H., Mayo J.G., Boyd M.R.
Feasibility of a high-flux anticancer drug screen using a diverse panel of cultured human tumor cell lines.
J. Natl. Cancer Inst. 83:757-766(1991)
PubMed=7693337
Li H., Hamou M.-F., de Tribolet N., Jaufeerally R., Hofmann M., Diserens A.-C., Van Meir E.G.
Variant CD44 adhesion molecules are expressed in human brain metastases but not in glioblastomas.
Cancer Res. 53:5345-5349(1993)
PubMed=7763724
Satoh M., Takeuchi M.
Cross-contamination of cell lines as revealed by DNA fingerprinting in the IFO animal cell bank.
Res. Commun. Inst. Ferment. 16:18-23(1993)
PubMed=8104691
Pershouse M.A., Stubblefield E., Hadi A., Killary A.M., Yung W.-K.A., Steck P.A.
Analysis of the functional role of chromosome 10 loss in human glioblastomas.
Cancer Res. 53:5043-5050(1993)
DOI=10.1016/B978-0-12-333530-2.50005-8
Nister M., Westermark B.
Human glioma cell lines.
(In book chapter) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.17-42; Academic Press; New York; USA (1994)
PubMed=8878451; DOI=10.1111/j.1349-7006.1996.tb02118.x; PMCID=PMC5921198
Zhang S., Endo S., Koga H., Ichikawa T., Feng X., Onda K., Washiyama K., Kumanishi T.
A comparative study of glioma cell lines for p16, p15, p53 and p21 gene alterations.
Jpn. J. Cancer Res. 87:900-907(1996)
PubMed=9090379; DOI=10.1038/ng0497-356
Steck P.A., Pershouse M.A., Jasser S.A., Yung W.-K.A., Lin H., Ligon A.H., Langford L.A., Baumgard M.L., Hattier T., Davis T., Frye C., Hu R., Swedlund B., Teng D.H.-F., Tavtigian S.V.
Identification of a candidate tumour suppressor gene, MMAC1, at chromosome 10q23.3 that is mutated in multiple advanced cancers.
Nat. Genet. 15:356-362(1997)
PubMed=9220028; DOI=10.1016/S0303-7207(97)00080-4
Sharif T.R., Luo W., Sharif M.
Functional expression of bombesin receptor in most adult and pediatric human glioblastoma cell lines; role in mitogenesis and in stimulating the mitogen-activated protein kinase pathway.
Mol. Cell. Endocrinol. 130:119-130(1997)
PubMed=9230885; DOI=10.1093/jnci/89.14.1036
Gomez-Manzano C., Fueyo J., Kyritsis A.P., McDonnell T.J., Steck P.A., Levin V.A., Yung W.-K.A.
Characterization of p53 and p21 functional interactions in glioma cells en route to apoptosis.
J. Natl. Cancer Inst. 89:1036-1044(1997)
PubMed=9614553; DOI=10.1038/sj.gt.3300605
Tada M., Sakuma S., Iggo R.D., Saya H., Sawamura Y., Fujiwara T., Roth J.A.
Monitoring adenoviral p53 transduction efficiency by yeast functional assay.
Gene Ther. 5:339-344(1998)
PubMed=9842975; DOI=10.1002/(SICI)1097-0215(19981218)79:6<640::AID-IJC15>3.0.CO;2-z
Weller M., Rieger J., Grimmel C., Van Meir E.G., De Tribolet N., Krajewski S., Reed J.C., von Deimling A., Dichgans J.
Predicting chemoresistance in human malignant glioma cells: the role of molecular genetic analyses.
Int. J. Cancer 79:640-644(1998)
DOI=10.1007/0-306-46861-1_11
Ali-Osman F.
Brain tumors.
(In book chapter) Human cell culture. Vol. 2. Cancer cell lines part 2; Masters J.R.W., Palsson B.O. (eds.); pp.167-184; Kluwer Academic Publishers; New York; USA (1999)
PubMed=10416987; DOI=10.1111/j.1750-3639.1999.tb00536.x; PMCID=PMC8098486
Ishii N., Maier D., Merlo A., Tada M., Sawamura Y., Diserens A.-C., Van Meir E.G.
Frequent co-alterations of TP53, p16/CDKN2A, p14ARF, PTEN tumor suppressor genes in human glioma cell lines.
Brain Pathol. 9:469-479(1999)
PubMed=10551321; DOI=10.1111/j.1349-7006.1999.tb00838.x; PMCID=PMC5926156
Zhang S.-J., Endo S., Ichikawa T., Yoshimura J., Onda K., Tanaka R., Washiyama K., Kumanishi T.
Rare-type mutations of MMAC1 tumor suppressor gene in human glioma cell lines and their tumors of origin.
Jpn. J. Cancer Res. 90:934-941(1999)
PubMed=10560660; DOI=10.1097/00005072-199911000-00007
Schmidt E.E., Ichimura K., Goike H.M., Moshref A., Liu L., Collins V.P.
Mutational profile of the PTEN gene in primary human astrocytic tumors and cultivated xenografts.
J. Neuropathol. Exp. Neurol. 58:1170-1183(1999)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=14614447; DOI=10.1038/sj.onc.1207198
Wischhusen J., Naumann U., Ohgaki H., Rastinejad F., Weller M.
CP-31398, a novel p53-stabilizing agent, induces p53-dependent and p53-independent glioma cell death.
Oncogene 22:8233-8245(2003)
PubMed=14655754; DOI=10.1111/j.1750-3639.2003.tb00479.x; PMCID=PMC8095903
Bahr O., Rieger J., Duffner F., Meyermann R., Weller M., Wick W.
P-glycoprotein and multidrug resistance-associated protein mediate specific patterns of multidrug resistance in malignant glioma cell lines, but not in primary glioma cells.
Brain Pathol. 13:482-494(2003)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=15900046; DOI=10.1093/jnci/dji133
Mashima T., Oh-hara T., Sato S., Mochizuki M., Sugimoto Y., Yamazaki K., Hamada J.-i., Tada M., Moriuchi T., Ishikawa Y., Kato Y., Tomoda H., Yamori T., Tsuruo T.
p53-defective tumors with a functional apoptosome-mediated pathway: a new therapeutic target.
J. Natl. Cancer Inst. 97:765-777(2005)
PubMed=16232199; DOI=10.1111/j.1349-7006.2005.00099.x; PMCID=PMC11159392
Saigusa K., Hashimoto N., Tsuda H., Yokoi S., Maruno M., Yoshimine T., Aoyagi M., Ohno K., Imoto I., Inazawa J.
Overexpressed Skp2 within 5p amplification detected by array-based comparative genomic hybridization is associated with poor prognosis of glioblastomas.
Cancer Sci. 96:676-683(2005)
PubMed=16391870; DOI=10.3892/or.15.2.463
Castigli E., Sciaccaluga M., Schiavoni G., Brozzi F., Fabiani R., Gorello P., Gianfranceschi G.L.
GL15 and U251 glioblastoma-derived human cell lines are peculiarly susceptible to induction of mitotic death by very low concentrations of okadaic acid.
Oncol. Rep. 15:463-470(2006)
PubMed=16697959; DOI=10.1016/j.ccr.2006.03.030
Lee J., Kotliarova S., Kotliarov Y., Li A.-G., Su Q., Donin N.M., Pastorino S., Purow B.W., Christopher N., Zhang W., Park J.K., Fine H.A.
Tumor stem cells derived from glioblastomas cultured in bFGF and EGF more closely mirror the phenotype and genotype of primary tumors than do serum-cultured cell lines.
Cancer Cell 9:391-403(2006)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=17595512; DOI=10.1159/000104150
Rieger J., Frank B., Weller M., Wick W.
Mechanisms of resistance of human glioma cells to Apo2 ligand/TNF-related apoptosis-inducing ligand.
Cell. Physiol. Biochem. 20:23-34(2007)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=19435942; DOI=10.1215/15228517-2009-025; PMCID=PMC2743214
Ichimura K., Pearson D.M., Kocialkowski S., Backlund L.M., Chan R., Jones D.T.W., Collins V.P.
IDH1 mutations are present in the majority of common adult gliomas but rare in primary glioblastomas.
Neuro-oncol. 11:341-347(2009)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20593219; DOI=10.1007/s11060-010-0283-9
Blough M.D., Beauchamp D.C., Westgate M.R., Kelly J.J.P., Cairncross J.G.
Effect of aberrant p53 function on temozolomide sensitivity of glioma cell lines and brain tumor initiating cells from glioblastoma.
J. Neurooncol. 102:1-7(2011)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22336246; DOI=10.1016/j.bmc.2012.01.017
Kong D.-X., Yamori T.
JFCR39, a panel of 39 human cancer cell lines, and its application in the discovery and development of anticancer drugs.
Bioorg. Med. Chem. 20:1947-1951(2012)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22570425; DOI=10.1093/neuonc/nos072; PMCID=PMC3367844
Bady P., Diserens A.-C., Castella V., Kalt S., Heinimann K., Hamou M.-F., Delorenzi M., Hegi M.E.
DNA fingerprinting of glioma cell lines and considerations on similarity measurements.
Neuro-oncol. 14:701-711(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=24810477; DOI=10.1002/cam4.219; PMCID=PMC4303149
Torsvik A., Stieber D., Enger P.O., Golebiewska A., Molven A., Svendsen A., Westermark B., Niclou S.P., Olsen T.K., Chekenya Enger M., Bjerkvig R.
U-251 revisited: genetic drift and phenotypic consequences of long-term cultures of glioblastoma cells.
Cancer Med. 3:812-824(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25894527; DOI=10.1371/journal.pone.0121314; PMCID=PMC4404347
Bausch-Fluck D., Hofmann A., Bock T., Frei A.P., Cerciello F., Jacobs A., Moest H., Omasits U., Gundry R.L., Yoon C., Schiess R., Schmidt A., Mirkowska P., Hartlova A.S., Van Eyk J.E., Bourquin J.-P., Aebersold R., Boheler K.R., Zandstra P.W., Wollscheid B.
A mass spectrometric-derived cell surface protein atlas.
PLoS ONE 10:E0121314-E0121314(2015)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27582061; DOI=10.1126/scitranslmed.aaf6853
Allen M., Bjerke M., Edlund H., Nelander S., Westermark B.
Origin of the U87MG glioma cell line: good news and bad news.
Sci. Transl. Med. 8:354re3.1-354re3.4(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29716531; DOI=10.1186/s12885-018-4394-6; PMCID=PMC5930953
Hegge B., Sjottem E., Mikkola I.
Generation of a PAX6 knockout glioblastoma cell line with changes in cell cycle distribution and sensitivity to oxidative stress.
BMC Cancer 18:496.1-496.19(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"