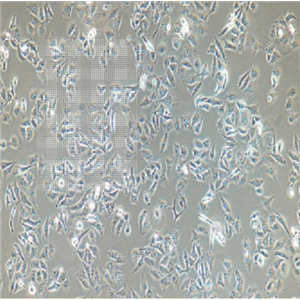
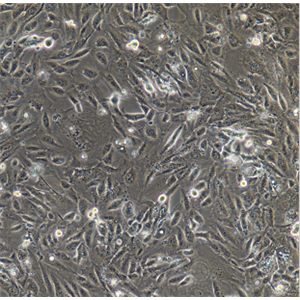
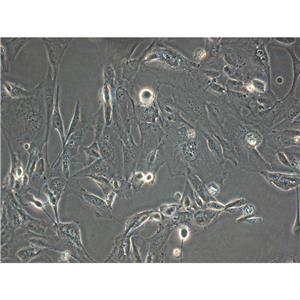
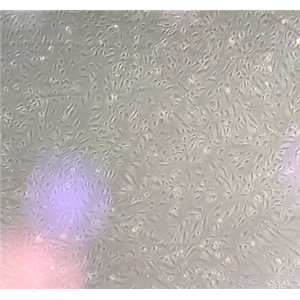
"HuH-7人肝癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
CORL51 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1299细胞、CF Pac1细胞、Hs746-T细胞
HTh-74 Cells;背景说明:未分化甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Mel-MeWo细胞、HSC-I细胞、YD-15细胞
MDAPCa-2b Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:TOV-112D细胞、U-373MG细胞、HS-683细胞
HuH-7人肝癌细胞代次低|培养基|送STR图谱
背景信息:是一种来源于人肝细胞癌(Hepatocellular Carcinoma, HCC)的细胞系,最初于1982年从一名56岁的日本男性患者的肝脏肿瘤中分离出来。据说,HuH-7细胞可产甲胎蛋白、胰酶α抗体、血浆铜蓝蛋白、纤维蛋白原、纤维粘连蛋白等。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
HuH-7人肝癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
RBL 2H3 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NCIH2023细胞、UPCI-SCC-90细胞、C4-2细胞
HRA19 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:UMNSAH-DF1细胞、SW-1463细胞、IGROV1细胞
TW-039 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RT-4细胞、102PT细胞、GalK 1细胞
LLC-PK-1 Cells;背景说明:肾;自发永生;Hampshire;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KHYG细胞、INS-1细胞、HEK293EBNA细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞株(系)的使用,为医学研究和测试工作带来了大的方便。但细胞的传代是有限制的,长期连续传代的细胞,不仅消耗大量的人力和物力,而且细胞的生长与形态等会有一定退变或转化,因而细胞失去原有的遗传性,有时还会由于细胞污染而造成传代中断,种子丢失。因此,在实际工作中常需冻存一定数量的细胞,以备替换使用。细胞冷冻与复苏是细胞培养 室的常规工作和通用技术。目前,细胞冻存Zui常用的技术是冷冻保存法,主要采用加适量保护剂的缓慢冷冻法冻存细胞。细胞在不加任何保护剂的情况下直接冷冻,细胞内外的水分会很快形成冰晶,从而引起一系列不良反应。如细胞脱水使局部电解质浓度增GAO,pH值改变,部分蛋白质由于上述原因而变性,引起细胞内部空间结构紊乱,溶酶体膜由此遭到损伤而释放出溶酶体酶,使细胞内结构成分造成破坏,线粒体肿胀,功能丢失,并造成能量代谢障碍。胞膜上的类脂蛋白复合体也易破坏引起细胞膜通透性的改变,使细胞内容物丢失。如果细胞内冰晶形成较多,随冷冻温度的降低,冰晶体积膨胀造成细胞核DNA空间构型发生不可逆的损伤,而致细胞死亡。因此,细胞冷冻技术的关键是尽可能地减少细胞内水分,减少细胞内冰晶的形成。采用甘油或二甲基亚砜作保护剂,这两种物质分子量小,溶解度大,易穿透细胞,可以使冰点下降,提GAO细胞膜对水的通透性,且对细胞无明显毒性。慢速冷冻方法又可使细胞内的水分渗出细胞外,减少胞内形成冰结晶的机会,从而减少冰晶对细胞的损伤。
Swiss 3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:JROECL 19细胞、624细胞、HNE2细胞
MB 157 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:UPCI:SCC090细胞、Z-138细胞、Hepatoma 22细胞
HEp-2 Cells;背景说明:最初认为这个细胞源自喉上皮癌,但随后通过同功酶分析、HeLa标记染色体和DNA指纹分析发现,起源细胞已被HeLa污染。 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Jurkat-E6细胞、HCC38细胞、beta TC6细胞
OVCA-433 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HOS细胞、NCC-IT细胞、Bowes melanoma cells细胞
A549-Taxol Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MLM细胞、KYSE410细胞、LNCaP-Clone-FGC细胞
ACCM Cells;背景说明:涎腺腺样囊性癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Rat Skin 1细胞、143B细胞、SW626细胞
AR4-2J Cells;背景说明:在肾上腺皮质激素刺激下可诱导出外分泌活性,并伴有广泛的内质网重构。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Epithelioma Papulosum Cyprini细胞、HNEpC细胞、HS0578T细胞
HPDEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO_320DM细胞、Jurkat Clone E6-1细胞、HRGEC细胞
LU65 Cells;背景说明:详见相关文献介绍;传代方法:1:10传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:130 T细胞、H2405细胞、SW 1573细胞
HCC-202 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:RT4细胞、NB1-RGB细胞、ND7/23细胞
U343 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HEK AD293细胞、FO [Mouse myeloma]细胞、SKNBE(2)细胞
293 F Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;悬浮生长;形态特性:上皮细胞样;相关产品有:Huh-7.5.1细胞、RS4-11细胞、U87细胞
HAPI Cells;背景说明:小胶质细胞;自发永生;Wistar;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:ARO细胞、H1944细胞、NCIH2227细胞
SKRC20 Cells;背景说明:肾癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C32 mel细胞、SV40-HCEC细胞、CCD-112CoN细胞
CCRF/CEM Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:M-1细胞、PL 5细胞、DHBE细胞
NCI522 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MDA-kb2细胞、Epithelioma Papulosum Cyprini细胞、OCI-LY-19细胞
NTERA2-D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TGBC11TKB细胞、H378细胞、BT-549细胞
Abcam A-549 SIRT2 KO Cells(提供STR鉴定图谱)
AF24 Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSH478 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRY604 Cells(提供STR鉴定图谱)
BCIRL/RP-HvE-CLG5 Cells(提供STR鉴定图谱)
CHO-K1/FLT3 Cells(提供STR鉴定图谱)
DA02739 Cells(提供STR鉴定图谱)
DA05210 Cells(提供STR鉴定图谱)
GCW28 Cells(提供STR鉴定图谱)
Hs 274 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代,每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:CFPAC-1细胞、TK-10细胞、AML 12细胞
HuH-7人肝癌细胞代次低|培养基|送STR图谱
MiaPaCa-2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:TW-039细胞、FET细胞、Potorous tridactylus Kidney 1细胞
SKOV-3 Cells;背景说明:SK-OV-3由G.Trempe和L.J.Old在1973年从卵巢肿瘤病人的腹水分离得到。 此细胞对肿瘤坏死因子和几种细胞毒性药物包括白喉毒素、顺铂和阿霉素均耐受。 在裸鼠中致瘤,且形成与卵巢原位癌一致的中度分化的腺癌。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:MOLT 3细胞、MADB-106细胞、G-292细胞
IEC-18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:McA-RH7777细胞、CTV1细胞、Kit 225细胞
H810 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:C-6细胞、L-929细胞、SW 756细胞
NCI-H1930 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:TCC Sup细胞、Hs695细胞、NCI-H1793细胞
18E9.G3.D7 Cells(提供STR鉴定图谱)
NCIH2347 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SW-48细胞、MFE296细胞、KOPN8细胞
LICCF Cells;背景说明:肝内胆管癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Emory University-3细胞、DB细胞、EJ1细胞
SK-LU-1 Cells;背景说明:该细胞系源于一位60岁的白人女性患者的肺腺癌组织。;传代方法:1:2传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:OCI-Ly 3细胞、LN 382细胞、211H细胞
SF763 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H510A细胞、MONO-MAC 6细胞、PBMC细胞
OCIAML5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Leukemia 1210细胞、HS-294细胞、HEL-92-1-7细胞
NTera2 cl.D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PaTu8988s细胞、253J B-V细胞、CoC1细胞
TCMK1 Cells;背景说明:肾小管;上皮细胞;SV40转化;C3H/Mai;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CMT.64细胞、OCI Ly1细胞、H-23细胞
RKO Cells;背景说明:RKO是一个低分化的结肠癌细胞系。RKO细胞含有野生型P53,但缺乏人甲状腺受体核受体(h-TRbeta1)。RKO细胞的P53蛋白的水平高于RKO-E6细胞。RKO细胞系是RKO-E6和RKO-AS45-1的亲本细胞系。该细胞系在裸鼠中成瘤,且在软琼脂中形成集落。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:WM451Lu细胞、SKML-28细胞、HSC(Human Schwann)细胞
HAP1 AKAP1 (-) 2 Cells(提供STR鉴定图谱)
HAP1 SIRT6 (-) 1 Cells(提供STR鉴定图谱)
IMR 32 Cells;背景说明:该细胞是1967年4月由NicholsWW,LeeJ和DwightS建立,来源于一名13月龄白人男婴腹部肿块,临床诊断为神经母细胞瘤,伴有极少部位的类器官样分化。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:存在两种细胞类型,小的神经母细胞样细胞和大的透明成纤维样细胞;相关产品有:RenCa细胞、MDA-MB-134 VI细胞、H1048细胞
PANC-08-13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SUM102细胞、JROECL21细胞、KYSE 150 KYSE150 Kyse150 KY150
细胞
BCaP 37 Cells;背景说明:源自一位48岁女性乳癌患者。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:LA-N-1细胞、PC615.3细胞、AG06814-N细胞
MARC-145 Cells;背景说明:胚肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MC-26细胞、HUVEC细胞、HOMEC细胞
DF1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:MD Anderson-Metastatic Breast-468细胞、H838细胞、MC3T3-E1 Subclone 14细胞
DBTRG-05MG Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:EBTr细胞、HB611细胞、NCI-SNU-216细胞
Panc 05.04 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KMST6细胞、CMEC/D3细胞、NUGC-4细胞
NFHIOSE-29 Cells;背景说明:卵巢;上皮细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCM356细胞、NCI-H2029细胞、NCIH1341细胞
HyCyte OCI-AML3 KO-hFGL2 Cells(提供STR鉴定图谱)
LCL-PI 92 Cells(提供STR鉴定图谱)
NAL1A clone C4E10 Cells(提供STR鉴定图谱)
P1 Cells(提供STR鉴定图谱)
RTM Cells(提供STR鉴定图谱)
Ubigene HCT 116 PAK6 KO Cells(提供STR鉴定图谱)
WAe001-A-6 Cells(提供STR鉴定图谱)
HG02861 Cells(提供STR鉴定图谱)
ARH77 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:H-9细胞、SNU407细胞、P19细胞
PT-K75 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:KBM5细胞、NCIH548细胞、HMVEC细胞
Hs940T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:A549/ATCC细胞、OCI AML5细胞、SGC-996细胞
JS1 Cells;背景说明:肝星形 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:786-O RCC细胞、GM07404A细胞、CHP-126细胞
P 815 Cells;背景说明:肥大细胞瘤;雄性;DBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SPCA1细胞、SNU354细胞、EU-3细胞
P 815 Cells;背景说明:肥大细胞瘤;雄性;DBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SPCA1细胞、SNU354细胞、EU-3细胞
SW 13 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCD18Co细胞、UMUC-14细胞、CAOV4细胞
SK-MEL-24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:HL 60细胞、EM-3细胞、NuTu 19细胞
Panc04.03 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NK92细胞、NF639细胞、Rabbit Kidney 13细胞
DR2R 1610 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GNM细胞、DLD 1细胞、Panc2_03细胞
P1-Raji Cells;背景说明:Raji细胞由PulvertaftRJV于1963年从一位11岁黑人男孩的左上颌骨的Burkitt淋巴瘤中分离建立的,是第一个人类造血系统的连续传代细胞,为B细胞起源。该细胞中含有EBV,需要在二级生物安全柜中操作;可作转染宿主。;传代方法:维持细胞浓度在4×105/ml-3×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:RCC-10RGB细胞、MG-HU-3细胞、OCI-AML-5细胞
P3/X63/Ag8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1666细胞、HFSF细胞、CAL-62细胞
NCI-H2108 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO 684细胞、D6P2T细胞、University of Arizona Cell Culture-893细胞
HOS Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞和上皮细胞的混合样;相关产品有:S16细胞、COLO680N细胞、MIA-PaCa-2细胞
H-1618 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:PCI:SG-231细胞、JROECL 33细胞、BC-019细胞
TER69 Cells(提供STR鉴定图谱)
Tu-212 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:SNU-475细胞、NCI-H526细胞、V79-4细胞
Hep-G2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Emory University-2细胞、FK81细胞、CCC-HSF-1细胞
H-841 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:5传代,;生长特性:混合型生长;形态特性:详见产品说明书;相关产品有:SUM 102PT细胞、MES-SA细胞、CEM/0细胞
BE2_C Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SJRH30细胞、HN4细胞、AE1201细胞
Hs-578Bst Cells;背景说明:乳腺 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MN 60细胞、P3-Jiyoye细胞、UMR-106细胞
LC1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PAa细胞、EA. hy 926细胞、AN3 CA细胞
HuH-7人肝癌细胞代次低|培养基|送STR图谱
PG-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SuDHL 5细胞、786-O细胞、Karpas299细胞
MM1 Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MV 4;11细胞、Tregs细胞、NIH-3T3细胞
Hs1.Tes Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:hCMEC/D3细胞、UT7细胞、RPTEC/TERT 1细胞
G-401 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NCIH889细胞、Merwin Plasma Cell tumor-11细胞、NCI-H1672细胞
MDA 231-LM2-4175 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:University of Michigan-Urothelial Carcinoma-14细胞、HBdSMC细胞、8305C_1细胞
MDA-MB 453 Cells;背景说明:该细胞系由CailleauR在1976年从一名48岁的患有转移性乳腺癌的白人女性的心包渗出液中分离建立的。该细胞表达FGF的受体。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:LoVo细胞、S-16细胞、IHH-4细胞
GP-293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LICR-LON-HN6-R细胞、ATDC5细胞、SUDHL-4细胞
NCI-H1693 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:H-2330细胞、HBZY 1细胞、B16F0细胞
BayGenomics ES cell line RRO094 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHB138 Cells(提供STR鉴定图谱)
H37-93 Cells(提供STR鉴定图谱)
Pas1CN5 Cells(提供STR鉴定图谱)
AH414-TC Cells(提供STR鉴定图谱)
HPCT-03-ts Cells(提供STR鉴定图谱)
" "PubMed=8224613; DOI=10.1096/fasebj.7.14.8224613
Puisieux A., Galvin K., Troalen F., Bressac B., Marcais C., Galun E., Ponchel F., Yakicier C., Ji J.-W., Ozturk M.
Retinoblastoma and p53 tumor suppressor genes in human hepatoma cell lines.
FASEB J. 7:1407-1413(1993)
PubMed=8389256; DOI=10.1093/carcin/14.5.987
Hsu I.-C., Tokiwa T., Bennett W.P., Metcalf R.A., Welsh J.A., Sun T.-T., Harris C.C.
p53 gene mutation and integrated hepatitis B viral DNA sequences in human liver cancer cell lines.
Carcinogenesis 14:987-992(1993)
PubMed=8835345; DOI=10.1002/(SICI)1096-9071(199602)48:2<133::AID-JMV3>3.0.CO;2-A
Tsuboi S., Nagamori S., Miyazaki M., Mihara K., Fukaya K.-i., Teruya K., Kosaka T., Tsuji T., Namba M.
Persistence of hepatitis C virus RNA in established human hepatocellular carcinoma cell lines.
J. Med. Virol. 48:133-140(1996)
DOI=10.11418/jtca1981.16.3_173
Mihara K., Miyazaki M., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ohashi R., Namba M.
The p53 gene status and other cellular characteristics of human cell lines maintained in our laboratory.
Tissue Cult. Res. Commun. 16:173-178(1997)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::AID-MC5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=9359923; DOI=10.18926/AMO/30789
Mihara K., Miyazaki M., Kondo T., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ishioka C., Namba M.
Yeast functional assay of the p53 gene status in human cell lines maintained in our laboratory.
Acta Med. Okayama 51:261-265(1997)
PubMed=10523694; DOI=10.3892/or.6.6.1267
Gao C., Ohashi R., Pu H., Inoue Y., Tsuji T., Miyazaki M., Namba M.
Yeast functional assay of the p53 gene status in 11 cell lines and 26 surgical specimens of human hepatocellular carcinoma.
Oncol. Rep. 6:1267-1271(1999)
PubMed=11152498; DOI=10.1128/JVI.75.3.1252-1264.2001; PMCID=PMC114031
Pietschmann T., Lohmann V., Rutter G., Kurpanek K., Bartenschlager R.F.W.
Characterization of cell lines carrying self-replicating hepatitis C virus RNAs.
J. Virol. 75:1252-1264(2001)
PubMed=12029633; DOI=10.1053/jhep.2002.33683
Yasui K., Arii S., Zhao C., Imoto I., Ueda M., Nagai H., Emi M., Inazawa J.
TFDP1, CUL4A, and CDC16 identified as targets for amplification at 13q34 in hepatocellular carcinomas.
Hepatology 35:1476-1484(2002)
PubMed=15708988; DOI=10.1128/JVI.79.5.2689-2699.2005; PMCID=PMC548482
Sumpter R.M. Jr., Loo Y.-M., Foy E.M., Li K., Yoneyama M., Fujita T., Lemon S.M., Gale M. Jr.
Regulating intracellular antiviral defense and permissiveness to hepatitis C virus RNA replication through a cellular RNA helicase, RIG-I.
J. Virol. 79:2689-2699(2005)
PubMed=15767549; DOI=10.1158/1535-7163.MCT-04-0234
Nakatsu N., Yoshida Y., Yamazaki K., Nakamura T., Dan S., Fukui Y., Yamori T.
Chemosensitivity profile of cancer cell lines and identification of genes determining chemosensitivity by an integrated bioinformatical approach using cDNA arrays.
Mol. Cancer Ther. 4:399-412(2005)
PubMed=16935386; DOI=10.1016/j.jhep.2006.05.019
Sun D.-X., Nassal M.
Stable HepG2- and Huh7-based human hepatoma cell lines for efficient regulated expression of infectious hepatitis B virus.
J. Hepatol. 45:636-645(2006)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23285155; DOI=10.1371/journal.pone.0052697; PMCID=PMC3527576
Murayama A., Sugiyama N., Yoshimura S., Ishihara-Sugano M., Masaki T., Kim S., Wakita T., Mishiro S., Kato T.
A subclone of HuH-7 with enhanced intracellular hepatitis C virus production and evasion of virus related-cell cycle arrest.
PLoS ONE 7:E52697-E52697(2012)
PubMed=23505090; DOI=10.1002/hep.26402
Wang K., Lim H.Y., Shi S., Lee J., Deng S.-B., Xie T., Zhu Z., Wang Y.-L., Pocalyko D., Yang W.J., Rejto P.A., Mao M., Park C.-K., Xu J.-C.
Genomic landscape of copy number aberrations enables the identification of oncogenic drivers in hepatocellular carcinoma.
Hepatology 58:706-717(2013)
PubMed=23887712; DOI=10.1038/ncomms3218; PMCID=PMC3731665
Nault J.-C., Mallet M., Pilati C., Calderaro J., Bioulac-Sage P., Laurent C., Laurent A., Cherqui D., Balabaud C., Zucman-Rossi J.
High frequency of telomerase reverse-transcriptase promoter somatic mutations in hepatocellular carcinoma and preneoplastic lesions.
Nat. Commun. 4:2218.1-2218.7(2013)
PubMed=24973239; DOI=10.1099/vir.0.065995-0
Richter M., Reimann I., Schirrmeier H., Kirkland P.D., Beer M.
The viral envelope is not sufficient to transfer the unique broad cell tropism of Bungowannah virus to a related pestivirus.
J. Gen. Virol. 95:2216-2222(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25574106; DOI=10.3748/wjg.v21.i1.311; PMCID=PMC4284350
Cevik D., Yildiz G., Ozturk M.
Common telomerase reverse transcriptase promoter mutations in hepatocellular carcinomas from different geographical locations.
World J. Gastroenterol. 21:311-317(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27329724; DOI=10.18632/oncotarget.10161; PMCID=PMC5216950
Watari K., Nishitani A., Shibata T., Noda M., Kawahara A., Akiba J., Murakami Y., Yano H., Kuwano M., Ono M.
Phosphorylation of mTOR Ser2481 is a key target limiting the efficacy of rapalogs for treating hepatocellular carcinoma.
Oncotarget 7:47403-47417(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29610054; DOI=10.1016/j.dmpk.2018.03.003; PMCID=PMC6309175
Shi J., Wang X.-W., Lyu L.-Y., Jiang H., Zhu H.-J.
Comparison of protein expression between human livers and the hepatic cell lines HepG2, Hep3B, and Huh7 using SWATH and MRM-HR proteomics: Focusing on drug-metabolizing enzymes.
Drug Metab. Pharmacokinet. 33:133-140(2018)
PubMed=29774518; DOI=10.1007/s13577-018-0212-3; PMCID=PMC6002425
Kasai F., Hirayama N., Ozawa M., Satoh M., Kohara A.
HuH-7 reference genome profile: complex karyotype composed of massive loss of heterozygosity.
Hum. Cell 31:261-267(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31063779; DOI=10.1053/j.gastro.2019.05.001
Caruso S., Calatayud A.-L., Pilet J., La Bella T., Rekik S., Imbeaud S., Letouze E., Meunier L., Bayard Q., Rohr-Udilova N., Peneau C., Grasl-Kraupp B., de Koning L., Ouine B., Bioulac-Sage P., Couchy G., Calderaro J., Nault J.-C., Zucman-Rossi J., Rebouissou S.
Analysis of liver cancer cell lines identifies agents with likely efficacy against hepatocellular carcinoma and markers of response.
Gastroenterology 157:760-776(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31378681; DOI=10.1016/j.ccell.2019.07.001; PMCID=PMC7505724
Qiu Z.-X., Li H., Zhang Z.-T., Zhu Z.-F., He S., Wang X.-J., Wang P.-C., Qin J.-J., Zhuang L.-P., Wang W., Xie F.-B., Gu Y., Zou K.-K., Li C., Li C., Wang C.-H., Cen J., Chen X.-T., Shu Y.-J., Zhang Z., Sun L.-L., Min L.-H., Fu Y., Huang X.-W., Lv H., Zhou H., Ji Y., Zhang Z.-G., Meng Z.-Q., Shi X.-L., Zhang H.-B., Li Y.-X., Hui L.-J.
A pharmacogenomic landscape in human liver cancers.
Cancer Cell 36:179-193.e11(2019)
PubMed=31395879; DOI=10.1038/s41467-019-11415-2; PMCID=PMC6687785
Yu K., Chen B., Aran D., Charalel J., Yau C., Wolf D.M., van 't Veer L.J., Butte A.J., Goldstein T., Sirota M.
Comprehensive transcriptomic analysis of cell lines as models of primary tumors across 22 tumor types.
Nat. Commun. 10:3574.1-3574.11(2019)
PubMed=31903165; DOI=10.18632/oncotarget.27361; PMCID=PMC6925031
Zheng A., Chevalier N., Calderoni M., Dubuis G., Dormond O., Ziros P.G., Sykiotis G.P., Widmann C.
CRISPR/Cas9 genome-wide screening identifies KEAP1 as a sorafenib, lenvatinib, and regorafenib sensitivity gene in hepatocellular carcinoma.
Oncotarget 10:7058-7070(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
PubMed=32899426; DOI=10.3390/cancers12092510; PMCID=PMC7565451
Scherer D., Davila-Lopez M., Goeppert B., Abrahamsson S., Gonzalez Silos R., Nova I., Marcelain K., Roa J.C., Ibberson D., Umu S.U., Rounge T.B., Roessler S., Lorenzo-Bermejo J.
RNA sequencing of hepatobiliary cancer cell lines: data and applications to mutational and transcriptomic profiling.
Cancers (Basel) 12:2510.1-2510.14(2020)
PubMed=33193621; DOI=10.3389/fgene.2020.546106; PMCID=PMC7581915
Kawamoto M., Yamaji T., Saito K., Shirasago Y., Satomura K., Endo T., Fukasawa M., Hanada K., Osada N.
Identification of characteristic genomic markers in human hepatoma Huh-7 and Huh7.5.1-8 cell lines.
Front. Genet. 11:546106.1-546106.10(2020)"