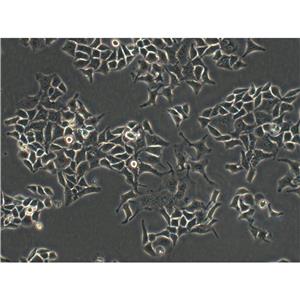
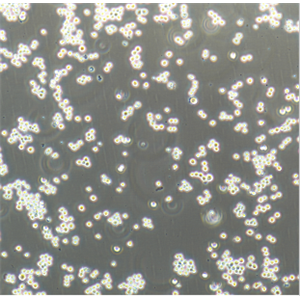
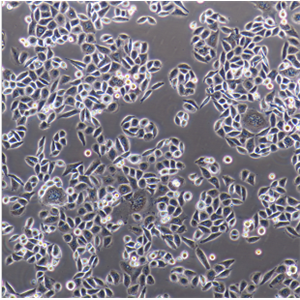
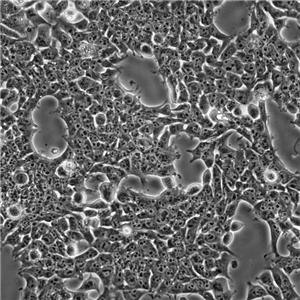
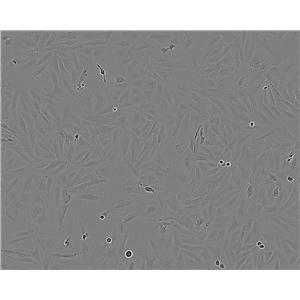
"Hep G2人肝癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
KU-812F Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:E304细胞、LAN-6细胞、BpRc1细胞
PCI-4M Cells;背景说明:喉鳞癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:hTERT RPE1细胞、52PE细胞、WM451Lu细胞
Hs746-T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:DU 4475细胞、NCI-H2126细胞、KY-270细胞
Hep G2人肝癌细胞代次低|培养基|送STR图谱
背景信息:是一种人肝癌细胞系,是来自15岁男性白人的组织,该患者患有高度分化的肝细胞癌;Hep G2细胞形态为上皮细胞样,模式染色体数为55;Hep G2细胞在免疫抑制小鼠中不致瘤。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
绝大部分细胞消化只要用胰酶润洗一遍即可:吸去胰酶后,残留的那些无法计算体积的附着在细胞表面的微量胰酶在37℃一般不到2min足够消化细胞(绝大部分1min不到)。对于这些细胞原则上不要用胰酶孵育细胞,连续这样传代,对细胞伤害很大。简单的程序是PBS润洗吸去,胰酶润洗吸去,然后37℃消化。什么算是消化好了呢?不需要把细胞全部消化成间隔分布很离散的单个圆形才算消化好了,一般你肉眼观察贴壁细胞层,只要能移动了,多半呈沙状移动,其实已经是可以了。一般能移动了,说明细胞与培养基质材料的附着已经消失了,细胞之间的附着也已经消失了,细胞已经独立分布了(虽然没有呈现很广的离散分布)。这个时候应该停止消化,不要等到看到镜下所有细胞都分离得非常好,间隙很大,才停止。细胞系在贴壁的过程中仍然会聚集,这个是贴壁培养的细胞,尤其是肿瘤细胞的一个特性,你可以尝试,准备100%的单个细胞悬液,贴壁后观察细胞,仍然是几个几个细胞聚集在一起。一些悬浮培养细胞也是如此,容易聚集,不要过几个小时就拿出来吹打成单细胞悬液。细胞只要能从基质上脱离下来,即使是成片的(比如Calu-3细胞),吹打不超过20次(一般10次即可),成小规模聚集(10个细胞左右)是正常的,不要再去延长消化时间,等待单细胞悬液出现。比较难消化的细胞:润洗方法5min还不能消化,以结肠癌细胞为例,比如:HCT15、LS411和KM12细胞,胰酶消化,一般10 cm培养皿,一次加入300ul-500ul就足够了。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。一些正常细胞也会有难消化的时候,比如tsDC细胞,用胰酶孵育,3min左右即可看到成片沙状移动。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
Hep G2人肝癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
H1417 Cells;背景说明:小细胞肺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jiyoye细胞、GM03671C细胞、GM2131细胞
RCC-10 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKCO-1细胞、OCI-Ly 18细胞、NCIH1355细胞
TE-1 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OC316细胞、Sf-21细胞、HCC-1395细胞
SW 48 Cells;背景说明:1971-1975年间,A. Leibovitz从结肠腺癌中分离建立十个细胞系,SW48为为其中之一。该细胞合成癌胚抗原,能在裸鼠中成瘤。;传代方法:1:3传代,2-3天传一代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCe-8693细胞、MFHL-2细胞、Detroit-562细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞复苏后贴壁细胞较少的问题分析:总结1:复苏过程没有问题,是否是从拿出直接放入温水,还有培养箱,二氧化碳浓度,培养基、PH值等环节。要么加GAO浓度FBS 15-20%,看看能否帮助贴壁,当然也需要考虑血清问题,还有确信拿来的细胞没问题。总结2:首先应该怀疑冻存,实际上复苏出问题的可能非常小,因为操作简单,而且死板。1、你冻存的时候是不是消化的时间过长,这是一般人所注意不到的,即使书上也不讲这个问题,太长的消化时间会让细胞复苏时失去贴壁能力,表现为先贴后死,原因是在你复苏的时候细胞已进入凋亡程序,不可逆转的死亡。2、你的冻存HAO不HAO,是什么,甘油还是DMSO,质量非常重要,否则也会死亡。3、你的冻存的量加的是不是太多,AC推荐是不超过7%,大于5%,太多也不HAO。4、你在冻存的时候是不是把DMSO混均匀,这个有一些影响,但不算太大。5、你的冻存是否按部就班,就是所温度梯度是不是把握严格,很多人容易忘却这个事情,因为这个东西流程长。6、如果你细胞污染,你是否能很快看到,我比我的导师能早一天看到污染。从这个角度讲建议去除离心这步。7、你的细胞在冻存前是否过密。还有,不赞成孵箱污染这个概念的,所有在一个孵箱里的细胞都污染一个细菌的话,这个细菌是源于孵箱的,但这不代表孵箱污染,因为孵箱无论你如何处理都有大量的细菌,问题在操作。每次污染的原因都要尽可能的找,以后就不犯同样的问题,这个很重要,不能靠猜,否则你就有可能细胞养绝Zui后换课题,这个见得太多了,别不当会事。
HSC-1 Cells;背景说明:皮肤鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:293 H细胞、HOP 62细胞、WI-38细胞
FT-293 Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:B104 [Rat neuroblastoma]细胞、ARH-77细胞、NPA87-1细胞
bEnd3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OCI-LY-7细胞、T 98 G细胞、HEL9217细胞
CORL279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MILE SVEN1细胞、SR细胞、U87 MG细胞
NCIH1734 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SUDHL-1细胞、NCI-H1395细胞、SW-403细胞
OCI Ly3 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Tokyo Medical and Dental university 8细胞、MDAMB436细胞、CESS细胞
Malme-3M Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:NCI-H920细胞、HEK-EBNA细胞、Hs 343.T细胞
EFO27 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH2171细胞、H1417细胞、Balb/c 3T3细胞
NCI-H2198 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ca761细胞、OVCAR5细胞、DSL6A/C1细胞
CCC-HIE-2 Cells;背景说明:胚胎;肠粘膜;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK293EBNA细胞、293 H细胞、Gejiu Lung Carcinoma-82细胞
U-87 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LLC-WRC 256细胞、TE-12细胞、McArdle RH-7777细胞
U-138 MG Cells;背景说明:星形细胞瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH196细胞、A549/DDP细胞、VMRCRCZ细胞
HGBEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BEAS-2B细胞、MGH-U1 (EJ)细胞、LNCaP C4-2B细胞
BRL3A Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:WEHI-164细胞、NTera-2D1细胞、COLO-205细胞
Panc02-H0 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:PANC-08-13细胞、Y-1细胞、VA-ES-BJ细胞
4T1-LUC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEL-92.1.7细胞、BT-325细胞、H650细胞
OCILY3 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Madison细胞、mouse Inner Medullary Collecting Duct-3细胞、HEK AD293细胞
Abcam A-549 TXNRD1 KO Cells(提供STR鉴定图谱)
AG03511 Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSH870 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RST130 Cells(提供STR鉴定图谱)
BEP2D H1ATN Cells(提供STR鉴定图谱)
CHO-tsH1-C11B6 Cells(提供STR鉴定图谱)
DA02919 Cells(提供STR鉴定图谱)
EndoC-betaH2 Cells(提供STR鉴定图谱)
GM06013 Cells(提供STR鉴定图谱)
JROECL 19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BI-Mel细胞、KRC Y细胞、HBSMC细胞
Hep G2人肝癌细胞代次低|培养基|送STR图谱
OVCAR420 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SiHa细胞、MDA MB 134VI细胞、SF767细胞
NK-92 MI Cells;背景说明:NK细胞;淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:H2291细胞、GP293细胞、BE(2)M17细胞
B16/F0 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3/ag细胞、SW-839细胞、ATN1细胞
CCRF-CEM C1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HIMEC细胞、FaDu细胞、HCT-GEO细胞
GH3 Cells;背景说明:GH3细胞系是由TashjianAH等在1965年7月从一只7月龄的雌性Wistar-Furth大鼠的垂体肿瘤中分离建立的。GH3细胞系不是直接来源于GH1细胞系的克隆,而是从原代培养的GH1细胞在大鼠身上传代两次形成的肿瘤中建立的。上皮样的GH3细胞比GH1分泌更高水平的生长激素,也可产生催乳素。对调控GH3细胞分泌蛋白类激素的研究表明,化可的松可以刺激生长激素的分泌、抑制催乳素的产生。;传代方法:1:2传代;生长特性:疏松贴壁,有漂浮的细胞簇;形态特性:上皮样;相关产品有:UPCI-SCC090细胞、HCT15细胞、293T细胞
ABLS-1 Cells(提供STR鉴定图谱)
HEp-2 Cells;背景说明:最初认为这个细胞源自喉上皮癌,但随后通过同功酶分析、HeLa标记染色体和DNA指纹分析发现,起源细胞已被HeLa污染。 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Jurkat-E6细胞、HCC38细胞、beta TC6细胞
H-1734 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:JM-1细胞、3T3NIH细胞、HBL-100细胞
U-343-MG Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:OUMS-27细胞、CMT 93细胞、H1568细胞
5-8F Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Hs852细胞、TE1细胞、NCI-H1417细胞
SKOV-3 Cells;背景说明:SK-OV-3由G.Trempe和L.J.Old在1973年从卵巢肿瘤病人的腹水分离得到。 此细胞对肿瘤坏死因子和几种细胞毒性药物包括白喉毒素、顺铂和阿霉素均耐受。 在裸鼠中致瘤,且形成与卵巢原位癌一致的中度分化的腺癌。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:MOLT 3细胞、MADB-106细胞、G-292细胞
H-28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCIH2126细胞、NCI-H2228细胞、UACC-812细胞
JEG-3 Cells;背景说明:这是一株超三倍体人类细胞株;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-295R细胞、GM02131A细胞、Shadyside Hospital Pittsburgh-77细胞
SKNEP Cells;背景说明:超微结构有少许微绒毛,连接复合物,形态完整的高尔基体,内质网多为光滑型,脂滴,没有病毒棵粒。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:上皮细胞;相关产品有:U-138MG细胞、U14细胞、BIC1细胞
GS-109-V-20 Cells(提供STR鉴定图谱)
HAP1 PPARD (-) 2 Cells(提供STR鉴定图谱)
U-343MG Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NCI-SNU-601细胞、SW480E细胞、DB细胞
GDM-1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:MCF7-GFP细胞、B16 melanoma F10细胞、UWB1289细胞
OVCAR.8 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-446细胞、HCM细胞、IPLB-Sf21AE细胞
C-Lu65 Cells;背景说明:详见相关文献介绍;传代方法:1:10传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:MOLP-2细胞、RCC10细胞、NCIH2009细胞
Panc 10.05 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:U373-MG细胞、CMT-93细胞、SNUC2A细胞
Mouse Bladder Tumor line-2 Cells;背景说明:膀胱移行细胞癌;C3H/He;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NPA87-1细胞、Porcine Kidney-15细胞、IPLB-SF-21细胞
MM1.S Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:混合生长;形态特性:淋巴母细胞样;相关产品有:TKB1细胞、LM8细胞、V-79细胞
A704 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:4传代,每周换液2-3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:VP229细胞、HFLS细胞、Hs606细胞
hTERT SMC PM151T Cells(提供STR鉴定图谱)
KTCTL-1M Cells(提供STR鉴定图谱)
MRC-5-50 Cells(提供STR鉴定图谱)
NZW-S1-M Cells(提供STR鉴定图谱)
RKID001 Cells(提供STR鉴定图谱)
Ubigene HEK293 EGFR KO Cells(提供STR鉴定图谱)
WG0680 Cells(提供STR鉴定图谱)
HC11-C24 Cells(提供STR鉴定图谱)
SH-SY5Y Cells;背景说明:据报道,该细胞的密度可高达1×106cells/cm2,具有中等水平的多巴胺β羟化酶的活性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HMEC1细胞、COR L279细胞、ES-2细胞
CCD19-Lu Cells;背景说明:肺成纤维细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:UCD-MLA-144细胞、NG 108-15细胞、AAV-293细胞
6-10B Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-ChA-1细胞、OVCA8细胞、SU-86-86细胞
H-748 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长 ;形态特性:详见产品说明书;相关产品有:Mel624细胞、MARC 145细胞、SK-RC 39细胞
Colon38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H125细胞、RC13细胞、JF305细胞
Colon38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H125细胞、RC13细胞、JF305细胞
SCL2 Cells;背景说明:皮肤鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-415细胞、4175细胞、TFK-1细胞
MN9D Cells;背景说明:多巴胺能神经元 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U251n细胞、Vero from pool #76细胞、HAPI细胞
KE37 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:H2107细胞、J111细胞、M14细胞
Mouse Podocyte Clone-5 Cells;背景说明:肾足细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DSL-6A/C1细胞、HCEC-12细胞、U-251细胞
CORL279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MILE SVEN1细胞、SR细胞、U87 MG细胞
COS7 Cells;背景说明:此细胞株源自CV-1细胞株,经转染起始点缺失的SV40病毒突变体得到;编码表达野生型T抗原,所以该细胞适合作为需要SV40T抗原表达的载体的转染宿主。该细胞表达T抗原,允许SV40病毒的溶解性生长,支持40℃时温度敏感性A209病毒的复制,支持起始区域缺陷的SV40突变体的复制。因含有SV40病毒的DNA序列,该细胞需要在2级生物安全柜中操作。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:BALB/3T3 cl. A31细胞、HR1K细胞、Vx2细胞
NOZAWA Cells;背景说明:患者有癌性腹膜炎。细胞为中等分化的管状胆囊癌。会分泌AFP和CEA。倍增时间48小时,板植率14-19%。细胞可在裸鼠中成瘤,形态与原发肿瘤相似。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:ME180细胞、NCI-SNU-878细胞、HepaRG细胞
Hs 683.T Cells;背景说明:该细胞源自76岁白人男性的左颞叶侧胶质瘤组织,有微绒毛,无桥粒。 ;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TPC1细胞、RPTC细胞、HS5细胞
4T1.2 Cells;背景说明:乳腺癌;雌性;BALB/cfC3H;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SHG-44细胞、143 B细胞、Colon26细胞
SW-OH1 Cells(提供STR鉴定图谱)
686LN-M4e Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HSC-2细胞、Dx5细胞、786-O RCC细胞
QG-56 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SY5Y细胞、Tu-686细胞、SB细胞
VERO76 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AN3CA细胞、SCC090细胞、CAKI.1细胞
HEK/293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:VMM5A细胞、Human Corneal Epithelial cells-Transformed细胞、L-M[TK-]细胞
LM(TK-) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC0015细胞、373 MG细胞、B/C3T3细胞
Sci-1 Cells;背景说明:胚胎;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Anip-973细胞、EC-9706细胞、ECC 10细胞
Hep G2人肝癌细胞代次低|培养基|送STR图谱
NPA87-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SUM 52PE细胞、Y3细胞、HT 1376细胞
HH [Human lymphoma] Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NCIH1184细胞、H-1882细胞、C2A细胞
BrCL18 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MUTZ3细胞、SKMEL-2细胞、VK-2/E6E7细胞
H1954 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Eca109细胞、KNS-42细胞、HO-8910PM细胞
JIMT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HECV细胞、A1847细胞、MDA-MB-157细胞
Hs688(A)T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:WM266-4细胞、NIH:OVCAR-10细胞、HT 1197细胞
J774A1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Centre Antoine Lacassagne-148细胞、P388细胞、MSB-1细胞
alphaTC1 Clone 6 Cells;背景说明:胰岛素瘤;a细胞;C57BL/6xDBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WM115mel细胞、WiDr细胞、SK-N-BE(1)n细胞
BayGenomics ES cell line RRS098 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA357 Cells(提供STR鉴定图谱)
iBMK D3.Hras-1RED-10 Cells(提供STR鉴定图谱)
PCRP-ORC1-1C4 Cells(提供STR鉴定图谱)
irPSCc2 Cells(提供STR鉴定图谱)
HPSI0516i-yibc_4 Cells(提供STR鉴定图谱)
" "Patent=US4393133
Knowles B.B., Aden D.P.
Human hepatoma derived cell line, process for preparation thereof, and uses therefor.
Patent number US4393133, 12-Jul-1983
PubMed=2439335; DOI=10.1111/j.1432-1033.1987.tb11497.x
Vincent C., Marceau M., Blangarin P., Bouic P., Madjar J.-J., Revillard J.-P.
Purification of alpha 1-microglobulin produced by human hepatoma cell lines. Biochemical characterization and comparison with alpha 1-microglobulin synthesized by human hepatocytes.
Eur. J. Biochem. 165:699-704(1987)
PubMed=8224613; DOI=10.1096/fasebj.7.14.8224613
Puisieux A., Galvin K., Troalen F., Bressac B., Marcais C., Galun E., Ponchel F., Yakicier C., Ji J.-W., Ozturk M.
Retinoblastoma and p53 tumor suppressor genes in human hepatoma cell lines.
FASEB J. 7:1407-1413(1993)
PubMed=8384076; DOI=10.1016/0165-4608(93)90227-D
Chen H.-L., Chiu T.-S., Chen P.-J., Chen D.-S.
Cytogenetic studies on human liver cancer cell lines.
Cancer Genet. Cytogenet. 65:161-166(1993)
PubMed=8389256; DOI=10.1093/carcin/14.5.987
Hsu I.-C., Tokiwa T., Bennett W.P., Metcalf R.A., Welsh J.A., Sun T.-T., Harris C.C.
p53 gene mutation and integrated hepatitis B viral DNA sequences in human liver cancer cell lines.
Carcinogenesis 14:987-992(1993)
PubMed=7972006; DOI=10.1073/pnas.91.23.11045; PMCID=PMC45163
Okamoto A., Demetrick D.J., Spillare E.A., Hagiwara K., Hussain S.P., Bennett W.P., Forrester K., Gerwin B.I., Serrano M., Beach D.H., Harris C.C.
Mutations and altered expression of p16INK4 in human cancer.
Proc. Natl. Acad. Sci. U.S.A. 91:11045-11049(1994)
PubMed=8050184; DOI=10.1111/j.1365-2249.1994.tb06089.x; PMCID=PMC1534706
Wadee A.A., Paterson A., Coplan K.A., Reddy S.G.
HLA expression in hepatocellular carcinoma cell lines.
Clin. Exp. Immunol. 97:328-333(1994)
PubMed=8835345; DOI=10.1002/(SICI)1096-9071(199602)48:2<133::AID-JMV3>3.0.CO;2-A
Tsuboi S., Nagamori S., Miyazaki M., Mihara K., Fukaya K.-i., Teruya K., Kosaka T., Tsuji T., Namba M.
Persistence of hepatitis C virus RNA in established human hepatocellular carcinoma cell lines.
J. Med. Virol. 48:133-140(1996)
DOI=10.11418/jtca1981.16.3_173
Mihara K., Miyazaki M., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ohashi R., Namba M.
The p53 gene status and other cellular characteristics of human cell lines maintained in our laboratory.
Tissue Cult. Res. Commun. 16:173-178(1997)
PubMed=9178645; DOI=10.1006/cimm.1997.1108
Nakao M., Sata M., Saitsu H., Yutani S., Kawamoto M., Kojiro M., Itoh K.
CD4+ hepatic cancer-specific cytotoxic T lymphocytes in patients with hepatocellular carcinoma.
Cell. Immunol. 177:176-181(1997)
PubMed=9359923; DOI=10.18926/AMO/30789
Mihara K., Miyazaki M., Kondo T., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ishioka C., Namba M.
Yeast functional assay of the p53 gene status in human cell lines maintained in our laboratory.
Acta Med. Okayama 51:261-265(1997)
PubMed=11050057; DOI=10.1053/jhep.2000.19349
Wong N., Lai P.B.-S., Pang E., Leung T.W.-T., Lau J.W.-Y., Johnson P.J.
A comprehensive karyotypic study on human hepatocellular carcinoma by spectral karyotyping.
Hepatology 32:1060-1068(2000)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=11981770; DOI=10.1053/jhep.2002.32668
Clemens D.L., Forman A., Jerrells T.R., Sorrell M.F., Tuma D.J.
Relationship between acetaldehyde levels and cell survival in ethanol-metabolizing hepatoma cells.
Hepatology 35:1196-1204(2002)
PubMed=12029633; DOI=10.1053/jhep.2002.33683
Yasui K., Arii S., Zhao C., Imoto I., Ueda M., Nagai H., Emi M., Inazawa J.
TFDP1, CUL4A, and CDC16 identified as targets for amplification at 13q34 in hepatocellular carcinomas.
Hepatology 35:1476-1484(2002)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
DOI=10.1385/CP:1:3-4:313
Pang R.T.-K., Poon T.C.-W., Wong N., Lai P.B.-S., Wong N.L.-Y., Chan C.M.-L., Yu J.W.S., Chan A.T.-C., Sung J.J.-Y.
Comparison of protein expression patterns between hepatocellular carcinoma cell lines and a hepatoblastoma cell line.
Clin. Proteomics 1:313-331(2004)
PubMed=14980111
Zhai B.-J., Wu F., Shao Z.-Y., Hu K., Zhao C.-L., Wang Z.-B.
Establishment of human hepatocellular carcinoma multidrug-resistance cell line (HepG2/Adm) and study apoptosis induced by low-frequency pulse ultrasound exposure.
Zhonghua Gan Zang Bing Za Zhi 12:95-98(2004)
PubMed=15767549; DOI=10.1158/1535-7163.MCT-04-0234
Nakatsu N., Yoshida Y., Yamazaki K., Nakamura T., Dan S., Fukui Y., Yamori T.
Chemosensitivity profile of cancer cell lines and identification of genes determining chemosensitivity by an integrated bioinformatical approach using cDNA arrays.
Mol. Cancer Ther. 4:399-412(2005)
PubMed=16181800; DOI=10.1016/j.biocel.2005.07.010
Donohue T.M., Osna N.A., Clemens D.L.
Recombinant Hep G2 cells that express alcohol dehydrogenase and cytochrome P450 2E1 as a model of ethanol-elicited cytotoxicity.
Int. J. Biochem. Cell Biol. 38:92-101(2006)
PubMed=16935386; DOI=10.1016/j.jhep.2006.05.019
Sun D.-X., Nassal M.
Stable HepG2- and Huh7-based human hepatoma cell lines for efficient regulated expression of infectious hepatitis B virus.
J. Hepatol. 45:636-645(2006)
PubMed=17254797; DOI=10.1016/j.biologicals.2006.10.001
Azari S., Ahmadi N., Jeddi-Tehrani M., Shokri F.
Profiling and authentication of human cell lines using short tandem repeat (STR) loci: report from the National Cell Bank of Iran.
Biologicals 35:195-202(2007)
PubMed=19215227; DOI=10.1111/j.1349-7006.2009.01082.x; PMCID=PMC11158180
Kuwahara Y., Li L., Baba T., Nakagawa H., Shimura T., Yamamoto Y., Ohkubo Y., Fukumoto M.
Clinically relevant radioresistant cells efficiently repair DNA double-strand breaks induced by X-rays.
Cancer Sci. 100:747-752(2009)
PubMed=19751877; DOI=10.1016/j.humpath.2009.07.003
Lopez-Terrada D.H., Cheung S.-W., Finegold M.J., Knowles B.B.
Hep G2 is a hepatoblastoma-derived cell line.
Hum. Pathol. 40:1512-1515(2009)
PubMed=20069059; DOI=10.1155/2010/437143; PMCID=PMC2801507
Srisomsap C., Sawangareetrakul P., Subhasitanont P., Chokchaichamnankit D., Chiablaem K., Bhudhisawasdi V., Wongkham S., Svasti J.
Proteomic studies of cholangiocarcinoma and hepatocellular carcinoma cell secretomes.
J. Biomed. Biotechnol. 2010:437143.1-437143.18(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20228232; DOI=10.1124/dmd.109.031831; PMCID=PMC2879958
Hart S.N., Li Y., Nakamoto K., Subileau E.-A., Steen D., Zhong X.-B.
A comparison of whole genome gene expression profiles of HepaRG cells and HepG2 cells to primary human hepatocytes and human liver tissues.
Drug Metab. Dispos. 38:988-994(2010)
PubMed=20937217; DOI=10.1170/149
Di Masi A., Viganotti M., Antoccia A., Magrelli A., Salvatore M., Azzalin G., Tosto F., Lorenzetti S., Maranghi F., Mantovani A., Macino G., Tanzarella C., Taruscio D.
Characterization of HuH6, Hep3B, HepG2 and HLE liver cancer cell lines by WNT/beta-catenin pathway, microRNA expression and protein expression profile.
Cell. Mol. Biol. 56:OL1299-OL1317(2010)
PubMed=21269460; DOI=10.1186/1752-0509-5-17; PMCID=PMC3039570
Burkard T.R., Planyavsky M., Kaupe I., Breitwieser F.P., Burckstummer T., Bennett K.L., Superti-Furga G., Colinge J.
Initial characterization of the human central proteome.
BMC Syst. Biol. 5:17.1-17.13(2011)
PubMed=22278370; DOI=10.1074/mcp.M111.014050; PMCID=PMC3316730
Geiger T., Wehner A., Schaab C., Cox J., Mann M.
Comparative proteomic analysis of eleven common cell lines reveals ubiquitous but varying expression of most proteins.
Mol. Cell. Proteomics 11:M111.014050-M111.014050(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23325432; DOI=10.1101/gr.147942.112; PMCID=PMC3589544
Varley K.E., Gertz J., Bowling K.M., Parker S.L., Reddy T.E., Pauli-Behn F., Cross M.K., Williams B.A., Stamatoyannopoulos J.A., Crawford G.E., Absher D.M., Wold B.J., Myers R.M.
Dynamic DNA methylation across diverse human cell lines and tissues.
Genome Res. 23:555-567(2013)
PubMed=23505090; DOI=10.1002/hep.26402
Wang K., Lim H.Y., Shi S., Lee J., Deng S.-B., Xie T., Zhu Z., Wang Y.-L., Pocalyko D., Yang W.J., Rejto P.A., Mao M., Park C.-K., Xu J.-C.
Genomic landscape of copy number aberrations enables the identification of oncogenic drivers in hepatocellular carcinoma.
Hepatology 58:706-717(2013)
PubMed=23887712; DOI=10.1038/ncomms3218; PMCID=PMC3731665
Nault J.-C., Mallet M., Pilati C., Calderaro J., Bioulac-Sage P., Laurent C., Laurent A., Cherqui D., Balabaud C., Zucman-Rossi J.
High frequency of telomerase reverse-transcriptase promoter somatic mutations in hepatocellular carcinoma and preneoplastic lesions.
Nat. Commun. 4:2218.1-2218.7(2013)
PubMed=24116068; DOI=10.1371/journal.pone.0075692; PMCID=PMC3792989
Weiskirchen R., Weimer J., Meurer S.K., Kron A., Seipel B., Vater I., Arnold N., Siebert R., Xu L.-M., Friedman S.L., Bergmann C.
Genetic characteristics of the human hepatic stellate cell line LX-2.
PLoS ONE 8:E75692-E75692(2013)
PubMed=24618588; DOI=10.1371/journal.pone.0091433; PMCID=PMC3950186
Chernobrovkin A.L., Zubarev R.A.
Detection of viral proteins in human cells lines by xeno-proteomics: elimination of the last valid excuse for not testing every cellular proteome dataset for viral proteins.
PLoS ONE 9:E91433-E91433(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25574106; DOI=10.3748/wjg.v21.i1.311; PMCID=PMC4284350
Cevik D., Yildiz G., Ozturk M.
Common telomerase reverse transcriptase promoter mutations in hepatocellular carcinomas from different geographical locations.
World J. Gastroenterol. 21:311-317(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26160117; DOI=10.1093/toxsci/kfv136; PMCID=PMC4583060
Sison-Young R.L.C., Mitsa D., Jenkins R.E., Mottram D., Alexandre E., Richert L., Aerts H., Weaver R.J., Jones R.P., Johann E., Hewitt P.G., Ingelman-Sundberg M., Goldring C.E.P., Kitteringham N.R., Park B.K.
Comparative proteomic characterization of 4 human liver-derived single cell culture models reveals significant variation in the capacity for drug disposition, bioactivation, and detoxication.
Toxicol. Sci. 147:412-424(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26825538; DOI=10.1016/j.jprot.2016.01.016
Wisniewski J.R., Vildhede A., Noren A., Artursson P.
In-depth quantitative analysis and comparison of the human hepatocyte and hepatoma cell line HepG2 proteomes.
J. Proteomics 136:234-247(2016)
PubMed=27027780; DOI=10.1007/s10565-016-9316-2
Wu Y., Geng X.-C., Wang J.-F., Miao Y.-F., Lu Y.-L., Li B.
The HepaRG cell line, a superior in vitro model to L-02, HepG2 and hiHeps cell lines for assessing drug-induced liver injury.
Cell Biol. Toxicol. 32:37-59(2016)
PubMed=27329724; DOI=10.18632/oncotarget.10161; PMCID=PMC5216950
Watari K., Nishitani A., Shibata T., Noda M., Kawahara A., Akiba J., Murakami Y., Yano H., Kuwano M., Ono M.
Phosphorylation of mTOR Ser2481 is a key target limiting the efficacy of rapalogs for treating hepatocellular carcinoma.
Oncotarget 7:47403-47417(2016)
PubMed=27470094; DOI=10.1016/j.chroma.2016.07.042
Liu Z.-Y., Wang F.-J., Chen J., Zhou Y., Zou H.-F.
Modulating the selectivity of affinity absorbents to multi-phosphopeptides by a competitive substitution strategy.
J. Chromatogr. A 1461:35-41(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29610054; DOI=10.1016/j.dmpk.2018.03.003; PMCID=PMC6309175
Shi J., Wang X.-W., Lyu L.-Y., Jiang H., Zhu H.-J.
Comparison of protein expression between human livers and the hepatic cell lines HepG2, Hep3B, and Huh7 using SWATH and MRM-HR proteomics: Focusing on drug-metabolizing enzymes.
Drug Metab. Pharmacokinet. 33:133-140(2018)
PubMed=29660373; DOI=10.1016/j.bbagen.2018.04.012
Touat-Hamici Z., Bulteau A.-L., Bianga J., Jean-Jacques H., Szpunar J., Lobinski R., Chavatte L.
Selenium-regulated hierarchy of human selenoproteome in cancerous and immortalized cells lines.
Biochim. Biophys. Acta 1862:2493-2505(2018)
PubMed=30629668; DOI=10.1371/journal.pone.0210404; PMCID=PMC6328144
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30864654; DOI=10.1093/nar/gkz169; PMCID=PMC6486628
Zhou B., Ho S.S., Greer S.U., Spies N., Bell J.M., Zhang X.-L., Zhu X.-W., Arthur J.G., Byeon S., Pattni R., Saha I., Huang Y.-L., Song G., Perrin D., Wong W.H., Ji H.P., Abyzov A., Urban A.E.
Haplotype-resolved and integrated genome analysis of the cancer cell line HepG2.
Nucleic Acids Res. 47:3846-3861(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31063779; DOI=10.1053/j.gastro.2019.05.001
Caruso S., Calatayud A.-L., Pilet J., La Bella T., Rekik S., Imbeaud S., Letouze E., Meunier L., Bayard Q., Rohr-Udilova N., Peneau C., Grasl-Kraupp B., de Koning L., Ouine B., Bioulac-Sage P., Couchy G., Calderaro J., Nault J.-C., Zucman-Rossi J., Rebouissou S.
Analysis of liver cancer cell lines identifies agents with likely efficacy against hepatocellular carcinoma and markers of response.
Gastroenterology 157:760-776(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31378681; DOI=10.1016/j.ccell.2019.07.001; PMCID=PMC7505724
Qiu Z.-X., Li H., Zhang Z.-T., Zhu Z.-F., He S., Wang X.-J., Wang P.-C., Qin J.-J., Zhuang L.-P., Wang W., Xie F.-B., Gu Y., Zou K.-K., Li C., Li C., Wang C.-H., Cen J., Chen X.-T., Shu Y.-J., Zhang Z., Sun L.-L., Min L.-H., Fu Y., Huang X.-W., Lv H., Zhou H., Ji Y., Zhang Z.-G., Meng Z.-Q., Shi X.-L., Zhang H.-B., Li Y.-X., Hui L.-J.
A pharmacogenomic landscape in human liver cancers.
Cancer Cell 36:179-193.e11(2019)
PubMed=31395879; DOI=10.1038/s41467-019-11415-2; PMCID=PMC6687785
Yu K., Chen B., Aran D., Charalel J., Yau C., Wolf D.M., van 't Veer L.J., Butte A.J., Goldstein T., Sirota M.
Comprehensive transcriptomic analysis of cell lines as models of primary tumors across 22 tumor types.
Nat. Commun. 10:3574.1-3574.11(2019)
PubMed=31561354; DOI=10.3233/CH-199226; PMCID=PMC6918903
Schulz C., Kammerer S., Kupper J.-H.
NADPH-cytochrome P450 reductase expression and enzymatic activity in primary-like human hepatocytes and HepG2 cells for in vitro biotransformation studies.
Clin. Hemorheol. Microcirc. 73:249-260(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)"