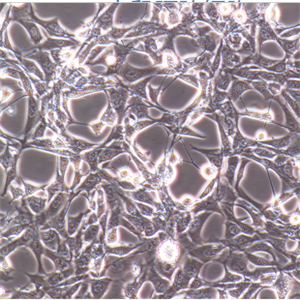
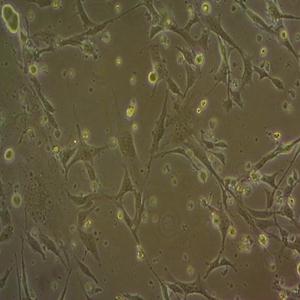
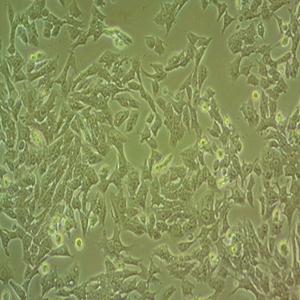
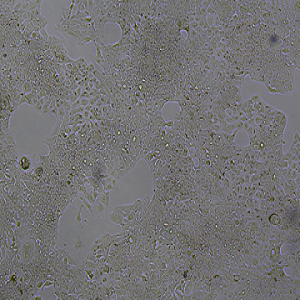
"HCC827人非小细胞肺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
换液周期:每周2-3次
SK-SH-SY5Y Cells;背景说明:据报道,该细胞的密度可高达1×106cells/cm2,具有中等水平的多巴胺β羟化酶的活性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:QGY-7701细胞、SKO007细胞、RS4;11细胞
H7721 Cells;背景说明:用Northernblot方法,未能检测到细胞中1.3kbLFIRE-1/HFREP-1mRNA的表达。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCI-Ly 18细胞、NCIH2073细胞、PY8119细胞
L-M(TK-) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GB-1细胞、BALB/3T3细胞、KMM1细胞
HCC827人非小细胞肺癌细胞代次低|培养基|送STR图谱
背景信息:这株细胞建于1994年三月。这株肺腺癌在EGFR酪激酶区域有一个获得性突变(E746-A750缺失)。患者在25岁到26岁时每个月抽1包烟。在诊断前12年不再抽烟。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞(adherent cells):该类细胞的生长必须有可以贴附的支持物表面,细胞依靠自身分泌的或培养基中提供的贴附因子才能在该表面上生长,繁殖;当细胞在该表面生长后,一般形成两种形态,即成纤维样细胞性或上皮样细胞;其生长过程分为游离期、贴壁期、潜伏期、对数期、平台期和衰退期。悬浮细胞(suspension cell):不贴附于生长物,细胞呈圆形,呈单个细胞或者细小细胞团,悬浮细胞生长空间大,传代方便,能够大量增殖。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
HCC827人非小细胞肺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
M 1 Cells;背景说明:髓系白血病;SL;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:32D.cl3细胞、NCIH187细胞、639 V细胞
Hela-Ap-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LC-2/ad细胞、HSKMC细胞、MKN1细胞
SK-ES Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:5传代;每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:LK 2细胞、Emory University-3细胞、U-138 MG细胞
CC-LP-I Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MES-SA Dx5细胞、NCIH2052细胞、OVHM细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞培养无菌操作步骤和注意事项:1.打开无菌工作台及净化室的紫外灯,消毒半小时以上。2.进入净化室前先关闭紫外灯,打开超净台风机,等待30min以上,以排尽臭氧。3.穿HAO隔离衣,戴HAO口罩,帽子。4.风淋2分钟。5.0.1%水泡手,擦干。6.点燃酒精灯;超净台内应避免放入过多的物品;使用的吸管,滴管,试管,培养瓶等均事先灭菌。7.打开各类瓶盖前先过火,以固定灰尘;打开的瓶口、试管口过火焰,镊子使用前应经火焰烧灼。8.水平式风机的超净台,应使瓶口斜置,应尽量避免瓶口敞开直立。9.同一根吸管或滴管不应连续用于几个不同的细胞系;吸取培养基的吸管应离开培养瓶或试管口0.5cm,避免伸入培养瓶口或试管口;以防止细胞系的互相混杂污染。10.漏在培养瓶上或台上的体,立即用酒精棉球擦净。11.操作完毕后恢复工作台面。
PSN1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SVEC4-10细胞、TOV21G细胞、DiFi细胞
IGR-OV1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1734细胞、EMT6细胞、MFD-1细胞
SNU449 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代;每周2-3次;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:BNL 1MEA.7R.1细胞、NCI-H1975细胞、HUC-1细胞
SK-N-F1 Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs 274.T细胞、IM-95细胞、SW 1271细胞
3T3 J2 Cells;背景说明:胚胎;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Henrietta Lacks cells细胞、RPMI 8402细胞、G-401细胞
NCI-H740 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LA-N-6细胞、AM细胞、Panc_08_13细胞
Instituto Biologico-Rim Suino-2 Cells;背景说明:肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SSP-25细胞、H6c7细胞、HANK细胞
HGC27 Cells;背景说明:未分化胃癌,能分泌粘液素。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SK BR 03细胞、MDA-134细胞、Hep G2细胞
IMR32 Cells;背景说明:该细胞是1967年4月由NicholsWW,LeeJ和DwightS建立,来源于一名13月龄白人男婴腹部肿块,临床诊断为神经母细胞瘤,伴有极少部位的类器官样分化。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:存在两种细胞类型,小的神经母细胞样细胞和大的透明成纤维样细胞;相关产品有:Tu686细胞、TE8细胞、266 Bl细胞
HHFK Cells;背景说明:毛囊;角质 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEMCSS细胞、MDA-MB-134细胞、Michigan Cancer Foundation-10A细胞
LC-2-Ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKRC 39细胞、HLFa细胞、WB-F344细胞
CL MC/9 Cells;背景说明:肥大细胞;C57BL/6 x A/J;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HFL 1细胞、Walker256-TC细胞、NOR-10细胞
Hs840T Cells;背景说明:详见相关文献介绍;传代方法:1:4—1:8传代,每周换液2—3次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:BTI-Tn5B14细胞、NCI H548细胞、HT-55细胞
HFL-I Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Tregs细胞、HT1376细胞、H-295细胞
BERH-2 Cells;背景说明:肝癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MM1-S细胞、HL-7702细胞、Roswell Park Memorial Institute 2650细胞
MIMVEC Cells;背景说明:肠粘膜微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A2780细胞、CCLP1细胞、SW-780细胞
RT-BM 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:B95.8细胞、SK MEL-28细胞、CemT4细胞
A6 Cells(提供STR鉴定图谱)
Abcam Raji ACAA1 KO Cells(提供STR鉴定图谱)
Ba/F3 ETV6-NTRK2-G709C Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRR237 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHD214 Cells(提供STR鉴定图谱)
CG0028 Cells(提供STR鉴定图谱)
DA01515 Cells(提供STR鉴定图谱)
EK [Anguilla] Cells(提供STR鉴定图谱)
GM05725 Cells(提供STR鉴定图谱)
B16/BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ketr 3细胞、Huh-7.5细胞、RWPE2细胞
HCC827人非小细胞肺癌细胞代次低|培养基|送STR图谱
LUDLU 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC9204细胞、CCD841CoTr细胞、NPC-TW-039细胞
DHL10 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:H1793细胞、TB-1 Lu细胞、32D CL3细胞
Hs729T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NIHOVCAR3细胞、EJ细胞、MDA361细胞
Wien-133 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-322细胞、NL20细胞、NCIH2030细胞
A 172 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:A427细胞、BEAS2B细胞、Tissue Culture-1细胞
A9(GM2621)-4 Cells(提供STR鉴定图谱)
RT-BM Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:DHL6细胞、H-740细胞、OKAC1细胞
HGSMC Cells;背景说明:胃平滑肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TE12细胞、RC92A细胞、NCI H82细胞
Cloudman S91 melanoma Cells;背景说明:黑色素瘤;雄性;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO 206细胞、CAL39细胞、NCI-522细胞
HEB Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:KMS26细胞、H-847细胞、EB1细胞
697 Cells;背景说明:B淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NUGC3细胞、CCD 841 CoN细胞、NCI-H841细胞
SHI1 Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RAG细胞、U266 Bl细胞、LIM1215细胞
H-2110 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮细胞;相关产品有:RCC 786-O细胞、Hs-729-T细胞、WC00079细胞
NTERA-2/D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RM-1细胞、H-520细胞、MAVER-1细胞
GM11045 Cells(提供STR鉴定图谱)
HAP1 CLK4 (-) 1 Cells(提供STR鉴定图谱)
MiaPaCa2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HEC151细胞、CA 46细胞、KM12SM细胞
NCIH2073 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:BCECs细胞、L23/P细胞、ECC1细胞
ACT-1 Cells;背景说明:未分化甲状腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-2-Ad细胞、SW1990细胞、H1688细胞
HBL 100 Cells;背景说明:该细胞由E.V.Gaffney及其同事从一位没有乳癌家族史的供者乳汁中建立,培养出来的细胞染色体组型在第7代时就不正常;电镜照片显示有微丝、张力原纤维和桥粒;Southern转移表明有整合型SV40病毒基因,当作正常细胞。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:RKOE6细胞、HCT 8细胞、ZR751细胞
SW 1116 Cells;背景说明:CSAp阴性(CSAp-)。 结肠抗原3,阴性。 角蛋白免疫过氧化物酶染色阳性。 癌基因c-myc, K-ras, H-ras, myb, sis 和fos的表达呈阳性。 未检测到癌基因N-myc和N-ras的表达。 表达肿瘤特异的核基质蛋白CC-4,CC-5和CC-6。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:WM239A细胞、PF382细胞、MCF-12A细胞
SUM 52PE Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OKa-C-1细胞、HT 1376细胞、NCI-H2198细胞
CTV1 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Intestinal Epithelioid Cell line No. 6细胞、NEC细胞、PANC.1细胞
Hs-343-T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:FL83B细胞、RCK-8细胞、HCT FET细胞
HG04198 Cells(提供STR鉴定图谱)
iMPD183.C6 Cells(提供STR鉴定图谱)
LUTC-5 Cells(提供STR鉴定图谱)
ND04066 Cells(提供STR鉴定图谱)
PDSHM147V#13 Cells(提供STR鉴定图谱)
U2OS TBK1 KO Cells(提供STR鉴定图谱)
UMGi013-A Cells(提供STR鉴定图谱)
HCT116-SLC35F2-KO-c3 Cells(提供STR鉴定图谱)
HTR-8/SV neo Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:BDCM细胞、RMa-bm细胞、NCI-H747细胞
EFM-19 Cells;背景说明:乳腺管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKMES细胞、RD-ES细胞、HT-115细胞
X63Ag8-653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCMEC(BL12-H)细胞、HCCLM3细胞、GNM细胞
NCIH82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:HT55细胞、NTera 2细胞、Stanford University-Diffuse Histiocytic Lymphoma-1细胞
SK-UT-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:12传代,2天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:TE-14细胞、3-LL细胞、HPAEpiC细胞
SK-UT-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:12传代,2天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:TE-14细胞、3-LL细胞、HPAEpiC细胞
SW954 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:KNS81细胞、MIA-Pa-Ca-2细胞、HCC15细胞
V79-1 Cells;背景说明:肺;自发永生;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MM96L细胞、Capan1细胞、HEY-A8细胞
PL12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:16HBE140细胞、PA-TU-8988S细胞、HIBEpiC细胞
SW 837 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液1-2次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:NCIH2122细胞、NCM-460细胞、Kasumi-1细胞
SK-RC-20 Cells;背景说明:肾癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CT 26细胞、L 1210细胞、TE 32.T细胞
SK-OV-3 Cells;背景说明:SK-OV-3由G.Trempe和L.J.Old在1973年从卵巢肿瘤病人的腹水分离得到。 此细胞对肿瘤坏死因子和几种细胞毒性药物包括白喉毒素、顺铂和阿霉素均耐受。 在裸鼠中致瘤,且形成与卵巢原位癌一致的中度分化的腺癌。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:WM-239-A细胞、TCMK1细胞、NCIH2405细胞
OVCAR8/ADR Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CTLA4 Ig-24细胞、RBL-2H3细胞、LN229细胞
LS123 Cells;背景说明:详见相关文献介绍;传代方法:1:4—1:8传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HS-68细胞、A-172 MG细胞、HSMC细胞
HuT 78 Cells;背景说明:皮肤;T淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:LS 174T细胞、SKMEL3细胞、ST细胞
SCVIi081-A Cells(提供STR鉴定图谱)
Z-138 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:15传代;每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H838细胞、LNCaP-FGC细胞、D283 Med细胞
Walker-256 Cells;背景说明:乳腺癌;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:AR4-IP细胞、HCC0366细胞、HEL-92_1_7细胞
KATOIII Cells;背景说明:详见相关文献介绍;传代方法:1:2传代。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCI-841细胞、Z310细胞、NIH:OVCAR-4细胞
SW 626 Cells;背景说明:卵巢癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-1/sq细胞、HK-2 [Human kidney]细胞、159 PT细胞
CWR22-R1 Cells;背景说明:22RV1是来自异种移植(在阉割引起前列腺癌衰退又在其父亲的雄性激素信赖型CWR22嫁接后复发的小鼠中连续传代)的人前列腺癌上皮细胞系。此细胞系表达前列腺特异抗原。二羟基睾丸脂酮轻微刺激细胞生长,经westernblot检测溶解产物与抗雄性激素受体抗体起免疫反应。EGF刺激细胞生长,但TGFβ-1不能抑制细胞生长。该细胞在裸鼠中成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Bat lung细胞、MCF-7B细胞、LS-411细胞
Caco-2 BBe Cells;背景说明:详见相关文献介绍;传代方法:1:6—1:10传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:RPMI 6666细胞、Human Lung Microvascular Endothelial Cell line-5a细胞、Colo-678细胞
HCC827人非小细胞肺癌细胞代次低|培养基|送STR图谱
U343 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HEK AD293细胞、FO [Mouse myeloma]细胞、SKNBE(2)细胞
Ly1 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Intestinal Porcine Epithelial Cell line-1细胞、Sp2/0细胞、NCIH847细胞
MB231 Cells;背景说明:MDA-MB-231来自患有转移乳腺腺癌的51岁女病人的胸水。在裸鼠和ALS处理的BALB/c小鼠中,它能形成低分化腺癌(III级)。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MKN 45细胞、KNS-81细胞、NRK 49F细胞
231-luc Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BTT739细胞、SKMEL-1细胞、IB-RS-2细胞
CAL 51 Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Bac 1.2F5细胞、H1755细胞、PLMVEC细胞
XuanWei Lung Cancer-05 Cells;背景说明:肺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM 2132细胞、HCC1806细胞、OKT3细胞
NCI-H810 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SW982细胞、Leydig细胞、DU_145细胞
LN-382 Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FRTL-5细胞、MDST8细胞、UT-7细胞
BayGenomics ES cell line CSH068 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRU442 Cells(提供STR鉴定图谱)
BBLC508.17 Cells(提供STR鉴定图谱)
Keck MirKO ES cell line Mirc8 Cells(提供STR鉴定图谱)
PCRP-ZNF341-3B10 Cells(提供STR鉴定图谱)
Neuroscreen-1 Cells(提供STR鉴定图谱)
" "PubMed=16187286; DOI=10.1002/ijc.21491
Garnis C., Lockwood W.W., Vucic E., Ge Y., Girard L., Minna J.D., Gazdar A.F., Lam S., MacAulay C., Lam W.L.
High resolution analysis of non-small cell lung cancer cell lines by whole genome tiling path array CGH.
Int. J. Cancer 118:1556-1564(2006)
PubMed=18083107; DOI=10.1016/j.cell.2007.11.025
Rikova K., Guo A.-L., Zeng Q.-F., Possemato A., Yu J., Haack H., Nardone J., Lee K., Reeves C., Li Y., Hu Y.-R., Tan Z.-P., Stokes M.P., Sullivan L., Mitchell J., Wetzel R., MacNeill J., Ren J.-M., Yuan J., Bakalarski C.E., Villen J., Kornhauser J.M., Smith B., Li D.-Q., Zhou X.-M., Gygi S.P., Gu T.-L., Polakiewicz R.D., Rush J., Comb M.J.
Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer.
Cell 131:1190-1203(2007)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20679594; DOI=10.1093/jnci/djq279; PMCID=PMC2935474
Gazdar A.F., Girard L., Lockwood W.W., Lam W.L., Minna J.D.
Lung cancer cell lines as tools for biomedical discovery and research.
J. Natl. Cancer Inst. 102:1310-1321(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22751098; DOI=10.1038/ng.2330; PMCID=PMC3408577
Zhang Z.-F., Lee J.C., Lin L.-P., Olivas V., Au V., LaFramboise T., Abdel-Rahman M., Wang X.-Q., Levine A.D., Rho J.K., Choi Y.J., Choi C.-M., Kim S.-W., Jang S.J., Park Y.S., Kim W.S., Lee D.H., Lee J.-S., Miller V.A., Arcila M.E., Ladanyi M., Moonsamy P., Sawyers C.L., Boggon T.J., Ma P.C., Costa C., Taron M., Rosell R., Halmos B., Bivona T.G.
Activation of the AXL kinase causes resistance to EGFR-targeted therapy in lung cancer.
Nat. Genet. 44:852-860(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=23733853; DOI=10.1101/gr.152322.112; PMCID=PMC3759720
Jia P.-L., Jin H.-L., Meador C.B., Xia J.-F., Ohashi K., Liu L., Pirazzoli V., Dahlman K.B., Politi K., Michor F., Zhao Z.-M., Pao W.
Next-generation sequencing of paired tyrosine kinase inhibitor-sensitive and -resistant EGFR mutant lung cancer cell lines identifies spectrum of DNA changes associated with drug resistance.
Genome Res. 23:1434-1445(2013)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30038707; DOI=10.18632/oncotarget.25642; PMCID=PMC6049873
Du L.-Q., Zhao Z.-Z., Suraokar M.B., Shelton S.S., Ma X.-Y., Hsiao T.-H., Minna J.D., Wistuba I.I., Pertsemlidis A.
LMO1 functions as an oncogene by regulating TTK expression and correlates with neuroendocrine differentiation of lung cancer.
Oncotarget 9:29601-29618(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31803961; DOI=10.1002/jcb.29564; PMCID=PMC7496084
Mulshine J.L., Ujhazy P., Antman M., Burgess C.M., Kuzmin I.A., Bunn P.A. Jr., Johnson B.E., Roth J.A., Pass H.I., Ross S.M., Aldige C.R., Wistuba I.I., Minna J.D.
From clinical specimens to human cancer preclinical models -- a journey the NCI-cell line database-25 years later.
J. Cell. Biochem. 121:3986-3999(2020)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)"