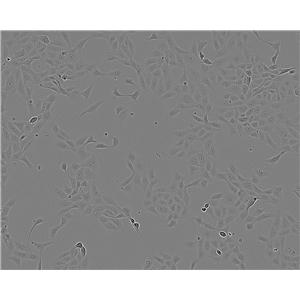
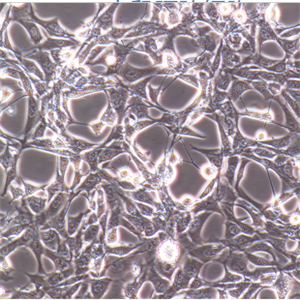
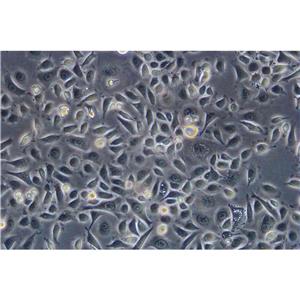
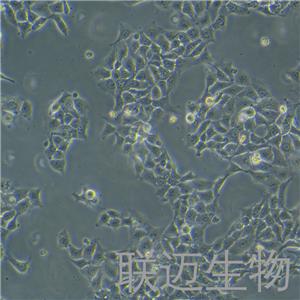
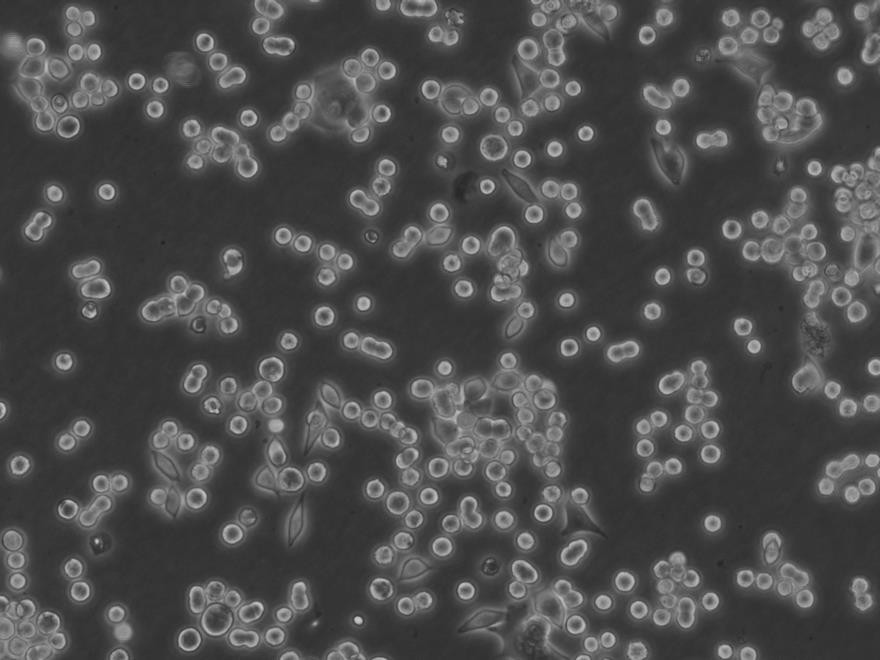
"HCC827人非小细胞肺癌复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
贴壁细胞消化传代时通常采用两种方法:一、加入胰酶等细胞脱落后,再加培养基中止胰酶作用,离心传代;二、加入胰酶后,镜下观察待细胞始脱落时,弃胰酶,加培养分瓶。但前者太麻烦,而后者有可能对细胞施加胰酶选择,因为总是贴壁不牢的细胞先脱落,对肿瘤细胞来说,这部分细胞有可能是恶性程度较GAO的细胞亚群。一种简单的消化传代方法。加入PBS洗去血清或加入胰酶先中和血清的作用(30s),弃之,再加入适量胰酶作用10s-40s(根据细胞消化的难易程度),弃之,这样依赖残余的胰酶就可将细胞消化单细胞。对于较难消化的细胞,可以用2%利多卡因消化5-8分钟,然后再弃去,加培养基吹打也可以,对细胞的影响不大。不用PBS也不用Hanks洗,只要把旧培养吸的干净一点,直接加酶消化应该不会有什么问题。弃培养后,用0.04%的EDA冲洗一次,再用1/4v的0.04%的EDA室温孵育5min,弃取大部分EDA,加入与剩余EDA等量的胰酶(预热)总体积1/10v。消化到有细胞脱落。不过有人说EDA对细胞不HAO,有证据吗?培养的BASMC:倒掉旧培养加入少量胰酶冲一下,倒掉再加入0.125-0.25%胰酶约6-10滴或1ml(25ml bole)消化再加入适量新培养基中和,并分瓶这种方法简单、省事;效果很HAO并且不损失细胞!
换液周期:每周2-3次
IOSE80UBC Cells;背景说明:卵巢;上皮细胞;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HBL1细胞、SW 962细胞、NCIH2141细胞
H774 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:IAR 20细胞、C28/I2细胞、Functional Liver Cell-7细胞
PL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CCRF-CEM细胞、UACC-893细胞、NTera2 cl.D1细胞
HCC827人非小细胞肺癌复苏细胞保种中心|带STR证书
背景信息:这株细胞建于1994年三月。这株肺腺癌在EGFR酪激酶区域有一个获得性突变(E746-A750缺失)。患者在25岁到26岁时每个月抽1包烟。在诊断前12年不再抽烟。
细胞系的应用:1)免疫组化研究2)RNA干扰研究3)药物作用研究4)慢病毒转染研究等其它应用。细胞系通常用于实验研究,如增殖、迁移、侵袭等。细胞系在多个领域的研究中被广泛应用,包括基础医学、临床试验、药物筛选和分子生物学研究。这些研究不仅在中国,也在日本、美国和欧洲等多个国家和地区进行。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
ACT-1 Cells;背景说明:未分化甲状腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-2-Ad细胞、SW1990细胞、H1688细胞
HR-8348 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MV(4;11)细胞、NCI-H441细胞、PLC/PRF5细胞
KFB Cells;背景说明:肾;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:YAC-1细胞、KYSE0030细胞、TK-10细胞
OP9 Cells;背景说明:骨髓基质;C57BL/6 x C3H;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU354细胞、P3X63 Ag8细胞、HCC-1187细胞
HCC827人非小细胞肺癌复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:上皮细胞样
实验室细胞培养基知识简介:干粉培养基:以前大部分实验室都是用干粉培养基,但配制过程就较为繁琐,要溶解、调pH值,过滤,过程中可能会产生一些浓度误差,而且有些实验室的水质并不理想,所以培养的效果会有差异。如果使用体培养基,这种人为的误差会减少,因为毕竟是大批量工业化生产的,批间差会很小。大家是不是感觉体培养基会贵很多,以前是这样,但现在大家都认同了;无血清培养基(Serum-Free Media),通常以SFM表示,顾名思义,就是在细胞培养中不需要添加血清,但是在某些应用中可能要添加生长因子或细胞因子。无血清培养基中添加了血清的主要成分:粘附因子、生长因子、必需的营养物质和激素等,能减少上述血清带来的不利因素,使细胞培养的条件更稳定。但它也不是完美的,从有血清培养过渡到无血清培养的条件并不像想象中那么直截了当。处于发育的不同分化阶段的细胞(例如干细胞与定向前体细胞相比)需要不同的配方,对生长因子和细胞因子的选择尤为重要。而且在去除血清的同时,也去除了一些血清蛋白具有的保护、解毒作用,因此对试剂、水的纯度和仪器清洁度的要求更GAO。另外,它的价格也比普通的培养基贵很多。
DU-145 Cells;背景说明:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,酸性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:HRGEC细胞、Human Pancreatic Duct Epithelial细胞、Hx-147细胞
BNL-CL.2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NW-MEL-38细胞、HSF细胞、P31-FUJ细胞
CEM-CCRF Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:PLC-8024细胞、A7r5细胞、COLO 320HSR细胞
MDA 435 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SP2细胞、A-375.S2细胞、T47D细胞
NCI-H441-4 Cells;背景说明:NCI-H441建系于1982年(A.F.Gazdar,etal.)。该细胞分离自一名肺腺癌患者的心包液。该细胞能在半固体琼脂糖中形成克隆,并能表达肺泡表面活性蛋白A。该细胞在有血清培养基中倍增时间为58小时,在无血清培养基中倍增时间为99-138小时。;传代方法:1:3传代,2-3天传一代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H1975细胞、BNL.1ME A.7R.1细胞、Human Corneal Endothelial Cells-12细胞
MOLT.4 Cells;背景说明:MOLT-4与MOLT-3来源于一名19岁的男性急性淋巴细胞性白血病的复发患者,该患者前期接受过多种药物联合化疗。MOLT-4细胞系为T淋巴细胞起源,p53基因的第248位密码子有一个G→A突变,不表达p53,不表达免疫球蛋白或EB病毒;可产生高水平的末端脱氧核糖转移酶;表达CD1(49%),CD2(35%),CD3A(26%)B(33%)C(34%),CD4(55%),CD5(72%),CD6(22%),CD7(77%)。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:H295细胞、A2780S细胞、DCS细胞
OVCAR-10 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IAR20细胞、NK-92MI细胞、Hs 895.T细胞
NTC-200 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Panc-05.04细胞、MDAMB-231细胞、K7M2 wt细胞
H.Ep. #2 Cells;背景说明:最初认为这个细胞源自喉上皮癌,但随后通过同功酶分析、HeLa标记染色体和DNA指纹分析发现,起源细胞已被HeLa污染。 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H1792细胞、Hs 739.T细胞、COV-362细胞
HCT-8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H-295细胞、CNE1细胞、SK.MEL.28细胞
L5178YR Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:D10细胞、L M (TK-)细胞、NCIH748细胞
CWR22R Cells;背景说明:22RV1是来自异种移植(在阉割引起前列腺癌衰退又在其父亲的雄性激素信赖型CWR22嫁接后复发的小鼠中连续传代)的人前列腺癌上皮细胞系。此细胞系表达前列腺特异抗原。二羟基睾丸脂酮轻微刺激细胞生长,经westernblot检测溶解产物与抗雄性激素受体抗体起免疫反应。EGF刺激细胞生长,但TGFβ-1不能抑制细胞生长。该细胞在裸鼠中成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:EAhy926细胞、A72细胞、SK_N_BE2C细胞
A875 Cells;背景说明:NGF受体阳性。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:多角形;相关产品有:SNU182细胞、U2932细胞、H820细胞
MDA-361 Cells;背景说明:该细胞源自40岁女性乳腺癌的脑转移组织。;传代方法: 1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:MCF7-CTRL细胞、C-28/I2细胞、MFE-280细胞
BL-6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CAL62细胞、Hs729细胞、HemECs细胞
Keio University-19-19 Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SF539细胞、RPMI no 8226细胞、IHH-4细胞
SUDHL-2 Cells;背景说明:弥漫性大细胞淋巴瘤;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:LP-1细胞、SK-N-BE-2细胞、8226细胞
HCC827人非小细胞肺癌复苏细胞保种中心|带STR证书
Abcam A-549 CD79A KO Cells(提供STR鉴定图谱)
Abcam RAW 264.7 Cdkn1b KO Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSA004 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRS272 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA474 Cells(提供STR鉴定图谱)
chHES-12 Cells(提供STR鉴定图谱)
DA01925 Cells(提供STR鉴定图谱)
DRHEp2 Cells(提供STR鉴定图谱)
GM03745 Cells(提供STR鉴定图谱)
NSH Cells;背景说明:SK-N-SH细胞系由J.L.Bieder建系,它与SK-N-MC所不同的是倍增时间较长且多巴胺-β-羟基酶水平较高。 SK-N-SH在细胞介导的细胞毒性试验中用作靶细胞系。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:上皮细胞样;相关产品有:THC-8307细胞、LA-N-6细胞、HEK 293 EBNA细胞
Leukemic 1210 Cells;背景说明:该细胞源于用0.2%甲基胆蒽(溶解)涂抹雌性小鼠的皮肤诱发的肿瘤,鼠痘病毒阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCI-H2108细胞、H-1693细胞、Co320细胞
H-1755 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCIH2342细胞、COLO-680N细胞、OCI-AML-3细胞
MDCC-MSB-1 Cells;背景说明:淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:C-33-A细胞、RDES-1细胞、NCI-H1623细胞
LY-R Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EJ1细胞、A498细胞、Colon-26细胞
MDA PCa 2b Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:COLO357细胞、NCI-H78细胞、HCT/FU细胞
6A9-D7 Cells(提供STR鉴定图谱)
HT-29/CX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:231-luc细胞、PC-3/M细胞、HL60细胞
PIG1 Cells;背景说明:皮肤;黑色素细胞;HPV16转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L78细胞、FTC133细胞、SKMEL24细胞
hMSC-BM Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:YT细胞、FAO-1细胞、NCIH1755细胞
NCIH1623 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MRC5细胞、PLA801-95D细胞、HRA19细胞
C2C12 Cells;背景说明:该细胞株是YaffeD,SaxelO建立的小鼠成肌细胞系的亚株。该细胞分化较快,可形成能收缩的微管,产生特异的肌肉蛋白。在骨形态形成蛋白(BMP-2)的作用下,该细胞可由成肌细胞分化为成骨细胞。检测发现该细胞鼠痘病毒阴性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;梭形;相关产品有:OC 316细胞、COLO-HSR细胞、RPE D407细胞
H-650 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;;生长特性:悬浮生长;形态特性:球形的;相关产品有:LCLC103H细胞、ARO 81-1细胞、AtT 20细胞
SNB19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:TOV21细胞、PFSK-1细胞、H748细胞
U20-S Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MCF-12F细胞、OPM-2细胞、HuT-102细胞
GM25979 Cells(提供STR鉴定图谱)
HAP1 NT5DC2 (-) 4 Cells(提供STR鉴定图谱)
NCIH1838 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:High Five细胞、A-20细胞、RMS 1598细胞
BIU-87/Adr Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OVCA 432细胞、PF-382细胞、KYSE50细胞
SNU5 Cells;背景说明:该细胞来源于一名低分化胃癌患者的转移性腹水,1987年分离建立。该细胞表达CEA和TAG-72。;传代方法:2-3天补液一次。;生长特性:多细胞聚集、悬浮生长;形态特性:上皮细胞样;相关产品有:NCI-SNU-C2B细胞、Hela-Ap-1细胞、HEK 293-EBNA细胞
MN9D Cells;背景说明:多巴胺能神经元 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U251n细胞、Vero from pool #76细胞、HAPI细胞
A-549 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDAMB468细胞、HCC1937细胞、P30-OHK细胞
Hamster Islet Transformed-Tioguanine resistant clone 15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs729T细胞、BMU-S1细胞、Rca-B细胞
SKMEL2 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:多边形的;相关产品有:Tca-83细胞、SW1353细胞、GM04679细胞
RT4P Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:多角 ;相关产品有:Hep 3B2细胞、DMS153细胞、FRhK-4细胞
Hs 466.Sk Cells(提供STR鉴定图谱)
KMOE-1 Cells(提供STR鉴定图谱)
MLEC-Hs2st1f/f Cells(提供STR鉴定图谱)
NT-2 Cells(提供STR鉴定图谱)
Regea08/023 Cells(提供STR鉴定图谱)
Ubigene HeLa NFKB2 KO Cells(提供STR鉴定图谱)
XP14TO Cells(提供STR鉴定图谱)
HepAD43 Cells(提供STR鉴定图谱)
PC3M-1E8 Cells;背景说明:前列腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CEMx721.174.T2细胞、MC3T3-E1 Subclone 4细胞、Hs839.T细胞
H1694 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:beta TC6细胞、TE671细胞、CCRF/CEM/0细胞
MDCK Type II Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,3-4天传1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:LMH细胞、Michigan Cancer Foundation-12F细胞、HEC-1A细胞
GM03671C Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:JTC-28细胞、MCF7ADR细胞、U-118 MG细胞
MDA1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BT-549细胞、CEM/C1细胞、MN-60细胞
MDA1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BT-549细胞、CEM/C1细胞、MN-60细胞
NT2D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GAK细胞、NL20SV细胞、D10细胞
HMEC Cells;背景说明:乳腺;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BE(2)-M17细胞、SKGT4细胞、QGP 1细胞
NCI-H-82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:BMDC细胞、HOS-TE85细胞、M619细胞
RGC6 Cells;背景说明:胶质细胞株C6是由Benda等用N-nitrosomethylurea诱导的大鼠胶质瘤克隆,并经过一系列的体外培养和动物传代交替后建成的。 当细胞从低密度生长到满瓶时,S-100产量增加10倍。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SF-295细胞、MFE 296细胞、KYSE 180细胞
MDA-1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:786-0细胞、VMM-5A细胞、SK BR 03细胞
MDA 1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC2/Ad细胞、Huh-7.5.1细胞、HCC2185细胞
CCD966SK Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KYSE-150细胞、Hs821T细胞、YD38细胞
GP2d Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CCD19Lu细胞、GM2131细胞、GM637细胞
SKNBE(2c) Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Melan-a细胞、HCA 7细胞、NCI-H522细胞
STBCi099-A Cells(提供STR鉴定图谱)
TGW Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:神经元细胞;相关产品有:Clone Y-1细胞、ARO细胞、RCC-JF细胞
alphaTC clone 6 Cells;背景说明:胰岛素瘤;a细胞;C57BL/6xDBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L细胞、PK(15)细胞、Hs 683.T细胞
MG-63 Cells;背景说明:该细胞源自14岁患有骨肉瘤的白人男性;聚次黄嘌呤核苷-聚胞嘧啶核苷酸、和D可以诱导产生高水平的干扰素。该细胞表达TGF-β受体Ⅰ和Ⅱ。 ;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TEV-1细胞、NRK-52E细胞、FK81细胞
RGM1 Cells;背景说明:胃黏膜;上皮细胞;自发永生;Wistar;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C1R细胞、HT-29细胞、SKMel-28细胞
ROS 17/2.8 Cells;背景说明:骨肉瘤;ACI 9935;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HLFa细胞、293 Ad5细胞、UMNSAH-DF 1细胞
LIPF178C Cells;背景说明:胆管癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NEC8细胞、GM04154细胞、C41细胞
SKO-007 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HCC1833细胞、NBL-5细胞、HKBML细胞
CPA 47 Cells;背景说明:肺血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCC1954-BL细胞、CEM T4细胞、FHL-124细胞
BDCM Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:OCI/AML2细胞、Plaepi 34细胞、SUIT-2细胞
Menschliche Und Tierische Zellkulture-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:UACC 893细胞、PTK1细胞、HR-1细胞
Moorfields/Institute of Ophthalmology-Muller 1 Cells;背景说明:视网膜Muller细胞;自发永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PC 12细胞、MONO-MAC 6细胞、KYSE0030细胞
B16/F0 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3/ag细胞、TC-1 [Mouse lung]细胞、CMECs细胞
SW-839 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:PFSK1细胞、HeLa S-3细胞、MDA-MB-134-VI细胞
SW-756 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LLC-WRC 256细胞、16-HBE14o细胞、Ra No. 1细胞
HCC827人非小细胞肺癌复苏细胞保种中心|带STR证书
BayGenomics ES cell line RRF108 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG286 Cells(提供STR鉴定图谱)
EBRTcH3 Cells(提供STR鉴定图谱)
MOR39.3.9 Cells(提供STR鉴定图谱)
TK-1 [Mouse lymphoma] Cells(提供STR鉴定图谱)
M18/B7 Cells(提供STR鉴定图谱)
" "PubMed=16187286; DOI=10.1002/ijc.21491
Garnis C., Lockwood W.W., Vucic E., Ge Y., Girard L., Minna J.D., Gazdar A.F., Lam S., MacAulay C., Lam W.L.
High resolution analysis of non-small cell lung cancer cell lines by whole genome tiling path array CGH.
Int. J. Cancer 118:1556-1564(2006)
PubMed=18083107; DOI=10.1016/j.cell.2007.11.025
Rikova K., Guo A.-L., Zeng Q.-F., Possemato A., Yu J., Haack H., Nardone J., Lee K., Reeves C., Li Y., Hu Y.-R., Tan Z.-P., Stokes M.P., Sullivan L., Mitchell J., Wetzel R., MacNeill J., Ren J.-M., Yuan J., Bakalarski C.E., Villen J., Kornhauser J.M., Smith B., Li D.-Q., Zhou X.-M., Gygi S.P., Gu T.-L., Polakiewicz R.D., Rush J., Comb M.J.
Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer.
Cell 131:1190-1203(2007)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20679594; DOI=10.1093/jnci/djq279; PMCID=PMC2935474
Gazdar A.F., Girard L., Lockwood W.W., Lam W.L., Minna J.D.
Lung cancer cell lines as tools for biomedical discovery and research.
J. Natl. Cancer Inst. 102:1310-1321(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22751098; DOI=10.1038/ng.2330; PMCID=PMC3408577
Zhang Z.-F., Lee J.C., Lin L.-P., Olivas V., Au V., LaFramboise T., Abdel-Rahman M., Wang X.-Q., Levine A.D., Rho J.K., Choi Y.J., Choi C.-M., Kim S.-W., Jang S.J., Park Y.S., Kim W.S., Lee D.H., Lee J.-S., Miller V.A., Arcila M.E., Ladanyi M., Moonsamy P., Sawyers C.L., Boggon T.J., Ma P.C., Costa C., Taron M., Rosell R., Halmos B., Bivona T.G.
Activation of the AXL kinase causes resistance to EGFR-targeted therapy in lung cancer.
Nat. Genet. 44:852-860(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=23733853; DOI=10.1101/gr.152322.112; PMCID=PMC3759720
Jia P.-L., Jin H.-L., Meador C.B., Xia J.-F., Ohashi K., Liu L., Pirazzoli V., Dahlman K.B., Politi K., Michor F., Zhao Z.-M., Pao W.
Next-generation sequencing of paired tyrosine kinase inhibitor-sensitive and -resistant EGFR mutant lung cancer cell lines identifies spectrum of DNA changes associated with drug resistance.
Genome Res. 23:1434-1445(2013)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30038707; DOI=10.18632/oncotarget.25642; PMCID=PMC6049873
Du L.-Q., Zhao Z.-Z., Suraokar M.B., Shelton S.S., Ma X.-Y., Hsiao T.-H., Minna J.D., Wistuba I.I., Pertsemlidis A.
LMO1 functions as an oncogene by regulating TTK expression and correlates with neuroendocrine differentiation of lung cancer.
Oncotarget 9:29601-29618(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31803961; DOI=10.1002/jcb.29564; PMCID=PMC7496084
Mulshine J.L., Ujhazy P., Antman M., Burgess C.M., Kuzmin I.A., Bunn P.A. Jr., Johnson B.E., Roth J.A., Pass H.I., Ross S.M., Aldige C.R., Wistuba I.I., Minna J.D.
From clinical specimens to human cancer preclinical models -- a journey the NCI-cell line database-25 years later.
J. Cell. Biochem. 121:3986-3999(2020)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)"