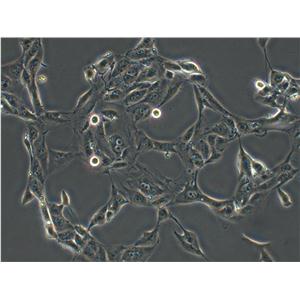
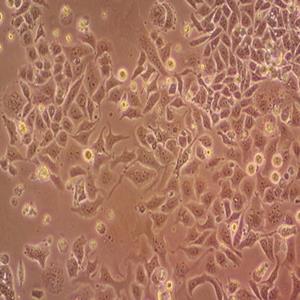
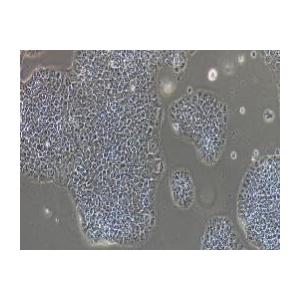
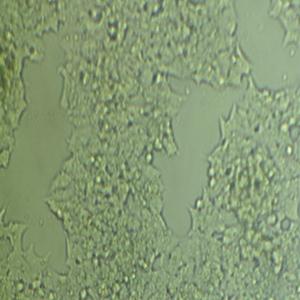
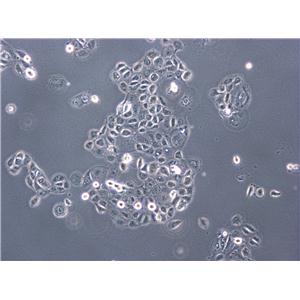
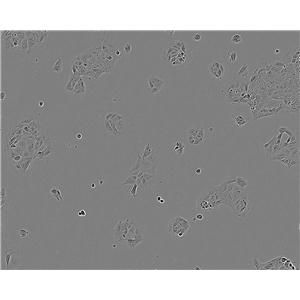
"BxPC-3人原位胰腺腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的应用:1)免疫组化研究2)RNA干扰研究3)药物作用研究4)慢病毒转染研究等其它应用。细胞系通常用于实验研究,如增殖、迁移、侵袭等。细胞系在多个领域的研究中被广泛应用,包括基础医学、临床试验、药物筛选和分子生物学研究。这些研究不仅在中国,也在日本、美国和欧洲等多个国家和地区进行。
换液周期:每周2-3次
YD38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AG06814-J细胞、OVCAR-8/ADR细胞、143B细胞
RCC4 Cells;背景说明:肾透明细胞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KM933细胞、NCI-SNU-423细胞、HEK 293-F细胞
SU86_86 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HFL-I细胞、Melan-a细胞、L-Wnt3A细胞
BxPC-3人原位胰腺腺癌细胞代次低|培养基|送STR图谱
背景信息:这个细胞株不表达囊肿性纤维化跨膜电导调节子(CFTR)。CFTR阳性的细胞株是Capan-1(ACHTB-79)。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
BxPC-3人原位胰腺腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
HEL92.1.7 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:MLMEC细胞、MDA157细胞、GM05372细胞
143 B Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代;每周换液2-3次;生长特性:贴壁生长;形态特性:混合型;相关产品有:NCI-SNU-407细胞、ARO-81细胞、SKM-1细胞
RS411 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:NT2-D1细胞、LAN-5细胞、HEK293-FT细胞
IPLB-SF 21AE Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKO3细胞、Intestinal Porcine Epithelial Cell line-J2细胞、HCC-2108细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞传代培养实验:体外培养的原代细胞或细胞株要在体外持续地培养就必须传代,以便获得稳定的细胞株或得到大量的同种细胞,并维持细胞种的延续。培养的细胞形成单层汇合以后,由于密度过大生存空间不足而引起营养枯竭,将培养的细胞分散,从容器中取出,以1:2或1:3以上的比率转移到另外的容器中进行培养,即为传代培养;细胞“一代”指从细胞接种到分离再培养的一段期间,与细胞世代或倍增不同。在一代中,细胞培增3~6次。细胞传代后,一般经过三个阶段:游离期、指数增生期和停止期。常用细胞分裂指数表示细胞增殖的旺盛程度,即细胞群的分裂相数/100个细胞。一般细胞分裂指数介于0.2%~0.5%,肿瘤细胞可达3~5%;细胞接种2~3天分裂增殖旺盛,是活力ZuiHAO时期,称指数增生期(对数生长期),适宜进行各种试验。实验步骤:1.将长成的培养细胞从二氧化碳培养箱中取出,在超净工作台中倒掉瓶内的培养,加入少许消化。(以面盖住细胞为宜),静置5~10分钟。2.在倒置镜下观察被消化的细胞,如果细胞变圆,相互之间不再连接成片,这时应立即在超净台中将消化倒掉,加入3~5ml新鲜培养,吹打,制成细胞悬。3.将细胞悬吸出2ml左右,加到另一个培养瓶中并向每个瓶中分别加3ml左右培养,盖HAO瓶塞,送回二氧化碳培养箱中,继续进行培养。一般情况,传代后的细胞在2小时左右就能附着在培养瓶壁上,2~4天就可在瓶内形成单层,需要再次进行传代。
Panc-02 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:UM-UC3细胞、LC-2 ad细胞、MARC-145细胞
L132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW 579细胞、UCLA SO M14细胞、SK-LMS-1细胞
Roswell Park Memorial Institute 8402 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Centre Antoine Lacassagne-39细胞、NCI-H378细胞、CAL-33细胞
SKGIIIA Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Strain L-929细胞、MDAMB415细胞、H1672细胞
MS 751 Cells;背景说明:这株细胞是J. Sykes于1974年建立的(参考ATCC HTB-33)。有报告称MS751细胞含有人乳头状瘤病毒18 (HPV-18)序列。[22995] [23180]后来发现MS751细胞包含HPV-45基因组的一部分,而其中E6/E7区域表达呈poly(A)+RNA的形式。[49721];传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:OVCA5细胞、BALB 3T3 clone A31细胞、NCI-H520细胞
Fao Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮细胞;相关产品有:RPMI #8226细胞、Keio University-19-19细胞、BEL 7402细胞
E6-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FRhK-4细胞、293 c18细胞、143 B细胞
HDLM-2 Cells;背景说明:霍奇金淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MHCC-97细胞、D407细胞、H2106细胞
LC1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PAa细胞、EA. hy 926细胞、AN3 CA细胞
MCF-12A Cells;背景说明:非致瘤性乳腺上皮细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LSC-1细胞、SKHEP-1细胞、EB-2细胞
L-M[TK-] Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FRTL 5细胞、H2286细胞、C57 Mouse Tumor 93细胞
TSU-Pr1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H1568细胞、COLO-829细胞、R.K.13细胞
786O Cells;背景说明:该细胞源自一位原发性肾透明细胞癌患者。该细胞有微绒毛和桥粒,能在软琼脂上生长。此细胞生成一种PTH(甲状旁腺素)样的多肽,与乳癌和肺癌中生成的肽相似,其N端序列与PTH相似,具有PTH样活性,分子量为6000D。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCI-Ly 1细胞、NCI/ADR-RES细胞、NCI-H441-4细胞
ARIP Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:AR4-2J细胞、H1666细胞、CFSC-2G细胞
WM 451-Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC2185细胞、H-716细胞、MKN 45细胞
293 H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RGC5细胞、NCI-H2291细胞、H-125细胞
A2780CP70 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P3 NS1 Ag4/1细胞、THLE2细胞、B16 melanoma细胞
Abcam A-549 ISM1 KO 1 Cells(提供STR鉴定图谱)
Abcam U-87MG GABRA1 KO Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSG038 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRT394 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC444 Cells(提供STR鉴定图谱)
CHO CT52 Cells(提供STR鉴定图谱)
DA02320 Cells(提供STR鉴定图谱)
DA05090 Cells(提供STR鉴定图谱)
GAK14 Cells(提供STR鉴定图谱)
TGBC11T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Co 205细胞、CLONE M3细胞、CMT167细胞
BxPC-3人原位胰腺腺癌细胞代次低|培养基|送STR图谱
MDA1386 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BT-549细胞、CEM/C1细胞、MN-60细胞
NCI-H2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:NCI-H102细胞、Mouse podocyte细胞、Evsa-T细胞
NCIH1648 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NRK-49F细胞、Hs343T细胞、NS-1细胞
Human Lung Microvascular Endothelial Cell line-5a Cells;背景说明:肺微血管;内皮细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HS940细胞、S.B.细胞、Ca761细胞
LS-411N Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:N-87细胞、H1648细胞、MESSA细胞
168FAR Cells(提供STR鉴定图谱)
NCIH295R Cells;背景说明:肾上腺皮质癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TI-73细胞、NCIH1755细胞、NCI-H2591细胞
RAW 264.7 Cells;背景说明:单核巨噬细胞白血病;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H2172细胞、MFM-223细胞、SK-GT-4细胞
H-1876 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:DHL-6细胞、SNU-182细胞、AML-193细胞
ECV 304 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SK-MEL24细胞、NCI-H596细胞、THLE-2细胞
H-740 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:UACC 812细胞、H-1436细胞、HNE-2细胞
HUSMC Cells;背景说明:子宫;平滑肌细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-847细胞、Hep G2细胞、COLO 320F细胞
HIEC Cells;背景说明:肠;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs 600.T细胞、DoHH-2细胞、CCRF.CEM细胞
343MG Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Fox/NY细胞、ECC-12细胞、BCP-1细胞
GM18365 Cells(提供STR鉴定图谱)
HAP1 IFI44L (-) Cells(提供STR鉴定图谱)
RH35 Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs606T细胞、SK-N-BE(2)C细胞、HCE-T细胞
639V Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs695T细胞、OPM2细胞、SUDHL-4细胞
Ramos.G6.C10 Cells;背景说明:详见相关文献介绍;传代方法: 维持细胞浓度在2×105/ml-1×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MC-3T3细胞、ONS-76细胞、HUC细胞
P3JHR-1 Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:SV40-MES13细胞、NCI-H920细胞、SKNBE细胞
KP-2 Cells;背景说明:胰腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NBL-S细胞、Colo-206F细胞、NCI-SNU-16细胞
HS-294 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:混合星状和多边形;相关产品有:KPL-4细胞、RIN-m5F细胞、Huh 7.5细胞
A2008 Cells;背景说明:宫颈鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OsACL细胞、AU-Mel细胞、H-35 Reuber细胞
H7721 Cells;背景说明:用Northernblot方法,未能检测到细胞中1.3kbLFIRE-1/HFREP-1mRNA的表达。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCI-Ly 18细胞、NCIH2073细胞、PY8119细胞
HPS0248 Cells(提供STR鉴定图谱)
JHU234i Cells(提供STR鉴定图谱)
MDA-MB-231/Luc Cells(提供STR鉴定图谱)
ND36883 Cells(提供STR鉴定图谱)
PrecisION hKv4.3/KCHiP1-CHO Cells(提供STR鉴定图谱)
Ubigene A-549 CACNG6 KO Cells(提供STR鉴定图谱)
UOK102 Cells(提供STR鉴定图谱)
Hep-G2-THRA MUT p.I116N;A225T;M388I Cells(提供STR鉴定图谱)
COLO680N Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCC-HSF-1细胞、SNU182细胞、LS411N细胞
B/C3T3 Cells;背景说明:胚胎;成纤维;自发永生;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1672细胞、HMC细胞、HNE3细胞
WERI-Rb 1 Cells;背景说明:WERI-Rb-I细胞株是1974年R.M. McFall 和 T.W. Sery建立的两株人眼癌细胞系中的一株。 细胞能在Difco Bacto-Agar中存活但不形成克隆。 扫描电镜显示在表面囊泡,板状伪足和微绒毛在数量上和频率上的改变。 细胞分化研究,肿瘤治疗的动物模型和生化评价都涉及这株细胞。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:圆形细胞聚集成葡萄状;相关产品有:C4-2细胞、RK13细胞、Y3-Ag1,2,3细胞
AN3-CA Cells;背景说明:AN3CA细胞建系于1964年。它衍生于子宫内膜癌患者淋巴结转移组织,具有癌细胞的基本特性,能在体外长期传代培养,接种实验动物产生明显肿瘤。但细胞的生物学特性及超微结构尚未深入研究,仅发现该细胞系促黑激素合成为阴性。细胞常用于人子宫内膜癌细胞生物学及其相关特性研究。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:UCLA-SO-M20细胞、GM16136细胞、SNK-1细胞
SK-MEL-31 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CHO cell clone K1细胞、Centre Antoine Lacassagne-39细胞、PC 61.5.3细胞
SK-MEL-31 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CHO cell clone K1细胞、Centre Antoine Lacassagne-39细胞、PC 61.5.3细胞
GM-215 Cells;背景说明:肺;自发永生;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2286细胞、KP-N-YN细胞、MonoMac 6细胞
Calf Pulmonary Artery Endothelial Cells;背景说明:肺血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GTL16细胞、T47-D细胞、MDA-468细胞
HELF Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:BAR-T细胞、MBT-2细胞、CAL-120细胞
GM637A Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HO-8910 PM细胞、686LN-M4e细胞、Hs 746.T细胞
SK-MEL24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:IGROV 1细胞、EFM192B细胞、COLO-1细胞
SK-MEL-1 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在1×105-2×105,每周补液2-3次。;生长特性:悬浮生长;形态特性:球形的;相关产品有:BV173细胞、ACC-M细胞、MDA-436细胞
HUT-226 Cells;背景说明:1980年分离建立。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:RCC23细胞、COLO-699细胞、OVCAR10细胞
MUS-M1 Cells;背景说明:小肠;平滑肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KE-37细胞、PCI-4M细胞、BNCL2细胞
BT Cells;背景说明:鼻甲;自发永生;Holstein;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OCI-Ly 19细胞、SHP-77细胞、SCC25细胞
SL1 Cells(提供STR鉴定图谱)
H-2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:PK 15细胞、NuTu-19细胞、HCC-1569细胞
HT144mel Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:MDAMB435细胞、HPAF细胞、CHL1细胞
Human Epidermoid carcinoma #2 Cells;背景说明:最初认为这个细胞源自喉上皮癌,但随后通过同功酶分析、HeLa标记染色体和DNA指纹分析发现,起源细胞已被HeLa污染。 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCC1569细胞、NBL-4细胞、NS20Y细胞
NCI-H508 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:293-H细胞、NCI660细胞、JVM3细胞
NCIH64 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Colo-201细胞、HL-1细胞、HOP62细胞
HR-8348 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MV(4;11)细胞、NCI-H441细胞、PLC/PRF5细胞
BxPC-3人原位胰腺腺癌细胞代次低|培养基|送STR图谱
EFM-192C Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C-33A细胞、MDAPC2B细胞、293-H细胞
HCC-1806 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:D324 Med细胞、RBL 2H3细胞、HCS2/8细胞
PC-9/GR Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PC 61-5-3细胞、NCI-H2172细胞、MDA MB 134VI细胞
GalK1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CaEs17细胞、OVCAR 432细胞、CEMC7细胞
K 562 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Walker/LLC-WRC 256细胞、Lewis lung carcinoma line 1细胞、SNU16细胞
SG231 Cells;背景说明:肝内胆管癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:K7M2细胞、BCP1细胞、HuH7细胞
MX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:QBC-939细胞、LS-411N细胞、L-6 myoblast细胞
Mo 59J Cells;背景说明:详见相关文献介绍;传代方法:1:6-1:8传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Hs852细胞、NCI-SNU-668细胞、MRC 5细胞
BayGenomics ES cell line CSG413 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRU317 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC771 Cells(提供STR鉴定图谱)
Keck MirKO ES cell line Mir124a-3 Cells(提供STR鉴定图谱)
PCRP-ZNF202-1C2 Cells(提供STR鉴定图谱)
mTX3 Cells(提供STR鉴定图谱)
" "PubMed=8426738
Kalthoff H., Schmiegel W.H., Roeder C., Kasche D., Schmidt A., Lauer G., Thiele H.-G., Honold G., Pantel K., Riethmuller G., Scherer E., Maurer J., Maacke H., Deppert W.
p53 and K-RAS alterations in pancreatic epithelial cell lesions.
Oncogene 8:289-298(1993)
PubMed=7961102; DOI=10.1111/j.1349-7006.1994.tb02898.x; PMCID=PMC5919355
Suwa H., Yoshimura T., Yamaguchi N., Kanehira K., Manabe T., Imamura M., Hiai H., Fukumoto M.
K-ras and p53 alterations in genomic DNA and transcripts of human pancreatic adenocarcinoma cell lines.
Jpn. J. Cancer Res. 85:1005-1014(1994)
PubMed=8026879; DOI=10.1002/ijc.2910580207
Berrozpe G., Schaeffer J., Peinado M.A., Real F.X., Perucho M.
Comparative analysis of mutations in the p53 and K-ras genes in pancreatic cancer.
Int. J. Cancer 58:185-191(1994)
PubMed=8194712; DOI=10.1016/0016-5085(94)90422-7
Simon B., Weinel R., Hohne M., Watz J., Schmidt J., Kortner G., Arnold R.
Frequent alterations of the tumor suppressor genes p53 and DCC in human pancreatic carcinoma.
Gastroenterology 106:1645-1651(1994)
PubMed=8286197; DOI=10.1038/bjc.1994.24; PMCID=PMC1968784
Lohr J.-M., Trautmann B., Gottler M., Peters S., Zauner I., Maillet B., Kloppel G.
Human ductal adenocarcinomas of the pancreas express extracellular matrix proteins.
Br. J. Cancer 69:144-151(1994)
PubMed=21607521; DOI=10.3892/or.1.6.1223
Iguchi H., Morita R., Yasuda D., Takayanagi R., Ikeda Y., Takada Y., Shimazoe T., Nawata H., Kono A.
Alterations of the p53 tumor-suppressor gene and ki-ras oncogene in human pancreatic cancer-derived cell-lines with different metastatic potential.
Oncol. Rep. 1:1223-1227(1994)
PubMed=9788440; DOI=10.1038/sj.onc.1202118
Villanueva A., Garcia C., Paules Blazquez A.B., Vicente M., Megias M., Reyes G., de Villalonga P., Agell N., Lluis F., Bachs O., Capella G.
Disruption of the antiproliferative TGF-beta signaling pathways in human pancreatic cancer cells.
Oncogene 17:1969-1978(1998)
PubMed=10027410; DOI=10.1016/S0002-9440(10)65298-4; PMCID=PMC1850008
Ghadimi B.M., Schrock E., Walker R.L., Wangsa D., Jauho A., Meltzer P.S., Ried T.
Specific chromosomal aberrations and amplification of the AIB1 nuclear receptor coactivator gene in pancreatic carcinomas.
Am. J. Pathol. 154:525-536(1999)
PubMed=10408907; DOI=10.1016/S0304-3835(98)00380-2
Bartsch D.K., Barth P., Bastian D., Ramaswamy A., Gerdes B., Chaloupka B., Deiss Y., Simon B., Schudy A.
Higher frequency of DPC4/Smad4 alterations in pancreatic cancer cell lines than in primary pancreatic adenocarcinomas.
Cancer Lett. 139:43-49(1999)
PubMed=11115575; DOI=10.3892/or.8.1.89
Sun C.-L., Yamato T., Furukawa T., Ohnishi Y., Kijima H., Horii A.
Characterization of the mutations of the K-ras, p53, p16, and SMAD4 genes in 15 human pancreatic cancer cell lines.
Oncol. Rep. 8:89-92(2001)
PubMed=11169959; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1049>3.0.CO;2-C
Sirivatanauksorn V., Sirivatanauksorn Y., Gorman P.A., Davidson J.M., Sheer D., Moore P.S., Scarpa A., Edwards P.A.W., Lemoine N.R.
Non-random chromosomal rearrangements in pancreatic cancer cell lines identified by spectral karyotyping.
Int. J. Cancer 91:350-358(2001)
PubMed=12692724; DOI=10.1007/s00428-003-0784-4
Sipos B., Moser S., Kalthoff H., Torok V., Lohr J.-M., Kloppel G.
A comprehensive characterization of pancreatic ductal carcinoma cell lines: towards the establishment of an in vitro research platform.
Virchows Arch. 442:444-452(2003)
PubMed=15126341; DOI=10.1158/0008-5472.CAN-03-3159
Heidenblad M., Schoenmakers E.F.P.M., Jonson T., Gorunova L., Veltman J.A., van Kessel A.G., Hoglund M.
Genome-wide array-based comparative genomic hybridization reveals multiple amplification targets and novel homozygous deletions in pancreatic carcinoma cell lines.
Cancer Res. 64:3052-3059(2004)
PubMed=15367885; DOI=10.1097/00006676-200410000-00004
Loukopoulos P., Kanetaka K., Takamura M., Shibata T., Sakamoto M., Hirohashi S.
Orthotopic transplantation models of pancreatic adenocarcinoma derived from cell lines and primary tumors and displaying varying metastatic activity.
Pancreas 29:193-203(2004)
PubMed=15688027; DOI=10.1038/sj.onc.1208383
Heidenblad M., Lindgren D., Veltman J.A., Jonson T., Mahlamaki E.H., Gorunova L., van Kessel A.G., Schoenmakers E.F.P.M., Hoglund M.
Microarray analyses reveal strong influence of DNA copy number alterations on the transcriptional patterns in pancreatic cancer: implications for the interpretation of genomic amplifications.
Oncogene 24:1794-1801(2005)
PubMed=16912165; DOI=10.1158/0008-5472.CAN-06-0721
Calhoun E.S., Hucl T., Gallmeier E., West K.M., Arking D.E., Maitra A., Iacobuzio-Donahue C.A., Chakravarti A., Hruban R.H., Kern S.E.
Identifying allelic loss and homozygous deletions in pancreatic cancer without matched normals using high-density single-nucleotide polymorphism arrays.
Cancer Res. 66:7920-7928(2006)
PubMed=18298655; DOI=10.1111/j.1582-4934.2008.00289.x; PMCID=PMC3828895
Pilarsky C., Ammerpohl O., Sipos B., Dahl E., Hartmann A., Wellmann A., Braunschweig T., Lohr J.-M., Jesenofsky R., Friess H., Wente M.N., Kristiansen G., Jahnke B., Denz A., Ruckert F., Schackert H.K., Kloppel G., Kalthoff H., Saeger H.-D., Grutzmann R.
Activation of Wnt signalling in stroma from pancreatic cancer identified by gene expression profiling.
J. Cell. Mol. Med. 12:2823-2835(2008)
PubMed=18380791; DOI=10.1111/j.1349-7006.2008.00779.x; PMCID=PMC11158928
Suzuki A., Shibata T., Shimada Y., Murakami Y., Horii A., Shiratori K., Hirohashi S., Inazawa J., Imoto I.
Identification of SMURF1 as a possible target for 7q21.3-22.1 amplification detected in a pancreatic cancer cell line by in-house array-based comparative genomic hybridization.
Cancer Sci. 99:986-994(2008)
CLPUB00416
Oberlin L.
Treatment of pancreatic carcinoma cell lines in vitro and vivo with a monoclonal antibody against the transferrin receptor.
Thesis VMD (2009); Justus-Liebig-Universitat Giessen; Giessen; Germany
PubMed=20037478; DOI=10.4161/cbt.8.21.9685; PMCID=PMC2824894
Kent O.A., Mullendore M.E., Wentzel E.A., Lopez-Romero P., Tan A.-C., Alvarez H., West K.M., Ochs M.F., Hidalgo M., Arking D.E., Maitra A., Mendell J.T.
A resource for analysis of microRNA expression and function in pancreatic ductal adenocarcinoma cells.
Cancer Biol. Ther. 8:2013-2024(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20418756; DOI=10.1097/MPA.0b013e3181c15963; PMCID=PMC2860631
Deer E.L., Gonzalez-Hernandez J., Coursen J.D., Shea J.E., Ngatia J.G., Scaife C.L., Firpo M.A., Mulvihill S.J.
Phenotype and genotype of pancreatic cancer cell lines.
Pancreas 39:425-435(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
DOI=10.4172/2324-9293.1000104
Wagenhauser M.U., Ruckert F., Niedergethmann M., Grutzmann R., Saeger H.-D.
Distribution of characteristic mutations in native ductal adenocarcinoma of the pancreas and pancreatic cancer cell lines.
Cell Biol. Res. Ther. 2:1000104.1-1000104.5(2013)
PubMed=25167228; DOI=10.1038/bjc.2014.475; PMCID=PMC4453732
Hamidi H., Lu M., Chau K., Anderson L., Fejzo M.S., Ginther C., Linnartz R., Zubel A., Slamon D.J., Finn R.S.
KRAS mutational subtype and copy number predict in vitro response of human pancreatic cancer cell lines to MEK inhibition.
Br. J. Cancer 111:1788-1801(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27073551; DOI=10.3892/ol.2016.4289; PMCID=PMC4812595
Fujiwara M., Kanayama K., Hirokawa Y.S., Shiraishi T.
ASF-4-1 fibroblast-rich culture increases chemoresistance and mTOR expression of pancreatic cancer BxPC-3 cells at the invasive front in vitro, and promotes tumor growth and invasion in vivo.
Oncol. Lett. 11:2773-2779(2016)
PubMed=27229158; DOI=10.1038/cgt.2016.23; PMCID=PMC5007605
Schreiber R., Mezencev R., Matyunina L.V., McDonald J.F.
Evidence for the role of microRNA 374b in acquired cisplatin resistance in pancreatic cancer cells.
Cancer Gene Ther. 23:241-245(2016)
PubMed=27259358; DOI=10.1074/mcp.M116.058313; PMCID=PMC4974343
Humphrey E.S., Su S.-P., Nagrial A.M., Hochgrafe F., Pajic M., Lehrbach G.M., Parton R.G., Yap A.S., Horvath L.G., Chang D.K., Biankin A.V., Wu J.-M., Daly R.J.
Resolution of novel pancreatic ductal adenocarcinoma subtypes by global phosphotyrosine profiling.
Mol. Cell. Proteomics 15:2671-2685(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27910856; DOI=10.1038/cgt.2016.71; PMCID=PMC5159445
Mezencev R., Matyunina L.V., Wagner G.T., McDonald J.F.
Acquired resistance of pancreatic cancer cells to cisplatin is multifactorial with cell context-dependent involvement of resistance genes.
Cancer Gene Ther. 23:446-453(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"