
4YR企业会员
发布人:上海优宁维生物科技股份有限公司
发布日期:2025/1/2 17:14:20
蛋白翻译后修饰(PTM),如磷酸化、泛素化、乙酰化、SUMO化等,在细胞信号转导、蛋白稳定和转换、蛋白识别和相互作用、空间定位等方面发挥着重要作用。上一期在小优分享了PTM在疾病研究中的综述后,很多小伙伴在后台纷纷留言要PTM检测方法&PTM研究工具,这不今天就为大家带来了PTM研究超全指南(下)~
Part 01 PTM检测方法
研究PTM时会遇到的技术问题:
低化学计量:蛋白质形态往往是亚结构化的,然而任何一种蛋白质形态都只占到蛋白质总数的1-5%,这就使得它们很难被检测到;
高度动态化:蛋白质形式是动态的,通常会对特定的信号做出短暂的响应;
细胞或组织的特异性:蛋白质形式具有细胞或组织类型特异性;
样品制备中PTM的得失:在样品处理过程中,PTM 图谱可能会发生改变;
设置对照:对照组对于验证结果的准确性至关重要。
上面列举了检测 PTM 蛋白形态时具有挑战性的一些主要技术问题,接下来小优就带大家来看看有哪些PTM的检测方法吧~
1. PTM的富集
由于低化学计量,因此我们需要富集PTM蛋白,这通常是通过免疫沉淀(IP)技术实现的1-3。IP法利用抗体、蛋白结合域、金属离子亲和力和其他附着在固体支撑基质(如琼脂糖树脂)上的特异性结合来进行富集。这些亲和基质与靶蛋白或PTM结合,而复合体裂解物中的非靶蛋白则不会被捕获,并通过洗涤步骤去除。然后使用浓缩的洗脱缓冲液将富集的PTM蛋白从支撑基质中分离出来。分离出来的蛋白质群在下游应用(如 WB或MS)中进行分析,以确定靶蛋白质是否经过翻译后修饰。
内源性 PTM 蛋白形态富集有两种方法:一种方法是使用能识别目标蛋白的亲和基质,另一种方法是使用相关 PTM 的特异性亲和基质。针对相关目标蛋白的亲和基质的IP可能会免疫沉淀出该特定蛋白的所有蛋白形式,而使用 PTM 特异性亲和基质的 IP 则可能免疫沉淀出几乎所有经该 PTM 修饰的蛋白。
2. 蛋白质印迹分析(WB)
WB是一种标准的分子生物学方法,用于从复杂的蛋白质混合物或裂解物中鉴定特定的相关蛋白质。在进行 PTM 蛋白形态分析时,将富集的样品与 SDS 混合,在SDS-PAGE胶中根据大小分离到特定区域,然后转移到印迹膜上,并通过抗体识别进行特异性鉴定。
图1A显示了泛素化的表皮生长因子受体(EGFR)用泛素亲和试剂富集,并用EGFR抗体显色的例子,WB结果显示泛素化的EGFR具有明确的特征。从裂解缓冲液中移除去泛素化酶抑制剂 NEM 后,泛素化的EGFR条带消失,从而使去泛素化得以发生。对照组使用EGFR抗体进行IP富集,然后用泛素抗体探测富集的蛋白质,图1B显示了用这种方法检测到的最小泛素化的EGFR。这种显著差异可能是由于结合的泛素蛋白阻断了EGFR抗体的识别位点(图 1C 和 D)。先前的研究表明,一些PTM修饰可能会阻断靶蛋白上的抗体结合位点,并阻断与抗体的相互作用,从而产生假阴性结果4。
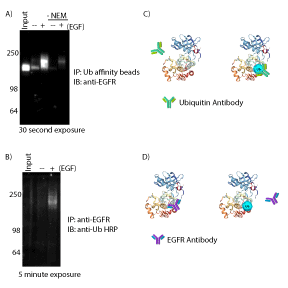 图1:EGFR泛素化的检测
图1:EGFR泛素化的检测
3. 质谱分析(MS)
质谱法(MS)是一种分析方法,可以根据质荷比来鉴定PTM蛋白形态。目前有两种不同的MS方法来研究PTM蛋白形态,一种是自下而上的分析,另一种是自上而下的分析。质谱法的优势在于检测的精确性、PTM 位点特异性以及无需抗体。使用MS时面临的技术挑战包括蛋白质丰度偏差5、灵敏度6和具备该方法的专业知识,如样品准备、消化策略、分馏方法和其他注意事项7。利用 MS 研究 PTM 蛋白形态既需要对生物模型有全面的了解,也需要仪器/分析方面的专业技术,因此,想要利用这种方法成功地研究 PTM 蛋白形态需要分子生物学家和 MS 专家的通力合作。
4. 过表达与突变
过度表达是一种成熟的系统,在这种系统中,将靶蛋白的标记质粒转染到细胞中,从而提高其表达水平。表达的蛋白质通常带有一个标签(如 His),该标签能被特异性的抗体识别。表达量的增加和抗体的优化富集提高了鉴定 PTM 蛋白质形态的几率,通常采用 WB进行后续分析。当蛋白质形态难以进行内源性的研究时,过表达系统就显得尤为重要,因为它可以通过突变方法控制表达、定位等。并且,突变方法使特定位点的PTM检测和表征成为了可能。
5. 体外生化分析
生化分析是利用靶蛋白的纯化或体外翻译来确定它是否可以被特异性的PTM修饰,将纯化的蛋白质、酶(如泛素连接酶)和适当的底物(如泛素)、辅因子等一起加入试管中,经过孵育再对样品进行WB分析。然而,体外生化分析并不适用于所有类型的PTM,它通常被作研究磷酸化8、泛素化9、SUMO化10和其他常规PTM。此外,进行体外生化分析的一个难点是获得纯化的蛋白和修饰酶11。
6. 邻近连接技术(PLA)
PLA是一种新的免疫分析技术,可用于研究蛋白相互作用和PTM,PLA 的独特之处在于它能够鉴定固定组织和细胞中的蛋白质形态12。PLA-PTM 的原理是利用两种抗体:一种抗体靶向PTM,另一种抗体结合特定蛋白质13。最初的步骤与免疫荧光染色类似,一抗结合目标表位,二抗识别各自的一抗,而PLA 的不同之处在于这些二抗上共价连接了短 DNA 链(这些抗体-DNA 复合物称为 PLA 探针)。如果两个 PLA 探针距离足够近(可能是因为两个抗体都与蛋白结合),它们就会形成环状 DNA,然后对环状 DNA 进行 PCR 扩增,加入荧光标记的互补 DNA 探针进行观察。由于 DNA 的放大倍数高达几百倍,显微镜下只能看到极少分子的荧光信号。
7. 其它新兴技术
对 PTM 领域产生深远影响的一项新兴技术是遗传密码扩增,可以在不需要特定修饰酶的情况下,将PTM特异性地添加到靶蛋白上14。拥有同源纯化的 PTM 蛋白形态对于结构研究、体外功能研究等至关重要。除了遗传密码扩增外,还有许多其他新兴技术,例如,核磁共振光谱法可用于蛋白质形式的位点特异性和结构洞察15,热解离测定法可用于检测酶的 PTM 去除16,纳米孔技术可用于无标记 PTM 检测17。
针对每一种特定的PTM,也都开发了很多专业的检测方法。例如,研究人员利用活细胞中原生蛋白质的热点热剖析技术(Hotspot Thermal Profiling)来确定哪些特定位点的磷酸化事件会改变蛋白质的稳定性18;利用新型泛素剪切机制来深入了解 poylubiquitin 链和结构19。此外,研究人员还开发了新方法,利用反相高效液相色谱法(HPLC)量化赖氨酸乙酰转移酶等调控酶的动力学参数20。
Part 02 PTM研究工具
1. PTMScan®工具
Cell Signaling Technology (CST)创建了PTMScan®技术,这一专利蛋白质组实验方法可在进行液相色谱串联质谱(LC-MS/MS)分析之前,使用由CST开发且经验证的PTM抗体和基序特异性抗体来富集含PTM的肽段。使用PTMScan技术可识别并量化成百上千个即使丰度很低的PTM位点,然后可将这些位点精选至最相关的可行性靶标上。相较于固相金属亲和层析(IMAC)等其他策略PTMScan技术使用了更集中式的PTM肽段富集法。现在CST推出的新一代PTMScan® HS试剂盒,相比于PTMScan®更灵敏、更特异和更便捷。
PTMScan® HS试剂盒的优势有:
① 所需样品更少:与传统试剂盒相比,消化高效,免疫捕获灵敏度高,使降低对样本量的要求成为可能;
② PTM肽更纯净:经优化的捕获和洗涤方法确保仅洗脱高度富集的PTM肽,而不存在抗体污染;
③ 适合自动化:使用磁珠富集样品,适用于主流的自动化平台,使PTMScan HS试剂盒成为PTM样品自动化制备的理想选择;
④ 方法学可靠:数以百计的文献充分说明PTMScan免疫富集技术的实用性和稳定性。
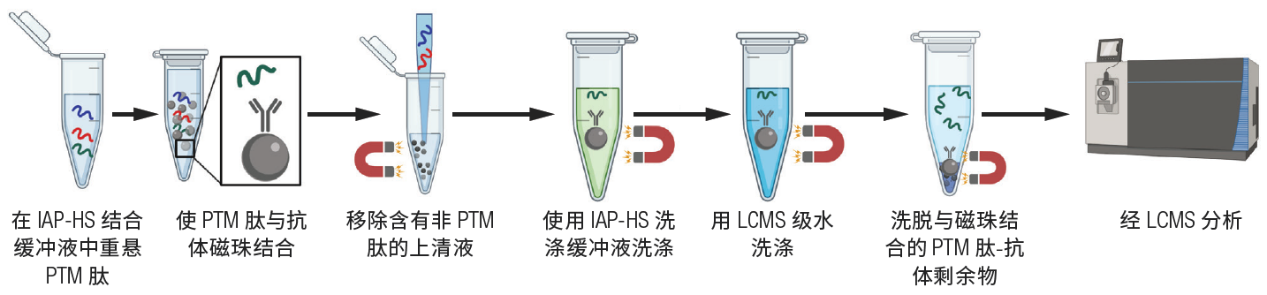 图2:PTMScan HS方法的第3天的流程概况,展示了肽富集和分析过程
图2:PTMScan HS方法的第3天的流程概况,展示了肽富集和分析过程
2. Signal-Seeker™工具
Cytoskeleton公司的Signal-Seeker™试剂盒提供了一种亲和磁珠,用于从细胞或组织裂解液中提取并富集修饰蛋白,然后通过使用目的蛋白的一抗来进行WB实验从而分析富集的蛋白群,最终再使用自身的抗体来检测所需的修饰蛋白。通过使用一套经过严格验证的Signal-Seeker™工具,研究人员无需再开发专门的方法,省去实验方案优化的烦恼,从而极大促进了PTM的发现和后续功能研究。
Signal-Seeker™试剂盒的优势有:
① 试剂盒配套相应的抗体,能对任意蛋白的翻译后修饰进行研究;
② 试剂盒提供了通用的裂解过滤系统和定量工具,能轻松从给定的细胞或组织裂解液中提取蛋白并进行总蛋白的定量;
③ Signal-Seeker™亲和磁珠,富集效果强,配合对照磁珠,提供PTM蛋白精准定量;
④ Signal-Seeker™亲和磁珠可用于捕获PTM修饰的蛋白,然后用于进行进一步的分析(如WB和质谱检测);
⑤ 含有去修饰化抑制剂,防止检测过程中的去修饰;
⑥ 可以用于研究多重PTM的串扰(例如目的蛋白的泛素化和乙酰化);
⑦ 市面上最全面的PTM试剂盒:包括通用裂解过滤系统、蛋白质定量工具、IP浓缩试剂、洗涤试剂、洗脱试剂、化学发光试剂。
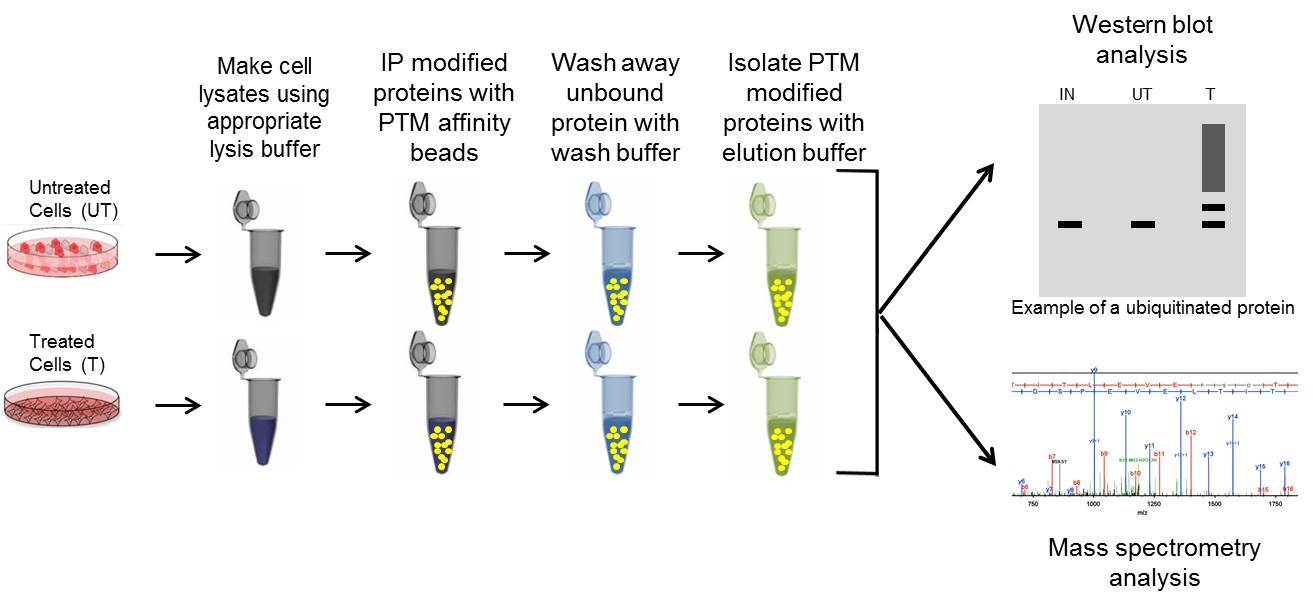 图3:Signal-Seeker™的实验流程
图3:Signal-Seeker™的实验流程
3. PTM查询小工具
PTM研究方法固然重要,但是前期在设计实验方案时了解靶蛋白可能的PTM也是必不可少的,因此小优也整理了一些好用的PTM查询小工具分享给大家:
UniProt:为科学界提供全面、高质量和可免费获取的蛋白质序列和功能信息资源,为任何目标蛋白质提供有关既定 PTM 的信息;
Entrez-Gene:整合了来自众多物种的信息,包括术语、参考序列(RefSeqs)、图谱、途径、变异、表型以及与全球基因组、表型和基因座特定资源的链接;
ModPred:是一种基于序列的蛋白质潜在翻译后修饰 (PTM) 位点预测器,它由 34 个逻辑回归模型组合组成,这些模型分别在一组经实验验证的 126,036 个非冗余位点上进行训练,这些位点来自公共数据库和临时文献检索,涉及 23 种不同的修饰;
Cell:可以从《Cell》系列期刊中查找有关各种 PTM 的最新高影响力文章。
相关产品推荐
参考文献:
1. Johnson H, Eyers CE. Analysis of post-translational modifications by LC-MS/MS. Methods Mol Biol. 2010;658:93-108.
2. Wu R, Haas W, Dephoure N, Huttlin EL, Zhai B, Sowa ME, et al. A large-scale method to measure absolute protein phosphorylation stoichiometries. Nat Methods. 2011;8(8):677-83.
3. Ordureau A, Munch C, Harper JW. Quantifying ubiquitin signaling. Mol Cell. 2015;58(4):660-76.
4. Fuchs SM, Strahl BD. Antibody recognition of histone post-translational modifications: emerging issues and future prospects. Epigenomics. 2011;3(3):247-9.
5. Nordon IM, Brar R, Hinchliffe RJ, Cockerill G, Thompson MM. Proteomics and pitfalls in the search for potential biomarkers of abdominal aortic aneurysms. Vascular. 2010;18(5):264-8.
6. Toby TK, Fornelli L, Kelleher NL. Progress in Top-Down Proteomics and the Analysis of Proteoforms. Annu Rev Anal Chem (Palo Alto Calif). 2016;9(1):499-519.
7. Feist PE, Sun L, Liu X, Dovichi NJ, Hummon AB. Bottom-up proteomic analysis of single HCT 116 colon carcinoma multicellular spheroids. Rapid Commun Mass Spectrom. 2015;29(7):654-8.
8. Glickman JF. Assay Development for Protein Kinase Enzymes. In: Sittampalam GS, Coussens NP, Brimacombe K, Grossman A, Arkin M, Auld D, et al., editors. Assay Guidance Manual. Bethesda (MD)2004.
9. Lim SO, Li CW, Xia W, Cha JH, Chan LC, Wu Y, et al. Deubiquitination and Stabilization of PD-L1 by CSN5. Cancer Cell. 2016.
10. Sarge KD. Analysis of Protein Sumoylation. Curr Protoc Protein Sci. 2016;83:14 8 1-8.
11. Brockly F, Piechaczyk M, Bossis G. Production and Purification of Recombinant SUMOylated Proteins Using Engineered Bacteria. Methods Mol Biol. 2016;1475:55-65.
12. Leuchowius KJ, Weibrecht I, Soderberg O. In situ proximity ligation assay for microscopy and flow cytometry. Curr Protoc Cytom. 2011;Chapter 9:Unit 9 36.
13. Elfineh L, Classon C, Asplund A, Pettersson U, Kamali-Moghaddam M, Lind SB. Tyrosine phosphorylation profiling via in situ proximity ligation assay. BMC Cancer. 2014;14:435.
14. Chen H, Venkat S, McGuire P, Gan Q, Fan C. Recent Development of Genetic Code Expansion for Posttranslational Modification Studies. Molecules. 2018;23(7).
15. Kumar A, Narayanan V, Sekhar A. Characterizing Post-Translational Modifications and Their Effects on Protein Conformation Using NMR Spectroscopy. Biochemistry. 2019.
16. Eskonen V, Tong-Ochoa N, Valtonen S, Kopra K, Harma H. Thermal Dissociation Assay for Time-Resolved Fluorescence Detection of Protein Post-Translational Modifications. ACS Omega. 2019;4(15):16501-7.
17. Restrepo-Perez L, Wong CH, Maglia G, Dekker C, Joo C. Label-Free Detection of Post-translational Modifications with a Nanopore. Nano Lett. 2019;19(11):7957-64.
18. Huang JX, Lee G, Cavanaugh KE, Chang JW, Gardel ML, Moellering RE. High throughput discovery of functional protein modifications by Hotspot Thermal Profiling. Nat Methods. 2019;16(9):894-901.
19. Swatek KN, Usher JL, Kueck AF, Gladkova C, Mevissen TET, Pruneda JN, et al. Insights into ubiquitin chain architecture using Ub-clipping. Nature. 2019;572(7770):533-7.
20. Njeri CW, Ononye OE, Balakrishnan L. Quantification of In Vitro Protein Lysine Acetylation by Reversed Phase HPLC. Methods Mol Biol. 2019;1983:49-56.
上海优宁维生物科技股份有限公司
试剂 | 耗材 | 仪器 | 软件 | 定制 | 实验服务 | 供应链
免费热线:4008-168-068
咨询邮箱:info@univ-bio.com
订购商城:www.univ-bio.com
微信公众平台:优宁维抗体专家,欢迎关注!
小优博士(小程序):5大课堂, 让你的科研不再难!


相关新闻资讯