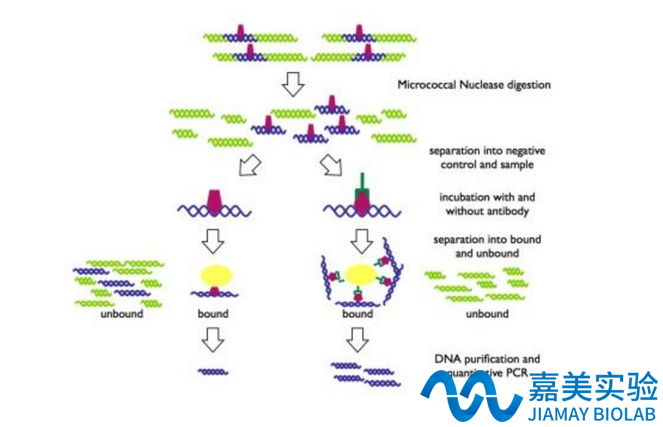
核染色质免疫共沉淀CHIP
Chromatin Immunoprecipitation,ChIP
询价
5 mg
起订
北京 更新日期:2026-02-15
产品详情:
- 中文名称:
- 核染色质免疫共沉淀CHIP
- 英文名称:
- Chromatin Immunoprecipitation,ChIP
- 产品类别:
- 技术服务 免疫生化实验
公司简介
北京嘉美臻元生物技术有限公司(嘉美实验)是一家专注于生命科学领域的高科技企业,公司创立于2005年,由在国内科研试剂、实验技术、质量管理领域有着十几年从业经验的专业、专家团队组建而成;2009年在北京重组,先后成立了北京嘉美诺斯生物科技有限公司(主要开展生物相关产品进、出口业务),北京嘉美纽诺生物科技有限公司(主要为大型药厂、诊断试剂公司提供服务)。为方便实验服务项目开展,成立了北京嘉美臻元生物技术有限公司,以“嘉美实验”品牌提供实验外包服务。近五年来,公司已为全国各类科研用户提供了近5000个大大小小的专项实验外包或整体课题外包项目服务,实验项目涵盖了生命科学、医学的各研究领域。经过多年努力,公司已成为
| 成立日期 | (11年) |
| 注册资本 | 100.000000万人民币 |
| 员工人数 | 50-100人 |
| 年营业额 | ¥ 100万-300万 |
| 经营模式 | 服务 |
| 主营行业 | 生化试剂,细胞培养,微生物学,细胞生物学,免疫安全 |
核染色质免疫共沉淀CHIP相关厂家报价
-

- Human核受体共启动因子3(NCOA3) ELISA Kit
- 上海博尔森生物科技有限公司 VIP
- 2026-02-28
- ¥3104
-
- 核受体共激活因子3(NCOA3)重组蛋白
- 上海泽叶生物科技有限公司 VIP
- 2026-02-28
- ¥2856
-

- 人免疫球蛋白E酶联免疫试剂盒
- 上海康朗生物科技有限公司 VIP
- 2026-02-28
- ¥1820