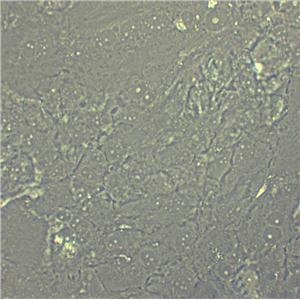
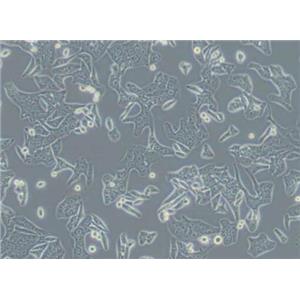
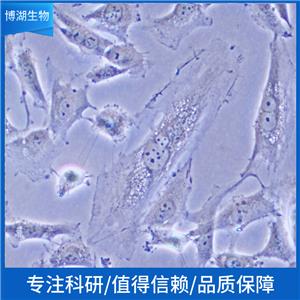
"LoVo人结肠癌复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞复苏后贴壁细胞较少的问题分析:总结1:复苏过程没有问题,是否是从拿出直接放入温水,还有培养箱,二氧化碳浓度,培养基、PH值等环节。要么加GAO浓度FBS 15-20%,看看能否帮助贴壁,当然也需要考虑血清问题,还有确信拿来的细胞没问题。总结2:首先应该怀疑冻存,实际上复苏出问题的可能非常小,因为操作简单,而且死板。1、你冻存的时候是不是消化的时间过长,这是一般人所注意不到的,即使书上也不讲这个问题,太长的消化时间会让细胞复苏时失去贴壁能力,表现为先贴后死,原因是在你复苏的时候细胞已进入凋亡程序,不可逆转的死亡。2、你的冻存HAO不HAO,是什么,甘油还是DMSO,质量非常重要,否则也会死亡。3、你的冻存的量加的是不是太多,AC推荐是不超过7%,大于5%,太多也不HAO。4、你在冻存的时候是不是把DMSO混均匀,这个有一些影响,但不算太大。5、你的冻存是否按部就班,就是所温度梯度是不是把握严格,很多人容易忘却这个事情,因为这个东西流程长。6、如果你细胞污染,你是否能很快看到,我比我的导师能早一天看到污染。从这个角度讲建议去除离心这步。7、你的细胞在冻存前是否过密。还有,不赞成孵箱污染这个概念的,所有在一个孵箱里的细胞都污染一个细菌的话,这个细菌是源于孵箱的,但这不代表孵箱污染,因为孵箱无论你如何处理都有大量的细菌,问题在操作。每次污染的原因都要尽可能的找,以后就不犯同样的问题,这个很重要,不能靠猜,否则你就有可能细胞养绝Zui后换课题,这个见得太多了,别不当会事。
换液周期:每周2-3次
Hs606 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代,2—3天换液一次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:JHH7细胞、HDMEC细胞、Panc 8.13细胞
H716 Cells;背景说明:从一位经5-尿嘧啶治疗的患者腹水中得到的细胞建立了这个细胞株。 与其它结直肠癌细胞系不同,这株细胞有多巴脱羧酶,细胞质中有核心致密的内分泌型颗粒。 这株细胞不表达TAG-72 或CA19-9抗原,也不生成癌胚抗原(CEA);传代方法:1:3—1:6传代,每周换液2—3次;生长特性:悬浮生长,聚团,少数贴壁;形态特性:上皮细胞样;相关产品有:Colo16细胞、BRL 3A细胞、NCI-H1666细胞
SBC-5 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SACC-LM细胞、Soleus clone 8细胞、253J-Bladder-V细胞
LoVo人结肠癌复苏细胞保种中心|带STR证书
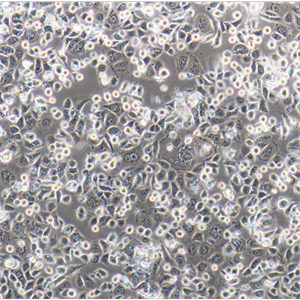
背景信息:LoVo建自1971年,从诊断为结肠腺癌的56岁男性白人的一个转移到左锁骨上区的肿瘤结节建系。癌基因C-myc,K-ras,H-ras,N-ras,Myb,sis和fos的表达呈阳性。癌基因N-myc的表达未做检测。它在裸鼠中能成瘤。与107细胞联合皮下接种5只裸鼠21天后全部成瘤。表达肿瘤特异的核基质蛋白蛋白CC-3和CC-4。
公司细胞系培养稳定、耐活、复苏好,提供人源、鼠源、兔源、猪源等不同组织如肺、气管、支气管、甲状腺、胰腺、垂体、肾上腺、扁桃体、胸腺、肾、膀胱、输尿管、前列腺、心脏、血管等细胞系。全程提供细胞系组织来源、生长特性、细胞形态、背景资料、培养条件、冻存条件、参考文献、运输方式等产品信息及技术服务。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
SUDHL-2 Cells;背景说明:弥漫性大细胞淋巴瘤;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:LP-1细胞、SK-N-BE-2细胞、8226细胞
32D clone 3 Cells;背景说明:骨髓淋巴瘤;C3H/HeJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A875细胞、MDA231-LM2细胞、N-9细胞
IEC6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HEK 293-EBNA细胞、COR-L23细胞、ME-180细胞
A-10 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Co-115细胞、CF-PAC1细胞、SJ-RH30细胞
LoVo人结肠癌复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:上皮细胞样
细胞传代培养(消化法)标准操作规程【原理】细胞在培养瓶长成致密单层后,已基本上饱和,为使细胞能继续生长,同时也将细胞数量扩大,就必须进行传代(再培养)。传代培养也是一种将细胞种保存下去的方法。同时也是利用培养细胞进行各种实验的必经过程。悬浮型细胞直接分瓶就可以,而贴壁细胞需经消化后才能分瓶。【材料和试剂】1)无菌生理缓冲(PBS);2)胰酶-EDA溶(0.05%胰酶-0.53mM EDA-4Na): 以10ml分装于15 ml无菌离心管中,保存于–20℃,使用前放在37℃水槽回温;3)新鲜培养基;4)无菌吸管/离心管/培养瓶【传代前准备操作】1)预热培养用:把已经配制HAO的装有培养、PBS和胰蛋白酶的瓶子放入37℃水浴锅内预热;2)用75%酒精擦拭经过紫外线照射的超净工作台和双手;3)正确摆放使用的器械:保证足够的操作空间,不仅便于操作而且可减少污染;4)点燃酒精灯:注意火焰不能太小;5)准备HAO将要使用的消毒后的空培养瓶;6)取出预热HAO的培养用,用酒精棉球擦拭HAO后方能放入超净台内;7)从培养箱内取出细胞:注意取出细胞时要旋紧瓶盖,用酒精棉球擦拭显微镜的台面,再在镜下观察细胞;8)打开瓶口:将各瓶口一一打开,同时要在酒精灯上烧口消毒。
MDA MB231 Cells;背景说明:MDA-MB-231来自患有转移乳腺腺癌的51岁女病人的胸水。在裸鼠和ALS处理的BALB/c小鼠中,它能形成低分化腺癌(III级)。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:GS-9L细胞、T-ALL1细胞、KMST-6细胞
H-2347 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Anglne细胞、WM-239细胞、TCC Sup细胞
ANA1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:C-127细胞、KBM7细胞、H-2081细胞
HCCLM3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HAEC细胞、Panc_03_27细胞、EB-3细胞
LS-174T Cells;背景说明:LS 174T是LS 180 (ATCC CL 187)结肠腺癌细胞株的胰蛋白酶化变种。 它比亲本更易传代,象LS 180一样生成大量的癌胚抗原(CEA)。 电镜研究表明有丰富的微丝和细胞质粘液素液泡。 直肠抗原3阳性。 p53抗原表达阴性,但mRNA表达阳性。 与ATCC CL-187来源于同一个肿瘤。LS 174T细胞角蛋白染色阳性。 癌基因c-myc, N-myc, H-ras, N-ras, Myb, 和 fos的表达呈阳性。 癌基因k-ras和sis的表达未做检测。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H4-II-EC3细胞、NCIH1869细胞、SKCol1细胞
NCIH295R Cells;背景说明:肾上腺皮质癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TI-73细胞、NCIH1755细胞、NCI-H2591细胞
Anip-973 Cells;背景说明:肺腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:D 407细胞、HR-8348细胞、RKOE6细胞
BEL/FU Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KOSC-2 cl3-43细胞、HBL1细胞、RT-BM细胞
WM-239A Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KYSE 410细胞、BRL-3A细胞、Mel-624细胞
HGC-27 Cells;背景说明:未分化胃癌,能分泌粘液素。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:FLS细胞、H510细胞、Roswell Park Memorial Institute 7951细胞
MDA-MB-436 Cells;背景说明:该细胞源于一名43岁患有乳腺腺癌女性的胸腔积液。;传代方法:1:2传代,每周换液2—3次;生长特性:贴壁生长;形态特性:多角形;相关产品有:CCRF/CEM/0细胞、MRASMC细胞、SCC090细胞
HPBALL Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HBL100细胞、FDCP1细胞、3T3-F442A细胞
SKCO1 Cells;背景说明:该细胞来源于结直肠病人的转移性腹水。;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:PC3M-1E8细胞、NG108-15细胞、RBVEC细胞
H2286 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:椭圆形;相关产品有:KU-812F细胞、MUVEC细胞、SuDHL 8细胞
IM 9 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:3T3 J2细胞、16HBEo-细胞、PANC 203细胞
P3X63 AG 8.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C33细胞、P-2003细胞、PANC-10-05细胞
P3-NS1/1Ag 4.1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SIRC细胞、VERO C 1008细胞、H-2122细胞
LoVo人结肠癌复苏细胞保种中心|带STR证书
24131 Cells(提供STR鉴定图谱)
Abcam HeLa PORCN KO Cells(提供STR鉴定图谱)
AG24289 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRH247 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XH291 Cells(提供STR鉴定图谱)
C11STH Cells(提供STR鉴定图谱)
D21-iPSC#2 Cells(提供STR鉴定图谱)
F2408-No.20 Cells(提供STR鉴定图谱)
GM07522 Cells(提供STR鉴定图谱)
OVCAR.8 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-446细胞、HCM细胞、IPLB-Sf21AE细胞
B16 Cells;背景说明:该细胞源于C57BL/6J小鼠黑色素瘤,可以产生黑色素,同基因小鼠体内移植可成瘤;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:梭形;相关产品有:Panc-08.13细胞、CCD33Co细胞、Panc-8_13细胞
Hs 729 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:SK Hep1细胞、YES-2细胞、JB-6 Cl 30细胞
MDAMB435S Cells;背景说明:MDA-MB-435S是一种纺锤形的细胞,1976年由其亲本(435)中筛选得到。435是从31岁的转移性乳腺导管腺癌女性患者胸水中分离得到。当用荧光染料对微管蛋白进行染色时亲本细胞显现散布特征(II型)。最近通过cDNA阵列研究表明,亲本(MDA-MB-435)可归入黑素瘤起源。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:纺锤形;相关产品有:Caco-2/BBe 1细胞、TOV21细胞、NCI-SNU-520细胞
IHH4 Cells;背景说明:甲状腺乳头状癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCTC929细胞、NCI-H1836细胞、VP267细胞
Roswell Park Memorial Institute 1788 Cells;背景说明:B淋巴细胞;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:EM3细胞、SGC7901/DDP细胞、C3H10T1/2细胞
B/C3T3.We Cells(提供STR鉴定图谱)
Detroit 562 Cells;背景说明:器官:咽头 疾病:癌 取材转移灶:胸水;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SUDHL6细胞、PaTu 8988s细胞、Hs746T细胞
UO-31 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Human Kidney-2细胞、PC2细胞、MC-3T3细胞
WEHI-164 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HSC 3细胞、3T3(A31)细胞、PC-3M-1E8细胞
Ontario Cancer Institute-Acute Myeloid Leukemia-5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:H-676B细胞、Centre Antoine Lacassagne-148细胞、RIN-m 14B细胞
LoMeT-ccRcc Cells;背景说明:肾透明细胞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RASMCs细胞、EoL-1细胞、OVCAR.5细胞
EFM192A Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:X63-Ag8-653细胞、HSC(Human Schwann)细胞、SU-DHL-8细胞
EFM192A Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:X63-Ag8-653细胞、HSC(Human Schwann)细胞、SU-DHL-8细胞
WEHI231 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LICR-HN6细胞、McA-RH 7777细胞、VMRC-RCZ细胞
HAP1 ARRB1 (-) 2 Cells(提供STR鉴定图谱)
HAP1 SLC36A4 (-) 2 Cells(提供STR鉴定图谱)
HCC-1806 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:D324 Med细胞、RBL 2H3细胞、HCS2/8细胞
VMRC-LCD Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:M059K细胞、NBL-S细胞、NCI-H1522细胞
LTEP-a2 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:S37细胞、Intestinal Porcine Epithelial Cell line-1细胞、WRL68细胞
Biologics Standards-Cercopithecus-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-810细胞、KAT-5细胞、MB39细胞
J774 Cells;背景说明:巨噬细胞;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LuCL4细胞、SKM1细胞、H-2066细胞
REC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,2-3天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NCI-H-82细胞、MDA-MB 361细胞、SY5Y细胞
SK-N-AS Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:FDCP-1细胞、NCI N87细胞、CCRF细胞
H766T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:8传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Hs27细胞、EAHY-926细胞、Y3Ag1.2.3细胞
IBEC line B Cells(提供STR鉴定图谱)
LIM2408 Cells(提供STR鉴定图谱)
NCC-GCTB8-C1 Cells(提供STR鉴定图谱)
PathHunter CHO-K1 AVPR2 beta-arrestin Cells(提供STR鉴定图谱)
S008885 Cells(提供STR鉴定图谱)
Ubigene HCT 116 PLCB1 KO Cells(提供STR鉴定图谱)
WAe001-A-F Cells(提供STR鉴定图谱)
HCORDi001-C Cells(提供STR鉴定图谱)
Ramos (RA) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BALB/3T3 clone A31细胞、H19-7细胞、Nb-2细胞
NCI-H676B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:J82细胞、CPAE细胞、A-427细胞
SKM1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:QBC-939细胞、KBM-5细胞、NCIH647细胞
CATH.a Cells;背景说明:神经;SV40转化;C57BL/6 x DBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ECC 12细胞、NCIADRRES细胞、SUP-M2细胞
D341 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长;形态特性:髓母细胞样;相关产品有:hFOB1.19细胞、CAL-120细胞、KASUMI6细胞
HCT GEO Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ISHI细胞、Rainbow Trout Embryo细胞、T_T_细胞
H322 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:P3-x63-Ag8 653细胞、Jiyoye细胞、293F细胞
CDC/EU.HMEC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NCIH2227细胞、SKN-SH细胞、NCI-H209细胞
BEL7405 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:GDM1细胞、MD Anderson-Metastatic Breast-361细胞、CAL33细胞
MCF-7B Cells;背景说明:浸润性导管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jurkat E6.1细胞、TFK-1细胞、NCIH1703细胞
HcerEpic Cells;背景说明:子宫颈;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-596细胞、Hepa1-6细胞、hUC-MSC细胞
HCT/Taxo1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NTERA2-D1细胞、RKN细胞、NW-MEL-38细胞
M-1 myeloid leukemia Cells;背景说明:髓系白血病;SL;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI H69细胞、H69/P细胞、RAT2细胞
H1155 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:HM细胞、C32 [Human melanoma]细胞、NCIH526细胞
HUVEC Cells;背景说明:脐静脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:QG-56细胞、HCC0078细胞、CL1-5细胞
TIG-3-50 Cells(提供STR鉴定图谱)
CAL12T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L6细胞、SEG1细胞、NCI H747细胞
OCI-Ly1 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:THPI细胞、Mahlavu细胞、HCC1500细胞
rHSC-99 Cells;背景说明:肝星状 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:624细胞、Onda 11细胞、Panc327细胞
NCI-SNU-C2A Cells;背景说明:详见相关文献介绍;传代方法:每周两次换液;生长特性:松散附着、多单元的聚合;形态特性:上皮细胞样;相关产品有:SU 86.86细胞、TE4细胞、TE4细胞
GTL16 Cells;背景说明:胃癌;肝转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1184细胞、H-1341细胞、MBVP细胞
hTERT RPE-1 Cells;背景说明:视网膜色素上皮;hTERT永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:QGY-7703细胞、CCRFCEM细胞、G519细胞
HEL-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SCC 4细胞、Adeno-293细胞、H2052细胞
CFSC-8B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L-6 myoblast细胞、HCAEC细胞、M20 [Human melanoma]细胞
E6-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FRhK-4细胞、293 c18细胞、143 B细胞
GM03569 Cells;背景说明:该细胞是由绵羊红细胞免疫的BALB/c小鼠脾细胞和P3X63Ag8骨髓瘤细胞融合得到的。该细胞不分泌免疫球蛋白,对20μg/ml的8-氮鸟嘌呤有抗性,对HAT比较敏感;该细胞可以作为细胞融合时的B细胞组分用于制备杂交瘤;鼠痘病毒阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:UCLA NPA-87-1细胞、BERH-2细胞、MC3T3-E1细胞
NCIH1915 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:SW260细胞、K7M2 wt细胞、HBSMC细胞
IMR-90 Cells;背景说明:W.W. Nichols及其同事从一位16周女婴的肺中取材,建立了人二倍体成纤维细胞株IMR-90。分裂潜能,病毒感受性和其它性质都得到了充分研究,因而这株细胞可以作为WI-38或其它标准人肺细胞株的替代株。有报道称这株细胞在表现出衰老前可倍增58次。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:LC1细胞、SCC-15细胞、NCI-H2052细胞
SMMC7721 Cells;背景说明:用Northernblot方法,未能检测到细胞中1.3kbLFIRE-1/HFREP-1mRNA的表达。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CCD19Lu细胞、SU-DHL-1细胞、Pan02细胞
87MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L1210细胞、675T细胞、HUT-226细胞
LoVo人结肠癌复苏细胞保种中心|带STR证书
BayGenomics ES cell line CSE075 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRT224 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC366 Cells(提供STR鉴定图谱)
J26-B7 Cells(提供STR鉴定图谱)
PCRP-TFAP2C-1B9 Cells(提供STR鉴定图谱)
LO-Tmat Cells(提供STR鉴定图谱)
" "PubMed=8464898; DOI=10.1073/pnas.90.7.2842; PMCID=PMC46192
Browning M.J., Krausa P., Rowan A.J., Bicknell D.C., Bodmer J.G., Bodmer W.F.
Tissue typing the HLA-A locus from genomic DNA by sequence-specific PCR: comparison of HLA genotype and surface expression on colorectal tumor cell lines.
Proc. Natl. Acad. Sci. U.S.A. 90:2842-2845(1993)
PubMed=8197130; DOI=10.1073/pnas.91.11.4751; PMCID=PMC43866
Bicknell D.C., Rowan A.J., Bodmer W.F.
Beta 2-microglobulin gene mutations: a study of established colorectal cell lines and fresh tumors.
Proc. Natl. Acad. Sci. U.S.A. 91:4751-4755(1994)
PubMed=7651727
Kastrinakis W.V., Ramchurren N., Rieger K.M., Hess D.T., Loda M., Steele G., Summerhayes I.C.
Increased incidence of p53 mutations is associated with hepatic metastasis in colorectal neoplastic progression.
Oncogene 11:647-652(1995)
PubMed=9000147
Cottu P.-H., Muzeau F., Estreicher A., Flejou J.-F., Iggo R.D., Thomas G., Hamelin R.
Inverse correlation between RER+ status and p53 mutation in colorectal cancer cell lines.
Oncogene 13:2727-2730(1996)
PubMed=9000572
Hoang J.-M., Cottu P.-H., Thuille B., Salmon R.J., Thomas G., Hamelin R.
BAT-26, an indicator of the replication error phenotype in colorectal cancers and cell lines.
Cancer Res. 57:300-303(1997)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::AID-MC5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=9294210; DOI=10.1073/pnas.94.19.10330; PMCID=PMC23362
Ilyas M., Tomlinson I.P.M., Rowan A.J., Pignatelli M., Bodmer W.F.
Beta-catenin mutations in cell lines established from human colorectal cancers.
Proc. Natl. Acad. Sci. U.S.A. 94:10330-10334(1997)
PubMed=9515795
Sparks A.B., Morin P.J., Vogelstein B., Kinzler K.W.
Mutational analysis of the APC/beta-catenin/Tcf pathway in colorectal cancer.
Cancer Res. 58:1130-1134(1998)
PubMed=9715273; DOI=10.1038/sj.onc.1201986
Eshleman J.R., Casey G., Kochera M.E., Sedwick W.D., Swinler S.E., Veigl M.L., Willson J.K.V., Schwartz S., Markowitz S.D.
Chromosome number and structure both are markedly stable in RER colorectal cancers and are not destabilized by mutation of p53.
Oncogene 17:719-725(1998)
PubMed=10674020; DOI=10.1016/S0959-8049(99)00206-3
Ku J.-L., Yoon K.-A., Kim D.-Y., Park J.-G.
Mutations in hMSH6 alone are not sufficient to cause the microsatellite instability in colorectal cancer cell lines.
Eur. J. Cancer 35:1724-1729(1999)
PubMed=10612807; DOI=10.1002/(SICI)1098-2264(200002)27:2<183::AID-GCC10>3.0.CO;2-P; PMCID=PMC4721570
Ghadimi B.M., Sackett D.L., Difilippantonio M.J., Schrock E., Neumann T., Jauho A., Auer G., Ried T.
Centrosome amplification and instability occurs exclusively in aneuploid, but not in diploid colorectal cancer cell lines, and correlates with numerical chromosomal aberrations.
Genes Chromosomes Cancer 27:183-190(2000)
PubMed=10737795; DOI=10.1073/pnas.97.7.3352; PMCID=PMC16243
Rowan A.J., Lamlum H., Ilyas M., Wheeler J.M.D., Straub J., Papadopoulou A., Bicknell D.C., Bodmer W.F., Tomlinson I.P.M.
APC mutations in sporadic colorectal tumors: a mutational 'hotspot' and interdependence of the 'two hits'.
Proc. Natl. Acad. Sci. U.S.A. 97:3352-3357(2000)
PubMed=11226274; DOI=10.1073/pnas.041603298; PMCID=PMC30173
Abdel-Rahman W.M., Katsura K., Rens W., Gorman P.A., Sheer D., Bicknell D.C., Bodmer W.F., Arends M.J., Wyllie A.H., Edwards P.A.W.
Spectral karyotyping suggests additional subsets of colorectal cancers characterized by pattern of chromosome rearrangement.
Proc. Natl. Acad. Sci. U.S.A. 98:2538-2543(2001)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=11526487; DOI=10.1038/sj.onc.1204611
Gayet J., Zhou X.-P., Duval A., Rolland S., Hoang J.-M., Cottu P.-H., Hamelin R.
Extensive characterization of genetic alterations in a series of human colorectal cancer cell lines.
Oncogene 20:5025-5032(2001)
PubMed=11668190; DOI=10.1177/002215540104901105
Quentmeier H., Osborn M., Reinhardt J., Zaborski M., Drexler H.G.
Immunocytochemical analysis of cell lines derived from solid tumors.
J. Histochem. Cytochem. 49:1369-1378(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=12584437; DOI=10.1159/000068544
Melcher R., Koehler S., Steinlein C., Schmid M., Mueller C.R., Luehrs H., Menzel T., Scheppach W., Moerk H., Scheurlen M., Koehrle J., Al-Taie O.
Spectral karyotype analysis of colon cancer cell lines of the tumor suppressor and mutator pathway.
Cytogenet. Genome Res. 98:22-28(2002)
PubMed=12615714
Hempen P.M., Zhang L., Bansal R.K., Iacobuzio-Donahue C.A., Murphy K.M., Maitra A., Vogelstein B., Whitehead R.H., Markowitz S.D., Willson J.K.V., Yeo C.J., Hruban R.H., Kern S.E.
Evidence of selection for clones having genetic inactivation of the activin A type II receptor (ACVR2) gene in gastrointestinal cancers.
Cancer Res. 63:994-999(2003)
PubMed=12661003; DOI=10.1002/gcc.10196
Seitz S., Wassmuth P., Plaschke J., Schackert H.K., Karsten U., Santibanez-Koref M.F., Schlag P.M., Scherneck S.
Identification of microsatellite instability and mismatch repair gene mutations in breast cancer cell lines.
Genes Chromosomes Cancer 37:29-35(2003)
PubMed=15771911; DOI=10.1016/j.cancergencyto.2004.08.023
Melcher R., Maisch S., Koehler S., Bauer M., Steinlein C., Schmid M., Kudlich T., Schauber J., Luehrs H., Menzel T., Scheppach W.
SKY and genetic fingerprinting reveal a cross-contamination of the putative normal colon epithelial cell line NCOL-1.
Cancer Genet. Cytogenet. 158:84-87(2005)
PubMed=15900046; DOI=10.1093/jnci/dji133
Mashima T., Oh-hara T., Sato S., Mochizuki M., Sugimoto Y., Yamazaki K., Hamada J.-i., Tada M., Moriuchi T., Ishikawa Y., Kato Y., Tomoda H., Yamori T., Tsuruo T.
p53-defective tumors with a functional apoptosome-mediated pathway: a new therapeutic target.
J. Natl. Cancer Inst. 97:765-777(2005)
PubMed=16418264; DOI=10.1073/pnas.0510146103; PMCID=PMC1327731
Liu Y., Bodmer W.F.
Analysis of p53 mutations and their expression in 56 colorectal cancer cell lines.
Proc. Natl. Acad. Sci. U.S.A. 103:976-981(2006)
PubMed=16854228; DOI=10.1186/1476-4598-5-29; PMCID=PMC1550420
Bandres Elizalde E.M., Cubedo E., Agirre X., Malumbres R., Zarate R., Ramirez N., Abajo A., Navarro A., Moreno I., Monzo M., Garcia-Foncillas J.
Identification by real-time PCR of 13 mature microRNAs differentially expressed in colorectal cancer and non-tumoral tissues.
Mol. Cancer 5:29.1-29.10(2006)
PubMed=18258742; DOI=10.1073/pnas.0712176105; PMCID=PMC2268141
Emaduddin M., Bicknell D.C., Bodmer W.F., Feller S.M.
Cell growth, global phosphotyrosine elevation, and c-Met phosphorylation through Src family kinases in colorectal cancer cells.
Proc. Natl. Acad. Sci. U.S.A. 105:2358-2362(2008)
PubMed=19927377; DOI=10.1002/gcc.20730; PMCID=PMC2818350
Knutsen T., Padilla-Nash H.M., Wangsa D., Barenboim-Stapleton L., Camps J., McNeil N.E., Difilippantonio M.J., Ried T.
Definitive molecular cytogenetic characterization of 15 colorectal cancer cell lines.
Genes Chromosomes Cancer 49:204-223(2010)
PubMed=19941903; DOI=10.1016/j.jviromet.2009.11.022
Karger A., Bettin B., Lenk M., Mettenleiter T.C.
Rapid characterisation of cell cultures by matrix-assisted laser desorption/ionisation mass spectrometric typing.
J. Virol. Methods 164:116-121(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20570890; DOI=10.1158/0008-5472.CAN-10-0192; PMCID=PMC2943514
Janakiraman M., Vakiani E., Zeng Z.-S., Pratilas C.A., Taylor B.S., Chitale D., Halilovic E., Wilson M., Huberman K., Ricarte Filho J.C.M., Persaud Y., Levine D.A., Fagin J.A., Jhanwar S.C., Mariadason J.M., Lash A., Ladanyi M., Saltz L.B., Heguy A., Paty P.B., Solit D.B.
Genomic and biological characterization of exon 4 KRAS mutations in human cancer.
Cancer Res. 70:5901-5911(2010)
PubMed=20606684; DOI=10.1038/sj.bjc.6605780; PMCID=PMC2920028
Bracht K., Nicholls A.M., Liu Y., Bodmer W.F.
5-fluorouracil response in a large panel of colorectal cancer cell lines is associated with mismatch repair deficiency.
Br. J. Cancer 103:340-346(2010)
PubMed=20831567; DOI=10.1111/j.1582-4934.2010.01170.x; PMCID=PMC3918049
Ma Y.-L., Zhang P., Wang F., Moyer M.P., Yang J.-J., Liu Z.-H., Peng J.-Y., Chen H.-Q., Zhou Y.-K., Liu W.-J., Qin H.-L.
Human embryonic stem cells and metastatic colorectal cancer cells shared the common endogenous human microRNA-26b.
J. Cell. Mol. Med. 15:1941-1954(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=24042735; DOI=10.1038/oncsis.2013.35; PMCID=PMC3816225
Ahmed D., Eide P.W., Eilertsen I.A., Danielsen S.A., Eknaes M., Hektoen M., Lind G.E., Lothe R.A.
Epigenetic and genetic features of 24 colon cancer cell lines.
Oncogenesis 2:e71.1-e71.8(2013)
PubMed=24755471; DOI=10.1158/0008-5472.CAN-14-0013
Mouradov D., Sloggett C., Jorissen R.N., Love C.G., Li S., Burgess A.W., Arango D., Strausberg R.L., Buchanan D., Wormald S., O'Connor L., Wilding J.L., Bicknell D.C., Tomlinson I.P.M., Bodmer W.F., Mariadason J.M., Sieber O.M.
Colorectal cancer cell lines are representative models of the main molecular subtypes of primary cancer.
Cancer Res. 74:3238-3247(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25926053; DOI=10.1038/ncomms8002
Medico E., Russo M., Picco G., Cancelliere C., Valtorta E., Corti G., Buscarino M., Isella C., Lamba S., Martinoglio B., Veronese S., Siena S., Sartore-Bianchi A., Beccuti M., Mottolese M., Linnebacher M., Cordero F., Di Nicolantonio F., Bardelli A.
The molecular landscape of colorectal cancer cell lines unveils clinically actionable kinase targets.
Nat. Commun. 6:7002.1-7002.10(2015)
PubMed=25944804; DOI=10.1158/1078-0432.CCR-14-2457
Bazzocco S., Dopeso H., Carton-Garcia F., Macaya I., Andretta E., Chionh F., Rodrigues P., Garrido M., Alazzouzi H., Nieto R., Sanchez A., Schwartz S. Jr., Bilic J., Mariadason J.M., Arango D.
Highly expressed genes in rapidly proliferating tumor cells as new targets for colorectal cancer treatment.
Clin. Cancer Res. 21:3695-3704(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26537799; DOI=10.1074/mcp.M115.051235; PMCID=PMC4762531
Holst S., Deuss A.J.M., van Pelt G.W., van Vliet S.J., Garcia-Vallejo J.J., Koeleman C.A.M., Deelder A.M., Mesker W.E., Tollenaar R.A.E.M., Rombouts Y., Wuhrer M.
N-glycosylation profiling of colorectal cancer cell lines reveals association of fucosylation with differentiation and caudal type homebox 1 (CDX1)/villin mRNA expression.
Mol. Cell. Proteomics 15:124-140(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28683746; DOI=10.1186/s12943-017-0691-y; PMCID=PMC5498998
Berg K.C.G., Eide P.W., Eilertsen I.A., Johannessen B., Bruun J., Danielsen S.A., Bjornslett M., Meza-Zepeda L.A., Eknaes M., Lind G.E., Myklebost O., Skotheim R.I., Sveen A., Lothe R.A.
Multi-omics of 34 colorectal cancer cell lines -- a resource for biomedical studies.
Mol. Cancer 16:116.1-116.16(2017)
PubMed=28854368; DOI=10.1016/j.celrep.2017.08.010; PMCID=PMC5583477
Roumeliotis T.I., Williams S.P., Goncalves E., Alsinet C., Del Castillo Velasco-Herrera M., Aben N., Ghavidel F.Z., Michaut M., Schubert M., Price S., Wright J.C., Yu L., Yang M., Dienstmann R., Guinney J.H., Beltrao P., Brazma A., Pardo M., Stegle O., Adams D.J., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Choudhary J.S.
Genomic determinants of protein abundance variation in colorectal cancer cells.
Cell Rep. 20:2201-2214(2017)
PubMed=29101300; DOI=10.15252/msb.20177701; PMCID=PMC5731344
Frejno M., Zenezini Chiozzi R., Wilhelm M., Koch H., Zheng R.-S., Klaeger S., Ruprecht B., Meng C., Kramer K., Jarzab A., Heinzlmeir S., Johnstone E., Domingo E., Kerr D.J., Jesinghaus M., Slotta-Huspenina J., Weichert W., Knapp S., Feller S.M., Kuster B.
Pharmacoproteomic characterisation of human colon and rectal cancer.
Mol. Syst. Biol. 13:951-951(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"