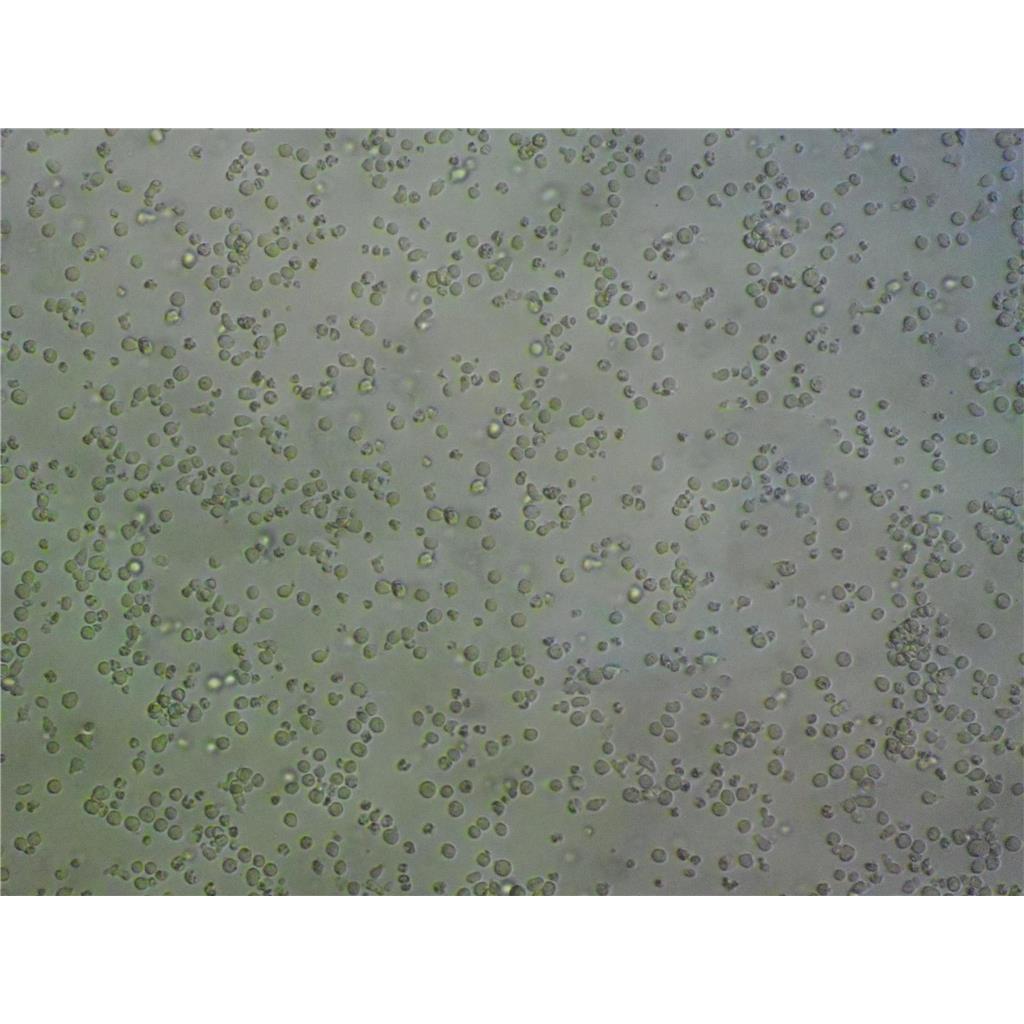
"JeKo-1人套细胞淋巴瘤复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:悬浮生长
正确的细胞复苏需知事项:细胞冻存HAO了,接下来要注意什么问题呢?没错,就是记得到时间了,拿出来复苏。那么,细胞复苏的过程中又有哪些该注意的事项呢?细胞活力和形态检查的作用何在?活力检查——千万不要使用不健康的细胞,可能有污染(真菌、支原体等),如果发现有污染毫不犹豫的丢弃!形态检查——检查细胞的固有形态和生长行为。冻存细胞:补充新的培养——在您开始冻存细胞的前一天补充新的培养。在细胞长至70%单层时收获细胞,计数活细胞数,用冻存调整细胞密度~5 x106 s/ml (根据不同的细胞类型调整);冻存——用冻存洗细胞并用冻存重悬细胞,有不同类型的冻存,根据细胞类型选择Zui合适的冻存(常用的冻存成分有):5-10% DMSO——注意确保DMSO不含有其他的毒性物质;5-15%甘油;如果细胞在无血清培养基内生长,应在50%条件培养基内(细胞在无血清培养基内生长24小时)内冻存和复苏。在冻存管上标记HAO细胞类型,日期,冻存人等信息,并保证每冻存管不超过1.5ml。放入罐之前记录冻存管的数量和位置。以Zui快的速度转移冻存管知罐内,因此,此步骤ZuiHAO使用干冰,或者把冻存管浸入装有的小盒内。此外还要注意,在冻存管上没有足够的空间记录细胞的详细信息,做HAO记录是非常非常重要的!还有一个Zui重要的,一定要在异地的罐内保存同样的一份细胞,以免其中的一个罐出现问题!细胞正确的复苏方式和正确的冻存方式同样重要,熟记以下要点:当从罐内取出细胞时,有可能会出现冻存管破裂的情况,使用保护面罩和防护服十分必要;其实,细胞复苏只是一个简单的实验,不过这其中却不可避免有一些需要注意的细节,不然,也不一定会尽如人意。例如说,人身健康方面:一定要记得做HAO防冻工作,戴上护目镜;尽量降低DMSO对细胞的损伤等等。
换液周期:每周2-3次
KP4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NS653细胞、F9细胞、VP229细胞
SNU761 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH1694细胞、H1755细胞、Bac 1.2F5细胞
HPAEpiC Cells;背景说明:肺泡;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Colon-38细胞、IMR-90细胞、hRPTC细胞
JeKo-1人套细胞淋巴瘤复苏细胞保种中心|带STR证书
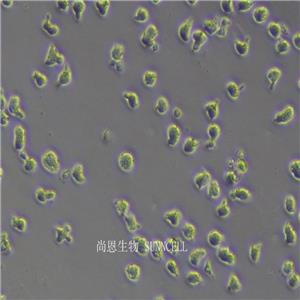
背景信息:一位套细胞淋巴瘤患者的巨细胞变种显示白血病转变,从其外周血单核细胞出发建立了MCL细胞株JeKo-1。 JeKo-1细胞EB病毒阴性,并表达一种B细胞表型的IgM。 细胞过表达cyclin D1, Bcl-2, c-Myc 及 Rb 蛋白。 Bcl-1/J(H)基因重排得到了PCR证实。 JeKo-1细胞在SCID小鼠中高成瘤。 [PubMed: 9753063]
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
CAL 39 Cells;背景说明:外阴鳞癌细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MM1S细胞、NCI-H1522细胞、MMAc.SF细胞
A3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SF-763细胞、X63-Ag8-653细胞、NPA87细胞
Hs27 Cells;背景说明:包皮;成纤维细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:K299细胞、OS-732细胞、HCC-44细胞
HCC1954 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:偶尔上皮细胞空泡;形态特性:上皮细胞样;相关产品有:MFE280细胞、A549-Taxol细胞、SKO3细胞
JeKo-1人套细胞淋巴瘤复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:淋巴母细胞样
细胞传代培养(消化法)标准操作规程【原理】细胞在培养瓶长成致密单层后,已基本上饱和,为使细胞能继续生长,同时也将细胞数量扩大,就必须进行传代(再培养)。传代培养也是一种将细胞种保存下去的方法。同时也是利用培养细胞进行各种实验的必经过程。悬浮型细胞直接分瓶就可以,而贴壁细胞需经消化后才能分瓶。【材料和试剂】1)无菌生理缓冲(PBS);2)胰酶-EDA溶(0.05%胰酶-0.53mM EDA-4Na): 以10ml分装于15 ml无菌离心管中,保存于–20℃,使用前放在37℃水槽回温;3)新鲜培养基;4)无菌吸管/离心管/培养瓶【传代前准备操作】1)预热培养用:把已经配制HAO的装有培养、PBS和胰蛋白酶的瓶子放入37℃水浴锅内预热;2)用75%酒精擦拭经过紫外线照射的超净工作台和双手;3)正确摆放使用的器械:保证足够的操作空间,不仅便于操作而且可减少污染;4)点燃酒精灯:注意火焰不能太小;5)准备HAO将要使用的消毒后的空培养瓶;6)取出预热HAO的培养用,用酒精棉球擦拭HAO后方能放入超净台内;7)从培养箱内取出细胞:注意取出细胞时要旋紧瓶盖,用酒精棉球擦拭显微镜的台面,再在镜下观察细胞;8)打开瓶口:将各瓶口一一打开,同时要在酒精灯上烧口消毒。
LS123 Cells;背景说明:详见相关文献介绍;传代方法:1:4—1:8传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HS-68细胞、A-172 MG细胞、HSMC细胞
NCI-H596 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MDCK supertube细胞、Vero C1008细胞、Los Angeles Prostate Cancer-4细胞
NCI-HUT-125 Cells;背景说明:腺鳞状肺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Ramos.2G6.4C10细胞、Jurkat E6细胞、C33细胞
QG-56 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SY5Y细胞、Tu-686细胞、SB细胞
Hep-G2/C3A Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:alpha TC1-6细胞、BJA-B-1细胞、aTC1-6细胞
MPC-83 Cells;背景说明:胰腺;腺泡 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PL9细胞、MC 116细胞、H1355细胞
HCC9724 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BIU87细胞、MRAEC细胞、T-47D细胞
SF-767 Cells;背景说明:脑瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU251细胞、IOSE-Mar细胞、CT 26细胞
MA-782 Cells;背景说明:乳腺癌;Balb/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NKT细胞、AR41P细胞、UCLA SO M14细胞
SUM 102 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Swiss-3T3细胞、ID8/MOSEC细胞、NFS60细胞
CAKI 1 Cells;背景说明:该细胞超微结构中包含许多微绒毛、少许微丝、许多小线粒体、发达的高尔基休和内质网、许多脂滴和多层体、次级溶酶体,没有发现病毒颗粒。;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCI Ly10细胞、MILE SVEN1细胞、RC-4B/C细胞
COLO-320HSR Cells;背景说明:该细胞1984年建系,源自一位33岁患有大肠腺癌男性经5-fu治疗后的腹水。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:详见产品说明书;相关产品有:UACC-812细胞、G-401细胞、NIH3T3细胞
KMB 17 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:MCF10A细胞、COV362细胞、SNU216细胞
E.G7-OVA Cells;背景说明:详见相关文献介绍;传代方法:细胞密度保持在1×105—1×106 cells/ml,每周换液2—3次 ;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:EOC20细胞、CAKI 1细胞、AD-293细胞
SCC25 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Hs-940细胞、Intestinal Epithelioid Cell line No. 6细胞、P3-Jiyoye细胞
HUC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NCI-H187细胞、Astrocyte type I clone细胞、TE-12细胞
H-292 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:WEHI164细胞、PC3M细胞、NCI-H-128细胞
JeKo-1人套细胞淋巴瘤复苏细胞保种中心|带STR证书
Abcam HCT 116 CARD11 KO Cells(提供STR鉴定图谱)
AG04553 Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSI182 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RST408 Cells(提供STR鉴定图谱)
BG04 Cells(提供STR鉴定图谱)
CHO421PV Cells(提供STR鉴定图谱)
DA03048 Cells(提供STR鉴定图谱)
DD0060 Cells(提供STR鉴定图谱)
GM01635 Cells(提供STR鉴定图谱)
SU86.86 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NT2D1细胞、BIC细胞、H-1435细胞
2T Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK-293A细胞、rUCMSCs细胞、NCM460细胞
NCI747 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:N87细胞、KYSE0520细胞、NCI-H735细胞
HuT102 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:圆形;淋巴母细胞样;相关产品有:PLA-801D细胞、RPMI-8226细胞、NCl-H157细胞
LC-1 sq Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CEM/C1细胞、NCI-H1618细胞、Bac1 2F5细胞
SK-MEL-24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:HL 60细胞、EM-3细胞、NuTu 19细胞
3C8 [Mouse hybridoma against human RB1] Cells(提供STR鉴定图谱)
GM3569 Cells;背景说明:该细胞是由绵羊红细胞免疫的BALB/c小鼠脾细胞和P3X63Ag8骨髓瘤细胞融合得到的。该细胞不分泌免疫球蛋白,对20μg/ml的8-氮鸟嘌呤有抗性,对HAT比较敏感;该细胞可以作为细胞融合时的B细胞组分用于制备杂交瘤;鼠痘病毒阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:P3.NS-1/1.Ag4.1细胞、MDA-MB361细胞、KNS-81细胞
M-20 Cells;背景说明:黑色素瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCCC9810细胞、CAL148细胞、MUVEC细胞
COV-362 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:BAEC细胞、MKN45细胞、PLB 985细胞
B16 BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KOSC2细胞、HeLa/S3细胞、Jurkat-77细胞
KG1 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:原粒细胞;相关产品有:BE2M17细胞、U-87MG ATCC细胞、Hs 766T细胞
RPTEC TERT1 Cells;背景说明:肾;近端小管上皮;HGNC-TERT转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:786O细胞、H2591细胞、CF PAC-1细胞
BE2-C Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCT/FU细胞、U-2-OS细胞、NCI-841细胞
MARC 145 Cells;背景说明:胚肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CoCL2细胞、CP70细胞、T-T细胞
GM21829 Cells(提供STR鉴定图谱)
HAP1 MAPK8 (-) 2 Cells(提供STR鉴定图谱)
SNU-C1 Cells;背景说明:详见相关文献介绍;传代方法:每3-5天换液。;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:NGP细胞、GLAG-66细胞、Ly7细胞
HBL 100 Cells;背景说明:该细胞由E.V.Gaffney及其同事从一位没有乳癌家族史的供者乳汁中建立,培养出来的细胞染色体组型在第7代时就不正常;电镜照片显示有微丝、张力原纤维和桥粒;Southern转移表明有整合型SV40病毒基因,当作正常细胞。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:RKOE6细胞、HCT 8细胞、ZR751细胞
BCECs Cells;背景说明:脑;毛细血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PANC403细胞、MCF-7/ADR-RES细胞、MDA-MB 468细胞
MCF-12A Cells;背景说明:非致瘤性乳腺上皮细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LSC-1细胞、SKHEP-1细胞、EB-2细胞
HDSMC Cells;背景说明:表皮血管平滑肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LTEPsm细胞、MHH-CALL-2细胞、Kit 225-K6细胞
MUVEC Cells;背景说明:脐静脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SCC-25细胞、MOLT-3细胞、SNB19细胞
RCC 7860 Cells;背景说明:该细胞源自一位原发性肾透明细胞癌患者。该细胞有微绒毛和桥粒,能在软琼脂上生长。此细胞生成一种PTH(甲状旁腺素)样的多肽,与乳癌和肺癌中生成的肽相似,其N端序列与PTH相似,具有PTH样活性,分子量为6000D。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:COLO-205细胞、WEHI231细胞、CAKI 2细胞
H647ell Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:SMMC-7721细胞、C3H10T1/2细胞、OCIAML4细胞
HPSI0614i-mesr_4 Cells(提供STR鉴定图谱)
KAT-7 Cells(提供STR鉴定图谱)
MELBA Cells(提供STR鉴定图谱)
NH50355 Cells(提供STR鉴定图谱)
RAE25 Cells(提供STR鉴定图谱)
Ubigene HeLa MAP3K20 KO Cells(提供STR鉴定图谱)
XLC303 Cells(提供STR鉴定图谱)
HEK293TN PBRM1 KO 1 Cells(提供STR鉴定图谱)
U031 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HEL299细胞、NCIH1385细胞、CCF-STTG1细胞
T 84 Cells;背景说明:T84细胞株是从一位72岁男性结肠癌患者的肺转移灶建立的可移植人类癌细胞株。 肿瘤组织皮下接种于BALB/c裸鼠,并连续进行移植。 [26072] 在裸鼠身上的移植过程中,细胞株始终保持结肠癌的原始组织性状。 [26072] 在无胸腺小鼠中传代23代后建立了T84细胞株。 这些细胞单层生长到饱和并在接触细胞间展现出紧密连接和桥粒。 [1155] 有很多关于多肽类激素和神经递质并维持定向电解质传输的受体。 [1155] 这株细胞展现了接触细胞中的紧密连接和桥粒。 [1155] 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2-1:4传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞,多角;相关产品有:SACC83细胞、Strain L-929细胞、BCP1细胞
RS1 Cells;背景说明:该细胞系来源于一大鼠的皮肤组织。2007年由中国科学院昆明细胞库建立。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HT 144细胞、MSTO-211细胞、Namalwa细胞
UACC812 Cells;背景说明:该细胞是由Liebovitz A等于1986年从一名43岁的白人女性乳腺导管癌患者的乳腺切除肿瘤组织中分离建立的;手术前该病人曾接受过广泛的化疗。该细胞HER-2/neu癌基因序列有15倍的扩增;雌激素受体ER、孕激素受体PR和糖蛋白P阴性。;传代方法:1:3传代;5-7天1次。 ;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SNU-719细胞、HAEC细胞、SW1990细胞
Ramos-RA1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-740细胞、H2052_MM细胞、870细胞
Ramos-RA1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-740细胞、H2052_MM细胞、870细胞
HEK-293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ramos 2G6.4C10细胞、OCI-Ly19细胞、NPA'87细胞
EO771 Cells;背景说明:恶性乳腺癌;雌性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH841细胞、BALB/3T3 cl. A31细胞、Hs 852.T细胞
HCC0202 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:B-CPAP细胞、HT-29/CX-1细胞、OV-1063细胞
SW-1463 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:8传代,每周换液1-2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:HO1-N-1细胞、MCF.7细胞、BEAS2B细胞
Ramos 2G6.4C10 Cells;背景说明:详见相关文献介绍;传代方法: 维持细胞浓度在2×105/ml-1×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NSI/1-Ag4-1细胞、Hx147细胞、no.11细胞
Ramos.G6.C10 Cells;背景说明:详见相关文献介绍;传代方法: 维持细胞浓度在2×105/ml-1×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MC-3T3细胞、ONS-76细胞、HUC细胞
LL/2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDA-436细胞、TCCPAN2细胞、BEP2D细胞
Det 562 Cells;背景说明:器官:咽头 疾病:癌 取材转移灶:胸水;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Loucy细胞、G402细胞、NCI-H1882细胞
CAL12T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L6细胞、SEG1细胞、NCI H747细胞
St42 Cells(提供STR鉴定图谱)
HT 1197.T Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代,2天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:RBSMC细胞、RMS 13细胞、KNS-42细胞
LN-382 Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:W256细胞、SVGp12细胞、U-266细胞
CEM/0 Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCI-H1734细胞、GM01232E细胞、NTera-2D1细胞
A-172 MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC-827细胞、COLO-824细胞、MEC1细胞
Walker/LLC-WRC 256 Cells;背景说明:乳腺癌;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM05384细胞、Panc8.13细胞、HCT 8细胞
HuH-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NIH:OVCAR-8细胞、DHL10细胞、NCI-H2291细胞
WSU-HN13 Cells;背景说明:舌鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPMI 8402细胞、H2073细胞、DAN-G细胞
P-3J Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:TE14细胞、NCI-H1944细胞、VMCUB-1细胞
SNU-484 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T.T细胞、KTC-1细胞、EBNA293细胞
Hs888Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:COLO 829细胞、NCTC clone 929细胞、SUM-159-PT细胞
IGROV-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:G361细胞、STC-1细胞、OV 2008细胞
HOK Cells;背景说明:口腔角质 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:T98 G细胞、Jurkat细胞、MEG01细胞
IHH Cells;背景说明:肝;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU-484细胞、PC10细胞、Hs_578t细胞
PK-13 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BT483细胞、U118细胞、CBRH7919细胞
JeKo-1人套细胞淋巴瘤复苏细胞保种中心|带STR证书
BayGenomics ES cell line RRF262 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG471 Cells(提供STR鉴定图谱)
EC-Slc22a6tm1Dgen Cells(提供STR鉴定图谱)
mPitA-6 Cells(提供STR鉴定图谱)
TPA2-14 Cells(提供STR鉴定图谱)
M4Be 9TP180 Cells(提供STR鉴定图谱)
" "PubMed=16448697; DOI=10.1016/j.leukres.2005.11.013
Camps J., Salaverria I., Garcia M.J., Prat E., Bea S., Pole J.C.M., Hernandez L., Del Rey J., Cigudosa J.C., Bernues M., Caldas C., Colomer D., Miro R., Campo E.
Genomic imbalances and patterns of karyotypic variability in mantle-cell lymphoma cell lines.
Leuk. Res. 30:923-934(2006)
PubMed=16960149; DOI=10.1182/blood-2006-06-026500
Mestre-Escorihuela C., Rubio-Moscardo F., Richter J.A., Siebert R., Climent J., Fresquet V., Beltran E., Agirre X., Marugan I., Marin M., Rosenwald A., Sugimoto K.-j., Wheat L.M., Karran E.L., Garcia J.F., Sanchez-Verde L., Prosper F., Staudt L.M., Pinkel D., Dyer M.J.S., Martinez-Climent J.A.
Homozygous deletions localize novel tumor suppressor genes in B-cell lymphomas.
Blood 109:271-280(2007)
PubMed=17332242; DOI=10.1182/blood-2006-11-057208
Pinyol M., Bea S., Pla L., Ribrag V., Bosq J., Rosenwald A., Campo E., Jares P.
Inactivation of RB1 in mantle-cell lymphoma detected by nonsense-mediated mRNA decay pathway inhibition and microarray analysis.
Blood 109:5422-5429(2007)
PubMed=21746927; DOI=10.1073/pnas.1018941108; PMCID=PMC3145731
Beltran E., Fresquet V., Martinez-Useros J., Richter-Larrea J.A., Sagardoy A., Sesma I., Almada L.L., Montes-Moreno S., Siebert R., Gesk S., Calasanz M.J., Malumbres R., Rieger M., Prosper F., Lossos I.S., Piris M.A., Fernandez-Zapico M.E., Martinez-Climent J.A.
A cyclin-D1 interaction with BAX underlies its oncogenic role and potential as a therapeutic target in mantle cell lymphoma.
Proc. Natl. Acad. Sci. U.S.A. 108:12461-12466(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25315077; DOI=10.3109/10428194.2014.970548
Fogli L.K., Williams M.E., Connors J.M., Reid Y.A., Brown K., O'Connor O.A.
Development and characterization of a Mantle Cell Lymphoma Cell Bank in the American Type Culture Collection.
Leuk. Lymphoma 56:2114-2122(2015)
PubMed=25355872; DOI=10.1128/JVI.02570-14; PMCID=PMC4301145
Cao S.-B., Strong M.J., Wang X., Moss W.N., Concha M., Lin Z., O'Grady T., Baddoo M., Fewell C., Renne R., Flemington E.K.
High-throughput RNA sequencing-based virome analysis of 50 lymphoma cell lines from the Cancer Cell Line Encyclopedia project.
J. Virol. 89:713-729(2015)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25688540; DOI=10.1002/cyto.a.22643
Maiga S., Brosseau C., Descamps G., Dousset C., Gomez-Bougie P., Chiron D., Menoret E., Kervoelen C., Vie H., Cesbron A., Moreau-Aubry A., Amiot M., Pellat-Deceunynck C.
A simple flow cytometry-based barcode for routine authentication of multiple myeloma and mantle cell lymphoma cell lines.
Cytometry A 87:285-288(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26194763; DOI=10.1182/blood-2015-01-624585; PMCID=PMC4582332
Bhatt S., Matthews J., Parvin S., Sarosiek K.A., Zhao D.-K., Jiang X.-Y., Isik E., Letai A.G., Lossos I.S.
Direct and immune-mediated cytotoxicity of interleukin-21 contributes to antitumor effects in mantle cell lymphoma.
Blood 126:1555-1564(2015)
PubMed=26439697; DOI=10.18632/oncotarget.5954; PMCID=PMC4741926
Restelli V., Chila R., Lupi M., Rinaldi A., Kwee I., Bertoni F., Damia G., Carrassa L.
Characterization of a mantle cell lymphoma cell line resistant to the Chk1 inhibitor PF-00477736.
Oncotarget 6:37229-37240(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27697772; DOI=10.1182/blood-2016-06-720490
Chiron D., Bellanger C., Papin A., Tessoulin B., Dousset C., Maiga S., Moreau A., Esbelin J., Trichet V., Chen-Kiang S., Moreau P., Touzeau C., Le Gouill S., Amiot M., Pellat-Deceunynck C.
Rational targeted therapies to overcome microenvironment-dependent expansion of mantle cell lymphoma.
Blood 128:2808-2818(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29666304; DOI=10.1158/1078-0432.CCR-17-3004
Pham L.V., Huang S.-J., Zhang H., Zhang J., Bell T., Zhou S.-H., Pogue E., Ding Z.-Y., Lam L., Westin J., Davis R.E., Young K.H., Medeiros L.J., Ford R.J. Jr., Nomie K., Zhang L., Wang M.
Strategic therapeutic targeting to overcome venetoclax resistance in aggressive B-cell lymphomas.
Clin. Cancer Res. 24:3967-3980(2018)
PubMed=30285677; DOI=10.1186/s12885-018-4840-5; PMCID=PMC6167786
Tan K.-T., Ding L.-W., Sun Q.-Y., Lao Z.-T., Chien W., Ren X., Xiao J.-F., Loh X.-Y., Xu L., Lill M., Mayakonda A., Lin D.-C., Yang H.H., Koeffler H.P.
Profiling the B/T cell receptor repertoire of lymphocyte derived cell lines.
BMC Cancer 18:940.1-940.13(2018)
PubMed=30629668; DOI=10.1371/journal.pone.0210404; PMCID=PMC6328144
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"
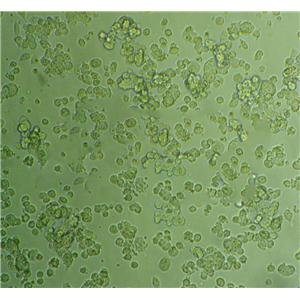




![Jeko-1[人套细胞淋巴瘤细胞]](https://img.chemicalbook.com/SupplyImg/2019-08-31/Large/201908311927376495942.jpg)