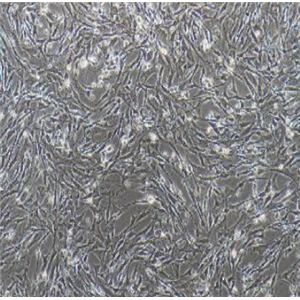
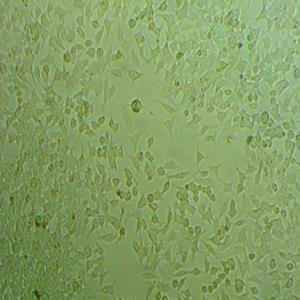
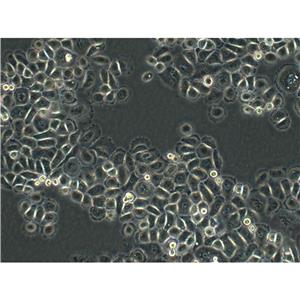
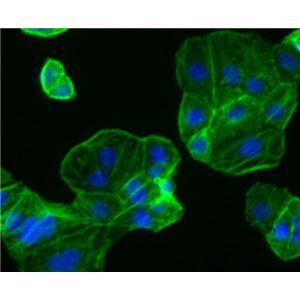
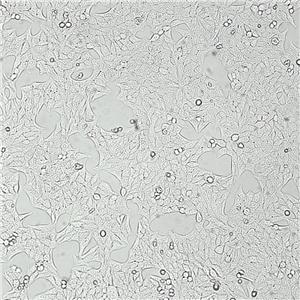
"Hep 3B2.1-7人肝癌复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞传代培养实验:体外培养的原代细胞或细胞株要在体外持续地培养就必须传代,以便获得稳定的细胞株或得到大量的同种细胞,并维持细胞种的延续。培养的细胞形成单层汇合以后,由于密度过大生存空间不足而引起营养枯竭,将培养的细胞分散,从容器中取出,以1:2或1:3以上的比率转移到另外的容器中进行培养,即为传代培养;细胞“一代”指从细胞接种到分离再培养的一段期间,与细胞世代或倍增不同。在一代中,细胞培增3~6次。细胞传代后,一般经过三个阶段:游离期、指数增生期和停止期。常用细胞分裂指数表示细胞增殖的旺盛程度,即细胞群的分裂相数/100个细胞。一般细胞分裂指数介于0.2%~0.5%,肿瘤细胞可达3~5%;细胞接种2~3天分裂增殖旺盛,是活力ZuiHAO时期,称指数增生期(对数生长期),适宜进行各种试验。实验步骤:1.将长成的培养细胞从二氧化碳培养箱中取出,在超净工作台中倒掉瓶内的培养,加入少许消化。(以面盖住细胞为宜),静置5~10分钟。2.在倒置镜下观察被消化的细胞,如果细胞变圆,相互之间不再连接成片,这时应立即在超净台中将消化倒掉,加入3~5ml新鲜培养,吹打,制成细胞悬。3.将细胞悬吸出2ml左右,加到另一个培养瓶中并向每个瓶中分别加3ml左右培养,盖HAO瓶塞,送回二氧化碳培养箱中,继续进行培养。一般情况,传代后的细胞在2小时左右就能附着在培养瓶壁上,2~4天就可在瓶内形成单层,需要再次进行传代。
换液周期:每周2-3次
P3J-HR-1 Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:HFF1细胞、HIEC6细胞、MCF7-GFP细胞
MOLP-8 Cells;背景说明:浆细胞骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs 742.T细胞、HSC-4细胞、SNU475细胞
NPC-TW01 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Pfeiffer细胞、H1770细胞、C57/B6-L细胞
Hep 3B2.1-7人肝癌复苏细胞保种中心|带STR证书
背景信息:是一种人肝癌细胞系,是来自8岁男性黑人的组织。Hep 3B2.1-7细胞的染色体模式数目为60,在裸鼠中能致瘤。Hep 3B2.1-7细胞整合了完整的乙型肝炎病毒基因组。
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
COLO 201 Cells;背景说明:该细胞源自一位70岁白人男性,CSAp (CSAp-)和CEA阴性。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮+贴壁;形态特性:淋巴细胞;相关产品有:Madin Darby Canine Kidney细胞、HuP-T3细胞、Bovine Turbinate细胞
PC 61.5.3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:alpha TC1 clone 6细胞、SK-MEL-MeWo细胞、U-251-MG细胞
H920 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HG2855细胞、SPCA-1细胞、MA-c细胞
DanG Cells;背景说明:胰腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:207细胞、TGW细胞、T2(174 x CEM.T2)细胞
Hep 3B2.1-7人肝癌复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:上皮细胞样
细胞冻存知识普及:细胞冻存是细胞保存的主要方法之一。利用冻存技术将细胞置于-196℃中低温保存,可以使细胞暂时脱离生长状态而将其细胞性保存起来,这样在需要的时候再复苏细胞用于实验。而且适度地保存一定量的细胞,可以防止因正在培养的细胞被污染或其他意外事件而使细胞丢种,起到了细胞保种的作用。除此之外,还可以利用细胞冻存的形式来购买、寄赠、交换和运送某些细胞。细胞冻存时向培养基中加入保护剂--终浓度5%.15%的甘油或二甲基亚砜(DMSO),可使溶冰点降低,加之在缓慢冻结条件下,细胞内水分透出,减少了冰晶形成,从而避免细胞损伤。 采用""慢冻快融""的方法能较HAO地保证细胞存活。标准冷冻速度开始为-1到-2℃/min,当温度低于-25℃时可加速,到-80℃之后可直接投入内(-196℃)。
NR8383 Cells;背景说明:NR8383(正常大鼠,1983年8月3日)来源于肺灌洗时的正常大鼠肺泡巨噬细胞。细胞在gerbil肺细胞连续培养液存在下培养了大约8-9个月。随后,不再需要外源生长因子。通过有限稀释法从单个细胞克隆并亚克隆NR8383细胞,并三次用软琼脂亚克隆。细胞表现出巨噬细胞的特性,吞噬酵母多糖和铜绿,非特异性脂酶活性,Fc受体,氧化降解;分泌IL-1,TNFbeta和IL-6,可重复地响应外源生长因子。NR8383细胞响应博莱霉素,分泌TGFbeta前体。在博莱霉素刺激下,TGFbe;传代方法:1:2传代;生长特性:半贴壁生长;形态特性:巨噬细胞;相关产品有:HAPI细胞、WM 451-Lu细胞、HEK293细胞
C17.2 Cells;背景说明:神经干细胞;C57BL/6 x CD-1;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U 138 MG细胞、Eca109细胞、U266S细胞
HEK293FT Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:U-87-MG细胞、251MG细胞、B16F10细胞
CATHa Cells;背景说明:神经;SV40转化;C57BL/6 x DBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KMB17细胞、GM 637细胞、GM 637细胞
NS20-Y Cells;背景说明:神经母细胞瘤;雄性;A/J;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:F442-A细胞、HeLa229细胞、FM-88细胞
MCAEC Cells;背景说明:冠状动脉内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM15452细胞、293T细胞、McA-RH 7777细胞
H-7721 Cells;背景说明:用Northernblot方法,未能检测到细胞中1.3kbLFIRE-1/HFREP-1mRNA的表达。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:TSU-Pr1细胞、TGBC-11-TKB细胞、HEp-2细胞
LN 382 Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-N-FI细胞、T98G细胞、HuNS1细胞
RPMI8402 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:C-33 A细胞、MDCC MSB1细胞、Rat 1细胞
RPMI 7951 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:X63.Ag8.653细胞、BEAS 2B细胞、SU-DHL-4细胞
HRCEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Calu3细胞、McArdle RH-7777细胞、OCI-Ly 10细胞
RPMI no 8226 Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCIH82细胞、KYSE-450细胞、ARH77细胞
CCRFCEM Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:MM-1S细胞、SR786细胞、HTori:3细胞
COLO824 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:U87MG细胞、G422细胞、H146细胞
NCIH1838 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:High Five细胞、A-20细胞、RMS 1598细胞
Mel526 Cells;背景说明:黑色素瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-MS细胞、KCL-22细胞、KRC/Y细胞
UACC 812 Cells;背景说明:该细胞是由Liebovitz A等于1986年从一名43岁的白人女性乳腺导管癌患者的乳腺切除肿瘤组织中分离建立的;手术前该病人曾接受过广泛的化疗。该细胞HER-2/neu癌基因序列有15倍的扩增;雌激素受体ER、孕激素受体PR和糖蛋白P阴性。;传代方法:1:3传代;5-7天1次。 ;生长特性:贴壁生长;形态特性:上皮样;相关产品有:RL-952细胞、VMM5细胞、Tu212细胞
Hep 3B2.1-7人肝癌复苏细胞保种中心|带STR证书
1328-8139 Cells(提供STR鉴定图谱)
Abcam HeLa EIF4EBP1 KO Cells(提供STR鉴定图谱)
AG20116 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRF118 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG302 Cells(提供STR鉴定图谱)
C0131 Cells(提供STR鉴定图谱)
CW11120 Cells(提供STR鉴定图谱)
EC-MZ-2 Cells(提供STR鉴定图谱)
GM05023 Cells(提供STR鉴定图谱)
RTE Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,3-4天换液一次;生长特性:贴壁生长;形态特性:多角;相关产品有:ECC-1细胞、UACC 893细胞、H-250细胞
Melan-a Cells;背景说明:黑色素细胞;自发永生;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCC15细胞、HNE1细胞、HPAF-2细胞
Neuro-2a Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HBSMC细胞、IOSE80细胞、Madin Darby Bovine Kidney细胞
KALS1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:多边形;相关产品有:NCIH209细胞、2BS细胞、Cloudman S91 melanoma细胞
NRK clone 49F Cells;背景说明:肾;成纤维细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H322T细胞、OVCA-420细胞、OE21细胞
MIA Paca2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HUT-28细胞、rRTEC细胞、BL2141细胞
9.4 Cells(提供STR鉴定图谱)
b.End3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Calu 6细胞、C1498细胞、8305-C细胞
CHP 126 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-23细胞、OVCAR.3细胞、Cates-1B细胞
TE671 Cells;背景说明:胚胎性横纹肌肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:AU-Mel细胞、SK.MEL.28细胞、Y3.AG.1.2.3细胞
LuCL4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CD 18细胞、BC-025细胞、Baby Hamster Kidney 21细胞
C3H/10T1/2 CL8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MT4细胞、NUGC3细胞、MCF.10A细胞
NCI-H865 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SU-DHL-6细胞、AZ-521细胞、SU-DHL-2细胞
TCC Sup Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Hs 695.T细胞、OCI-Ly01细胞、OCILY10细胞
R1800[RA] Cells;背景说明:星形胶质 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:EFM-192A细胞、NCI H716细胞、NCI-SNU-398细胞
GMA32 Cells(提供STR鉴定图谱)
HAP1 PIM1 (-) 3 Cells(提供STR鉴定图谱)
A2780CP70 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P3 NS1 Ag4/1细胞、THLE2细胞、B16 melanoma细胞
Tregs Cells;背景说明:调节性T Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Epstein-Barr-3细胞、SCC9细胞、SKNBE2细胞
Raji Cells;背景说明:Raji细胞由PulvertaftRJV于1963年从一位11岁黑人男孩的左上颌骨的Burkitt淋巴瘤中分离建立的,是第一个人类造血系统的连续传代细胞,为B细胞起源。该细胞中含有EBV,需要在二级生物安全柜中操作;可作转染宿主。;传代方法:维持细胞浓度在4×105/ml-3×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:VAESBJ细胞、Hs737T细胞、HGC-27细胞
BDCM Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:OCI/AML2细胞、Plaepi 34细胞、SUIT-2细胞
DoHH2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:VMRCRCZ细胞、KYSE 180细胞、H-2291细胞
COV-362 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:BAEC细胞、MKN45细胞、PLB 985细胞
TCC-PAN2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Suzhou Human Glioma-44细胞、LMH细胞、IOSE-Van细胞
SuDHL 8 Cells;背景说明:弥漫大B淋巴瘤;腹腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:ACC-M细胞、Duck embryo细胞、SK-MEL-28细胞
HSP-239 Cells(提供STR鉴定图谱)
KP-4-4 Cells(提供STR鉴定图谱)
MoOV-51 Cells(提供STR鉴定图谱)
NYSCF-10005-686-686-Skin-mR-iPSC Cells(提供STR鉴定图谱)
RGS-3A Cells(提供STR鉴定图谱)
Ubigene RAW 264.7 Cgas KO Cells(提供STR鉴定图谱)
Yub634 Cells(提供STR鉴定图谱)
HCC420 Cells(提供STR鉴定图谱)
CL 1-0 Cells;背景说明:肺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HFT-8810细胞、U373-MG细胞、NCI-H292细胞
SF-763 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCRF SB细胞、RH8994细胞、L cell line细胞
NCI-H2066 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:Panc_08_13细胞、Hs606T细胞、3T3细胞
H1092 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:MM134细胞、C166细胞、MCF/Adr细胞
CAMA1 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞贴壁斑块;紧凑,很少汇合;相关产品有:HL-7702细胞、MX-1细胞、2V6.11细胞
CAMA1 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞贴壁斑块;紧凑,很少汇合;相关产品有:HL-7702细胞、MX-1细胞、2V6.11细胞
CL-34 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDA134细胞、IOSE-29细胞、Hep 3B细胞
GM16136 Cells;背景说明:肺;自发永生;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RGM1细胞、MUS-M1细胞、C3H10T1-2细胞
Lilly Laboratories Cell-Porcine Kidney 1 Cells;背景说明:肾;自发永生;Hampshire;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LIM 1215细胞、HMC-1细胞、HuH28细胞
MRC-V Cells;背景说明:MRC-5细胞系来自14周龄男性胎儿的正常肺组织,该细胞老化前能传代42~46个倍增时间。;传代方法:1:2-1:5传代;每周1-2次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Human Pancreatic Duct Epithelial细胞、HSC 3细胞、HO8910细胞
Caov-4 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IR983F细胞、Cor L51细胞、SW13细胞
Capan1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SK-RC-20细胞、UWB1-289细胞、HRMC细胞
SCC-9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Ls-174-T细胞、TU 686细胞、A2780/CP70细胞
H292 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SNU620细胞、BALB/3T3 clone A31细胞、NCI-H157细胞
Hs 746.T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SK_N_BE2C细胞、U87 MG细胞、Pt K2 (NBL-5)细胞
SUP-B2 Cells(提供STR鉴定图谱)
CCRF/CEM-C7 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SLMT-1细胞、COLO206细胞、SCCVII/St细胞
RPPVEC Cells;背景说明:肺静脉;血管内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H209细胞、HCT FET细胞、UACC893细胞
IOSE-80 Cells;背景说明:卵巢;上皮细胞;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:mouse Inner Medullary Collecting Duct-3细胞、LO2细胞、H1781细胞
SW1990 Cells;背景说明:胰腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU1040细胞、CACO2细胞、C-4I细胞
MM1 Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MV 4;11细胞、Tregs细胞、NIH-3T3细胞
CL-11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:University of Michigan-Urothelial Carcinoma-1细胞、MGH-U3 (RN)细胞、RC13细胞
HN13 Cells;背景说明:舌鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:16-HBEo细胞、B16 F10细胞、R 1610细胞
E.L.4 Cells;背景说明:EL4是从用9,10-二甲基-1,2-并蒽在C57BL小鼠中诱导的淋巴瘤中建立的。 能抗0.1 mM 化可的松,对20 mcg/ml PHA敏感。 还有一个亚株(EL4.IL-2, ATCC TIB-181)可以生成高水平的IL-2。 检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:HEL9217细胞、HEL299细胞、SVHUC细胞
HTori-3 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LM6细胞、Hs 746T细胞、MUG-Chor1细胞
DMS-114 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MDAMB-231细胞、3396细胞、Panc_02_03细胞
RCK-8 Cells;背景说明:弥漫大B淋巴瘤;腹腔转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:University of Arizona Cell Culture-893细胞、NCIH727细胞、GM-3573细胞
UO-31 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Human Kidney-2细胞、GM637A细胞、LM8细胞
RL-65 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-415细胞、SKG IIIa细胞、TEV-1细胞
ACC2 Cells;背景说明:涎腺腺样囊性癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Evsa-T细胞、Mouse podocyte细胞、CPA47细胞
Hep 3B2.1-7人肝癌复苏细胞保种中心|带STR证书
BayGenomics ES cell line CSH473 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRY563 Cells(提供STR鉴定图谱)
Bhas42 Cells(提供STR鉴定图谱)
KRIX-1 Cells(提供STR鉴定图谱)
PEAA-3B5 Cells(提供STR鉴定图谱)
PC12-G11 Cells(提供STR鉴定图谱)
" "PubMed=6288577; DOI=10.1002/ijc.2910300106
Simon D., Aden D.P., Knowles B.B.
Chromosomes of human hepatoma cell lines.
Int. J. Cancer 30:27-33(1982)
Patent=US4393133
Knowles B.B., Aden D.P.
Human hepatoma derived cell line, process for preparation thereof, and uses therefor.
Patent number US4393133, 12-Jul-1983
PubMed=3023526; DOI=10.1099/0022-1317-67-11-2315
Aspinall S., Alexander J.J., Bos P.
Comparative expression of hepatitis B virus antigens in several cell model systems.
J. Gen. Virol. 67:2315-2323(1986)
DOI=10.1007/978-4-431-68349-0_4
Alexander J.J.
Human hepatoma cell lines.
(In book chapter) Neoplasms of the liver; Okuda K., Ishak K.G. (eds.); pp.47-56; Springer; Tokyo; Japan (1987)
PubMed=2439335; DOI=10.1111/j.1432-1033.1987.tb11497.x
Vincent C., Marceau M., Blangarin P., Bouic P., Madjar J.-J., Revillard J.-P.
Purification of alpha 1-microglobulin produced by human hepatoma cell lines. Biochemical characterization and comparison with alpha 1-microglobulin synthesized by human hepatocytes.
Eur. J. Biochem. 165:699-704(1987)
PubMed=3431441; DOI=10.1016/s0076-6879(87)51006-0
Darlington G.J.
Liver cell lines.
Methods Enzymol. 151:19-38(1987)
PubMed=8224613; DOI=10.1096/fasebj.7.14.8224613
Puisieux A., Galvin K., Troalen F., Bressac B., Marcais C., Galun E., Ponchel F., Yakicier C., Ji J.-W., Ozturk M.
Retinoblastoma and p53 tumor suppressor genes in human hepatoma cell lines.
FASEB J. 7:1407-1413(1993)
PubMed=8389256; DOI=10.1093/carcin/14.5.987
Hsu I.-C., Tokiwa T., Bennett W.P., Metcalf R.A., Welsh J.A., Sun T.-T., Harris C.C.
p53 gene mutation and integrated hepatitis B viral DNA sequences in human liver cancer cell lines.
Carcinogenesis 14:987-992(1993)
PubMed=7972006; DOI=10.1073/pnas.91.23.11045; PMCID=PMC45163
Okamoto A., Demetrick D.J., Spillare E.A., Hagiwara K., Hussain S.P., Bennett W.P., Forrester K., Gerwin B.I., Serrano M., Beach D.H., Harris C.C.
Mutations and altered expression of p16INK4 in human cancer.
Proc. Natl. Acad. Sci. U.S.A. 91:11045-11049(1994)
PubMed=8050184; DOI=10.1111/j.1365-2249.1994.tb06089.x; PMCID=PMC1534706
Wadee A.A., Paterson A., Coplan K.A., Reddy S.G.
HLA expression in hepatocellular carcinoma cell lines.
Clin. Exp. Immunol. 97:328-333(1994)
PubMed=8835345; DOI=10.1002/(SICI)1096-9071(199602)48:2<133::aid-jmv3>3.0.CO;2-A
Tsuboi S., Nagamori S., Miyazaki M., Mihara K., Fukaya K.-i., Teruya K., Kosaka T., Tsuji T., Namba M.
Persistence of hepatitis C virus RNA in established human hepatocellular carcinoma cell lines.
J. Med. Virol. 48:133-140(1996)
DOI=10.11418/jtca1981.16.3_173
Mihara K., Miyazaki M., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ohashi R., Namba M.
The p53 gene status and other cellular characteristics of human cell lines maintained in our laboratory.
Tissue Cult. Res. Commun. 16:173-178(1997)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::aid-mc5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=9359923; DOI=10.18926/AMO/30789
Mihara K., Miyazaki M., Kondo T., Fushimi K., Tsuji T., Inoue Y., Fukaya K.-i., Ishioka C., Namba M.
Yeast functional assay of the p53 gene status in human cell lines maintained in our laboratory.
Acta Med. Okayama 51:261-265(1997)
PubMed=10523694; DOI=10.3892/or.6.6.1267
Gao C., Ohashi R., Pu H., Inoue Y., Tsuji T., Miyazaki M., Namba M.
Yeast functional assay of the p53 gene status in 11 cell lines and 26 surgical specimens of human hepatocellular carcinoma.
Oncol. Rep. 6:1267-1271(1999)
PubMed=11050057; DOI=10.1053/jhep.2000.19349
Wong N., Lai P.B.-S., Pang E., Leung T.W.-T., Lau J.W.-Y., Johnson P.J.
A comprehensive karyotypic study on human hepatocellular carcinoma by spectral karyotyping.
Hepatology 32:1060-1068(2000)
PubMed=12029633; DOI=10.1053/jhep.2002.33683
Yasui K., Arii S., Zhao C., Imoto I., Ueda M., Nagai H., Emi M., Inazawa J.
TFDP1, CUL4A, and CDC16 identified as targets for amplification at 13q34 in hepatocellular carcinomas.
Hepatology 35:1476-1484(2002)
PubMed=15767549; DOI=10.1158/1535-7163.MCT-04-0234
Nakatsu N., Yoshida Y., Yamazaki K., Nakamura T., Dan S., Fukui Y., Yamori T.
Chemosensitivity profile of cancer cell lines and identification of genes determining chemosensitivity by an integrated bioinformatical approach using cDNA arrays.
Mol. Cancer Ther. 4:399-412(2005)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20454443; DOI=10.1155/2010/904767; PMCID=PMC2861168
Uphoff C.C., Denkmann S.A., Steube K.G., Drexler H.G.
Detection of EBV, HBV, HCV, HIV-1, HTLV-I and -II, and SMRV in human and other primate cell lines.
J. Biomed. Biotechnol. 2010:904767.1-904767.23(2010)
PubMed=20937217; DOI=10.1170/149
Di Masi A., Viganotti M., Antoccia A., Magrelli A., Salvatore M., Azzalin G., Tosto F., Lorenzetti S., Maranghi F., Mantovani A., Macino G., Tanzarella C., Taruscio D.
Characterization of HuH6, Hep3B, HepG2 and HLE liver cancer cell lines by WNT/beta-catenin pathway, microRNA expression and protein expression profile.
Cell. Mol. Biol. 56:OL1299-OL1317(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23505090; DOI=10.1002/hep.26402
Wang K., Lim H.Y., Shi S., Lee J., Deng S.-B., Xie T., Zhu Z., Wang Y.-L., Pocalyko D., Yang W.J., Rejto P.A., Mao M., Park C.-K., Xu J.-C.
Genomic landscape of copy number aberrations enables the identification of oncogenic drivers in hepatocellular carcinoma.
Hepatology 58:706-717(2013)
PubMed=23887712; DOI=10.1038/ncomms3218; PMCID=PMC3731665
Nault J.-C., Mallet M., Pilati C., Calderaro J., Bioulac-Sage P., Laurent C., Laurent A., Cherqui D., Balabaud C., Zucman-Rossi J.
High frequency of telomerase reverse-transcriptase promoter somatic mutations in hepatocellular carcinoma and preneoplastic lesions.
Nat. Commun. 4:2218.1-2218.7(2013)
PubMed=24116068; DOI=10.1371/journal.pone.0075692; PMCID=PMC3792989
Weiskirchen R., Weimer J., Meurer S.K., Kron A., Seipel B., Vater I., Arnold N., Siebert R., Xu L.-M., Friedman S.L., Bergmann C.
Genetic characteristics of the human hepatic stellate cell line LX-2.
PLoS ONE 8:E75692-E75692(2013)
PubMed=25574106; DOI=10.3748/wjg.v21.i1.311; PMCID=PMC4284350
Cevik D., Yildiz G., Ozturk M.
Common telomerase reverse transcriptase promoter mutations in hepatocellular carcinomas from different geographical locations.
World J. Gastroenterol. 21:311-317(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29610054; DOI=10.1016/j.dmpk.2018.03.003; PMCID=PMC6309175
Shi J., Wang X.-W., Lyu L.-Y., Jiang H., Zhu H.-J.
Comparison of protein expression between human livers and the hepatic cell lines HepG2, Hep3B, and Huh7 using SWATH and MRM-HR proteomics: Focusing on drug-metabolizing enzymes.
Drug Metab. Pharmacokinet. 33:133-140(2018)
PubMed=30629668; DOI=10.1371/journal.pone.0210404; PMCID=PMC6328144
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31063779; DOI=10.1053/j.gastro.2019.05.001
Caruso S., Calatayud A.-L., Pilet J., La Bella T., Rekik S., Imbeaud S., Letouze E., Meunier L., Bayard Q., Rohr-Udilova N., Peneau C., Grasl-Kraupp B., de Koning L., Ouine B., Bioulac-Sage P., Couchy G., Calderaro J., Nault J.-C., Zucman-Rossi J., Rebouissou S.
Analysis of liver cancer cell lines identifies agents with likely efficacy against hepatocellular carcinoma and markers of response.
Gastroenterology 157:760-776(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31378681; DOI=10.1016/j.ccell.2019.07.001; PMCID=PMC7505724
Qiu Z.-X., Li H., Zhang Z.-T., Zhu Z.-F., He S., Wang X.-J., Wang P.-C., Qin J.-J., Zhuang L.-P., Wang W., Xie F.-B., Gu Y., Zou K.-K., Li C., Li C., Wang C.-H., Cen J., Chen X.-T., Shu Y.-J., Zhang Z., Sun L.-L., Min L.-H., Fu Y., Huang X.-W., Lv H., Zhou H., Ji Y., Zhang Z.-G., Meng Z.-Q., Shi X.-L., Zhang H.-B., Li Y.-X., Hui L.-J.
A pharmacogenomic landscape in human liver cancers.
Cancer Cell 36:179-193.e11(2019)
PubMed=31395879; DOI=10.1038/s41467-019-11415-2; PMCID=PMC6687785
Yu K., Chen B., Aran D., Charalel J., Yau C., Wolf D.M., van 't Veer L.J., Butte A.J., Goldstein T., Sirota M.
Comprehensive transcriptomic analysis of cell lines as models of primary tumors across 22 tumor types.
Nat. Commun. 10:3574.1-3574.11(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)"

![Hep3B2.1-7 [Hep3B] 人肝癌细胞](https://img.chemicalbook.com/SupplyImg1/2025-08-21/Large/202508211523073448924.jpg)