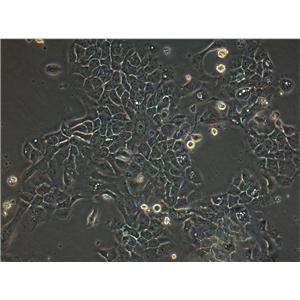
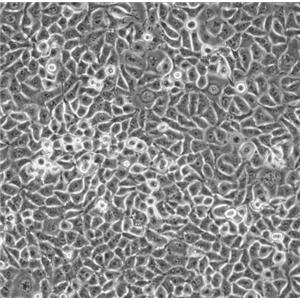
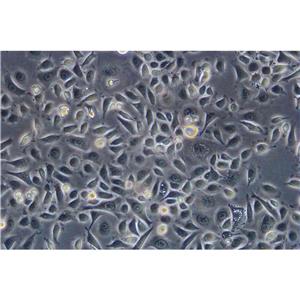
"COLO 205人结肠癌复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
贴壁消化难题:1,先用PBS 把细胞洗两遍,使瓶内没有血清了,减少对胰酶的中和,然后用新配的0.25%的胰酶加入3ml左右,放在37度,然后可以在细胞有些消化下来时,拿着瓶口,运用手腕的力量轻轻震荡瓶内体,这样细胞很快就下来了,还不需要吹打,分散也均匀;2,成团、絮状:消化里加入eda可以减少细胞成团的现象,血清可以终止胰酶的作用,如果是进口血清的话也能终止eda的作用。用胰酶消化后胰酶可以倒掉,也可以不倒,直接加血清终止,如果消化中加入了eda的话,就要将消化倒干净,如果细胞贴壁要求不是很严格的话,一般不需要进行离心。鼻咽癌细胞的贴壁能力很强,用0.5%胰酶(含0.1%EDA)一般要消化12~15min。用PBS洗涤时要洗净残余的培养基,加入胰酶后在培养箱中消化(避免细胞室温下受损以及在此温度时胰酶活性Zui强)至细胞收缩变圆(可显微镜下观察)且有少许细胞脱落(有流下来的趋势),随后立即弃去胰酶(如果脱落的细胞很多且需要大量细胞实验,则不能弃去胰酶),加入培养基仔细吹打(不能用无血清培养基或者PBS替代,否则细胞聚集成团块或絮状)。一般我都离心一次弃去上清(去除残留的胰酶及漂浮的死细胞或细胞碎片);消化过度:马上用培养基中和,用吸管吹打细胞,收集全部的细胞到以无菌的离心管中800RPM 3分钟。弃上清,用全培重悬,换新的培养瓶继续培养,状态不HAO的细胞在培养的过程中会死亡脱落,在换的时候可以清除掉!
换液周期:每周2-3次
P3J.HR1K Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:H-510A细胞、B16BL6细胞、SUSM1细胞
FM-88 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KM H-2细胞、MC26细胞、N1E115细胞
FBHE Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1781细胞、A2780/CP细胞、MHCC97L细胞
COLO 205人结肠癌复苏细胞保种中心|带STR证书
背景信息:该细胞系是1957年由T.U.Sle等从患有结肠癌的70岁男性白人的腹水中分离的。该病人在取腹水样前已用5-尿嘧啶治疗4~6周。角蛋白免疫过氧化物酶染色阳性;产生A、IL10。
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
Mv 1 Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Sf-21细胞、C4-1细胞、RL-65细胞
L-132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CAL851细胞、EVSA/T细胞、OCI/AML5细胞
AM-38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NRK52E细胞、EBNA-293细胞、HN-6细胞
Sf21 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:U-251_MG细胞、NHRF细胞、HT-3细胞
COLO 205人结肠癌复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:上皮细胞样
很多朋友讨论细胞培养问题,见到很多很HAO的经验总结,这里说一些我个人的经验,因为一些比较殊的原因,我自己培养接触过近300种细胞,常用于做实验的,累积有百余种,可以说遇到过许多各式各样古怪的细胞。这里挑细胞消化开始做我的第一篇专题,并不是因为细胞消化Zui重要,而是近期看到大家频繁讨论这个话题,我就抛砖引玉,下面几点拙见,可能与其它朋友总结的一些经验有点出入,仅供大家参考。许多同学对细胞消化做了许多研究,得出了很多有价值的经验,也见到很多人的困惑。其实细胞培养,尤其是细胞系的培养,就消化而言,不是太难,做得多了,善于总结经验,就能把细胞越养越HAO。一般的程序步骤,细节操作,我这里就不讲了,只讲一些基本操作背后的知识,如果不对之处,欢迎指正,总结下来,细胞培养中消化很重要,但其实消化并不是细胞培养的关键所在(虽然很重要),关键所在是细胞来源,血清质量和水源(自己配制溶的话)。我看到版上许多人养细胞遇到各种各样的问题,很多时候boleneck没有找到,虽然通过其它方法有所改善,但仍然是事倍功半。因为人们往往注意自己的操作,总觉得自己没有经验,而忽视试剂,溶尤其是细胞的质量,后者其实才是许多细胞培养实验室常见的问题。
Mc Coy Cells;背景说明:成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FHs74 Int细胞、526细胞、KASUMI1细胞
KU 812F Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RPMI-8226S细胞、MRC-9细胞、HCC1171细胞
VM-CUB1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HSC4细胞、MSCs(mUCMSCs)细胞、SW-948细胞
HLE-SRA-01/04 Cells;背景说明:晶状体;上皮细胞;SV40转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Panc2.03细胞、CEMC7细胞、NTera 2细胞
NCI-H2444 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MPC-5细胞、NCI-H2009细胞、KPL4细胞
B16-F0 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs737T细胞、AK细胞、HOEC细胞
KS-1 [Human glioblastoma] Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:BEL 7405细胞、BC-021细胞、TE-7细胞
SNU-423 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CNE-2细胞、SRA01/04 (HLE)细胞、CTLA4 Ig-24细胞
DSL-6A-C1 Cells;背景说明:胰腺癌;雄性;Lewis;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HTh 74细胞、SW-1271细胞、HMEC细胞
WEHI-3 Cells;背景说明:白血病;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:BERH-2细胞、PC-12细胞、SKGT4细胞
H4-II-E Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hmy.2 CIR细胞、PANC 203细胞、624细胞
H676B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CEM T4细胞、AN3细胞、ROS1728细胞
Ishikawa Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,3-4天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:C166细胞、CCD19Lu细胞、SUM-102PT细胞
RPMI.8226 Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCIH250细胞、KYSE 50细胞、KMB 17细胞
Acanthosis Nigricans 3rd attempt-CArcinoma Cells;背景说明:AN3CA细胞建系于1964年。它衍生于子宫内膜癌患者淋巴结转移组织,具有癌细胞的基本特性,能在体外长期传代培养,接种实验动物产生明显肿瘤。但细胞的生物学特性及超微结构尚未深入研究,仅发现该细胞系促黑激素合成为阴性。细胞常用于人子宫内膜癌细胞生物学及其相关特性研究。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:U251细胞、SNU1040细胞、MLFC细胞
OV2008 Cells;背景说明:宫颈鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NS1-Ag4/1细胞、HRC-99细胞、NCI-H727细胞
HEY-A8 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H1395细胞、SNU520细胞、CHO cell clone K1细胞
COLO 205人结肠癌复苏细胞保种中心|带STR证书
Abcam HCT 116 TSC1 KO Cells(提供STR鉴定图谱)
AG08543 Cells(提供STR鉴定图谱)
BayGenomics ES cell line HMA455 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XB632 Cells(提供STR鉴定图谱)
BoPr-49 Cells(提供STR鉴定图谱)
CNS1-hiPSC5 Cells(提供STR鉴定图谱)
DA03768 Cells(提供STR鉴定图谱)
Ff-XT18s03 Cells(提供STR鉴定图谱)
GM08776 Cells(提供STR鉴定图谱)
MV4-11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EBTr (NBL-4)细胞、AC16 [Human hybrid]细胞、SKLU01细胞
HCC-2108 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Human Embryonic Skin细胞、N-9细胞、KYSE-520细胞
COR-L279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NS1细胞、Caov-4细胞、HCC0015细胞
Centre Antoine Lacassagne-62 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MESSA细胞、IHC-ST1细胞、CEM-CCRF (CAMR)细胞
SK-RC 42 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW 13细胞、Hs 870.T细胞、USMC细胞
BCaP-37 Cells;背景说明:源自一位48岁女性乳癌患者。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCT-FET细胞、High Five细胞、H22-H8D8细胞
Ba/F3 EGFR-L718V Cells(提供STR鉴定图谱)
CEMC7 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HPDLF细胞、Jijoye细胞、H.Ep. #2细胞
MS 751 Cells;背景说明:这株细胞是J. Sykes于1974年建立的(参考ATCC HTB-33)。有报告称MS751细胞含有人乳头状瘤病毒18 (HPV-18)序列。[22995] [23180]后来发现MS751细胞包含HPV-45基因组的一部分,而其中E6/E7区域表达呈poly(A)+RNA的形式。[49721];传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:OVCA5细胞、BALB 3T3 clone A31细胞、NCI-H520细胞
L929(NCTC) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C-26细胞、GP-293细胞、AHH1细胞
HPAC Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:JVM-3细胞、H2198细胞、YES2细胞
A549 ATCC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ECC1细胞、NCI-H2023细胞、NCIH2122细胞
H810 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:C-6细胞、L-929细胞、SW 756细胞
UWB1.289+BRCA1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Capan 1细胞、MDA-MB-435细胞、SUM149细胞
MDA-453 Cells;背景说明:该细胞系由CailleauR在1976年从一名48岁的患有转移性乳腺癌的白人女性的心包渗出液中分离建立的。该细胞表达FGF的受体。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:JCA1细胞、CHP-100细胞、Caco-2/BBe 1细胞
GM19485 Cells(提供STR鉴定图谱)
HAP1 INPP5A (-) 2 Cells(提供STR鉴定图谱)
TE-1 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OC316细胞、Sf-21细胞、HCC-1395细胞
NRK49F Cells;背景说明:肾;成纤维细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SN4741细胞、GM04678细胞、3T3-L1 ad细胞
NIH:OVCAR-5 Cells;背景说明:卵巢癌;腹水转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Ca9-22细胞、HCT-GEO细胞、Hs870T细胞
Mv1.Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDA-MB-361细胞、H1975细胞、P3X63细胞
Clone M-3 Cells;背景说明:黑色素瘤;雄性;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA-MB435细胞、MOPC细胞、SNU-368细胞
Mel RM Cells;背景说明:黑色素瘤;神经节转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MFC细胞、HEK-293-F细胞、H2135细胞
DMS273 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CF PAC-1细胞、CC-LP-I细胞、P-815细胞
Huh7.5.1 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H716细胞、MM96L细胞、LoMeT-ccRcc细胞
HPS0680 Cells(提供STR鉴定图谱)
JMAR Cells(提供STR鉴定图谱)
MDCC-BO1(T) Cells(提供STR鉴定图谱)
ND40077 Cells(提供STR鉴定图谱)
PSALBV245 Cells(提供STR鉴定图谱)
Ubigene A-549 LAMC1 KO Cells(提供STR鉴定图谱)
US/VOT-E36 Cells(提供STR鉴定图谱)
HG01954 Cells(提供STR鉴定图谱)
130 T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:H2106细胞、3396细胞、MDAMB231细胞
D-283MED Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮细胞的多细胞聚集体,和一些贴壁 Cells;形态特性:上皮细胞;相关产品有:293 Ad5细胞、KMS-18细胞、P3-NS1/1-Ag4-1细胞
OV-90 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:P3JHR-1细胞、RK-13细胞、OK-WT细胞
CaEs17 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:X63Ag8-653细胞、SU-DHL-4细胞、B16/BL6细胞
HNE2 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKN-AS细胞、Panc-05.04细胞、HPAEC细胞
HNE2 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKN-AS细胞、Panc-05.04细胞、HPAEC细胞
HPF Cells;背景说明:肺;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:2B4-L细胞、SVHUC细胞、Hepatoma-22细胞
HONE-1 Cells;背景说明:鼻咽癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PG-4 S+L-细胞、GM05372E细胞、H1522细胞
IPLB-SF 21AE Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKO3细胞、Intestinal Porcine Epithelial Cell line-J2细胞、HCC-2108细胞
VERO76 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AN3CA细胞、SCC090细胞、CAKI.1细胞
HLMVEC Cells;背景说明:肺微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MKN 7细胞、ML-2细胞、HPaSteC(HPSC)细胞
HN4 Cells;背景说明:喉鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LA4细胞、MDA361细胞、H-1618细胞
SKG-IIIa Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:IOSE80细胞、BC-019细胞、H-2196细胞
OCI/AML-5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:EC-GI-10细胞、253J-Bladder-V细胞、MHCC97-H细胞
RA Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:T47D细胞、NCIH2291细胞、MV522细胞
SMBCi019-A Cells(提供STR鉴定图谱)
CORL105 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HT-29细胞、Ontario Cancer Institute-Acute Myeloid Leukemia-3细胞、MDA-MB 361细胞
50.B1 Cells;背景说明:角膜上皮;Ad12-SV40;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TOV21G细胞、WIL2 S细胞、SW579细胞
SUD6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NCI-H1623细胞、RCS细胞、Walker-256-TC细胞
SU-DHL6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:HuT78细胞、NCI-H661细胞、IOSE-29细胞
TI-73 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Hep 3B2.1-7细胞、KS-SLK细胞、P3J.HR1K细胞
UMNSAH/DF-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:NCM-460细胞、NCIH1666细胞、H-1993细胞
OCI AML4 Cells;背景说明:急性髓系白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CCRF细胞、H-1770细胞、T-HEECs细胞
SKN-SH Cells;背景说明:SK-N-SH细胞系由J.L.Bieder建系,它与SK-N-MC所不同的是倍增时间较长且多巴胺-β-羟基酶水平较高。 SK-N-SH在细胞介导的细胞毒性试验中用作靶细胞系。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:上皮细胞样;相关产品有:Hs606细胞、GOS-3细胞、B16-F0细胞
H-196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HCC941122细胞、NCIH510细胞、X63Ag8-653细胞
373 MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OEC19细胞、A-875细胞、HRC-99细胞
P3J-HR-1 Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:HFF1细胞、Co320细胞、Statens Seruminstitut Rabbit Cornea细胞
NCI-H1437 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周换液2次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H2141细胞、C57 Mouse Tumor 93细胞、TW01细胞
293H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AR42J细胞、PC 61-5-3细胞、HFLS细胞
SP2 Cells;背景说明:该细胞是由绵羊红细胞免疫的BALB/c小鼠脾细胞和P3X63Ag8骨髓瘤细胞融合得到的。该细胞不分泌免疫球蛋白,对20μg/ml的8-氮鸟嘌呤有抗性,对HAT比较敏感;该细胞可以作为细胞融合时的B细胞组分用于制备杂交瘤;鼠痘病毒阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:RL95细胞、NCI-H2405细胞、NCIH1355细胞
COLO 205人结肠癌复苏细胞保种中心|带STR证书
BayGenomics ES cell line CSA089 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRS388 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA589 Cells(提供STR鉴定图谱)
IE-3 Cells(提供STR鉴定图谱)
PCRP-PRDM7-3D3 Cells(提供STR鉴定图谱)
KRC-7 [Rat hepatoma] Cells(提供STR鉴定图谱)
" "PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2041050; DOI=10.1093/jnci/83.11.757
Monks A., Scudiero D.A., Skehan P., Shoemaker R.H., Paull K.D., Vistica D.T., Hose C.D., Langley J., Cronise P., Vaigro-Wolff A., Gray-Goodrich M., Campbell H., Mayo J.G., Boyd M.R.
Feasibility of a high-flux anticancer drug screen using a diverse panel of cultured human tumor cell lines.
J. Natl. Cancer Inst. 83:757-766(1991)
PubMed=7651727
Kastrinakis W.V., Ramchurren N., Rieger K.M., Hess D.T., Loda M., Steele G., Summerhayes I.C.
Increased incidence of p53 mutations is associated with hepatic metastasis in colorectal neoplastic progression.
Oncogene 11:647-652(1995)
PubMed=9023415; DOI=10.1006/cimm.1996.1062
Seki N., Hoshino T., Kikuchi M., Hayashi A., Itoh K.
HLA-A locus-restricted and tumor-specific CTLs in tumor-infiltrating lymphocytes of patients with non-small cell lung cancer.
Cell. Immunol. 175:101-110(1997)
PubMed=9294210; DOI=10.1073/pnas.94.19.10330; PMCID=PMC23362
Ilyas M., Tomlinson I.P.M., Rowan A.J., Pignatelli M., Bodmer W.F.
Beta-catenin mutations in cell lines established from human colorectal cancers.
Proc. Natl. Acad. Sci. U.S.A. 94:10330-10334(1997)
PubMed=10051639; DOI=10.1073/pnas.96.5.2316; PMCID=PMC26781
Efstathiou J.A., Liu D., Wheeler J.M.D., Kim H.C., Beck N.E., Ilyas M., Karayiannakis A.J., Mortensen N.J., Kmiot W.A.W., Playford R.J., Pignatelli M., Bodmer W.F.
Mutated epithelial cadherin is associated with increased tumorigenicity and loss of adhesion and of responsiveness to the motogenic trefoil factor 2 in colon carcinoma cells.
Proc. Natl. Acad. Sci. U.S.A. 96:2316-2321(1999)
PubMed=10674020; DOI=10.1016/S0959-8049(99)00206-3
Ku J.-L., Yoon K.-A., Kim D.-Y., Park J.-G.
Mutations in hMSH6 alone are not sufficient to cause the microsatellite instability in colorectal cancer cell lines.
Eur. J. Cancer 35:1724-1729(1999)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=10737795; DOI=10.1073/pnas.97.7.3352; PMCID=PMC16243
Rowan A.J., Lamlum H., Ilyas M., Wheeler J.M.D., Straub J., Papadopoulou A., Bicknell D.C., Bodmer W.F., Tomlinson I.P.M.
APC mutations in sporadic colorectal tumors: a mutational 'hotspot' and interdependence of the 'two hits'.
Proc. Natl. Acad. Sci. U.S.A. 97:3352-3357(2000)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=12584437; DOI=10.1159/000068544
Melcher R., Koehler S., Steinlein C., Schmid M., Mueller C.R., Luehrs H., Menzel T., Scheppach W., Moerk H., Scheurlen M., Koehrle J., Al-Taie O.
Spectral karyotype analysis of colon cancer cell lines of the tumor suppressor and mutator pathway.
Cytogenet. Genome Res. 98:22-28(2002)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20570890; DOI=10.1158/0008-5472.CAN-10-0192; PMCID=PMC2943514
Janakiraman M., Vakiani E., Zeng Z.-S., Pratilas C.A., Taylor B.S., Chitale D., Halilovic E., Wilson M., Huberman K., Ricarte Filho J.C.M., Persaud Y., Levine D.A., Fagin J.A., Jhanwar S.C., Mariadason J.M., Lash A., Ladanyi M., Saltz L.B., Heguy A., Paty P.B., Solit D.B.
Genomic and biological characterization of exon 4 KRAS mutations in human cancer.
Cancer Res. 70:5901-5911(2010)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=23272949; DOI=10.1186/1755-8794-5-66; PMCID=PMC3543849
Schlicker A., Beran G., Chresta C.M., McWalter G., Pritchard A., Weston S., Runswick S., Davenport S., Heathcote K., Castro D.A., Orphanides G., French T., Wessels L.F.A.
Subtypes of primary colorectal tumors correlate with response to targeted treatment in colorectal cell lines.
BMC Med. Genomics 5:66.1-66.15(2012)
PubMed=23631600; DOI=10.1021/pr400260h
Loftus N.J., Lai L., Wilkinson R.W., Odedra R., Wilson I.D., Barnes A.J.
Global metabolite profiling of human colorectal cancer xenografts in mice using HPLC-MS/MS.
J. Proteome Res. 12:2980-2986(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=24755471; DOI=10.1158/0008-5472.CAN-14-0013
Mouradov D., Sloggett C., Jorissen R.N., Love C.G., Li S., Burgess A.W., Arango D., Strausberg R.L., Buchanan D., Wormald S., O'Connor L., Wilding J.L., Bicknell D.C., Tomlinson I.P.M., Bodmer W.F., Mariadason J.M., Sieber O.M.
Colorectal cancer cell lines are representative models of the main molecular subtypes of primary cancer.
Cancer Res. 74:3238-3247(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25841592; DOI=10.1016/j.jprot.2015.03.019
Piersma S.R., Knol J.C., de Reus I., Labots M., Sampadi B.K., Pham T.V., Ishihama Y., Verheul H.M.W., Jimenez C.R.
Feasibility of label-free phosphoproteomics and application to base-line signaling of colorectal cancer cell lines.
J. Proteomics 127:247-258(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25926053; DOI=10.1038/ncomms8002
Medico E., Russo M., Picco G., Cancelliere C., Valtorta E., Corti G., Buscarino M., Isella C., Lamba S., Martinoglio B., Veronese S., Siena S., Sartore-Bianchi A., Beccuti M., Mottolese M., Linnebacher M., Cordero F., Di Nicolantonio F., Bardelli A.
The molecular landscape of colorectal cancer cell lines unveils clinically actionable kinase targets.
Nat. Commun. 6:7002.1-7002.10(2015)
PubMed=25944804; DOI=10.1158/1078-0432.CCR-14-2457
Bazzocco S., Dopeso H., Carton-Garcia F., Macaya I., Andretta E., Chionh F., Rodrigues P., Garrido M., Alazzouzi H., Nieto R., Sanchez A., Schwartz S. Jr., Bilic J., Mariadason J.M., Arango D.
Highly expressed genes in rapidly proliferating tumor cells as new targets for colorectal cancer treatment.
Clin. Cancer Res. 21:3695-3704(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26537799; DOI=10.1074/mcp.M115.051235; PMCID=PMC4762531
Holst S., Deuss A.J.M., van Pelt G.W., van Vliet S.J., Garcia-Vallejo J.J., Koeleman C.A.M., Deelder A.M., Mesker W.E., Tollenaar R.A.E.M., Rombouts Y., Wuhrer M.
N-glycosylation profiling of colorectal cancer cell lines reveals association of fucosylation with differentiation and caudal type homebox 1 (CDX1)/villin mRNA expression.
Mol. Cell. Proteomics 15:124-140(2016)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=28192450; DOI=10.1371/journal.pone.0171435; PMCID=PMC5305277
Fasterius E., Raso C., Kennedy S.A., Rauch N., Lundin P., Kolch W., Uhlen M., Al-Khalili Szigyarto C.
A novel RNA sequencing data analysis method for cell line authentication.
PLoS ONE 12:E0171435-E0171435(2017)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28683746; DOI=10.1186/s12943-017-0691-y; PMCID=PMC5498998
Berg K.C.G., Eide P.W., Eilertsen I.A., Johannessen B., Bruun J., Danielsen S.A., Bjornslett M., Meza-Zepeda L.A., Eknaes M., Lind G.E., Myklebost O., Skotheim R.I., Sveen A., Lothe R.A.
Multi-omics of 34 colorectal cancer cell lines -- a resource for biomedical studies.
Mol. Cancer 16:116.1-116.16(2017)
PubMed=28854368; DOI=10.1016/j.celrep.2017.08.010; PMCID=PMC5583477
Roumeliotis T.I., Williams S.P., Goncalves E., Alsinet C., Del Castillo Velasco-Herrera M., Aben N., Ghavidel F.Z., Michaut M., Schubert M., Price S., Wright J.C., Yu L., Yang M., Dienstmann R., Guinney J.H., Beltrao P., Brazma A., Pardo M., Stegle O., Adams D.J., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Choudhary J.S.
Genomic determinants of protein abundance variation in colorectal cancer cells.
Cell Rep. 20:2201-2214(2017)
PubMed=29718670; DOI=10.1021/acs.jproteome.8b00165; PMCID=PMC6670293
Clark D.J., Hu Y.-W., Bocik W., Chen L.-J., Schnaubelt M., Roberts R.R., Shah P., Whiteley G.R., Zhang H.
Evaluation of NCI-7 cell line panel as a reference material for clinical proteomics.
J. Proteome Res. 17:2205-2215(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"