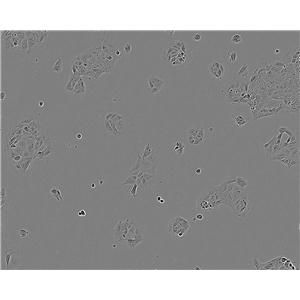
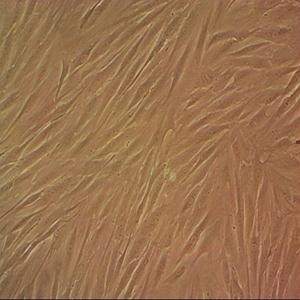
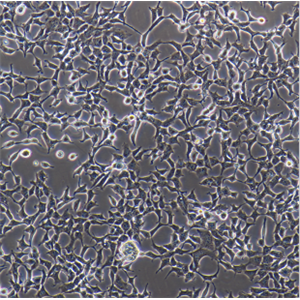
"BT-549人乳腺管癌复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞培养无菌操作步骤和注意事项:1.打开无菌工作台及净化室的紫外灯,消毒半小时以上。2.进入净化室前先关闭紫外灯,打开超净台风机,等待30min以上,以排尽臭氧。3.穿HAO隔离衣,戴HAO口罩,帽子。4.风淋2分钟。5.0.1%水泡手,擦干。6.点燃酒精灯;超净台内应避免放入过多的物品;使用的吸管,滴管,试管,培养瓶等均事先灭菌。7.打开各类瓶盖前先过火,以固定灰尘;打开的瓶口、试管口过火焰,镊子使用前应经火焰烧灼。8.水平式风机的超净台,应使瓶口斜置,应尽量避免瓶口敞开直立。9.同一根吸管或滴管不应连续用于几个不同的细胞系;吸取培养基的吸管应离开培养瓶或试管口0.5cm,避免伸入培养瓶口或试管口;以防止细胞系的互相混杂污染。10.漏在培养瓶上或台上的体,立即用酒精棉球擦净。11.操作完毕后恢复工作台面。
换液周期:每周2-3次
QGP1 Cells;背景说明:胰腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H220细胞、MT-2J细胞、LS-180细胞
HRA-19 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H441细胞、aTC1-6细胞、HL 60细胞
KASUMI1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:原粒细胞;相关产品有:9L细胞、Tca-8113细胞、NCI-H295R细胞
BT-549人乳腺管癌复苏细胞保种中心|带STR证书
背景信息:该细胞1978年由W.G.Coutinho和E.Y.Lasfargues建系,源自一位72岁患有乳腺导管癌的白人女性,来源组织包括乳头及浸润导管。该细胞形态包括上皮样细胞及多核巨细胞,可分泌一种粘性物质。
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
IR 983F Cells;背景说明:骨髓瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:MDA-MB-231 #4175细胞、Detroit-562细胞、HCT GEO细胞
SKNO-1 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HEC-151细胞、IEC-18细胞、C32 [Human melanoma]细胞
HTh74 Cells;背景说明:未分化甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1568细胞、KG1A细胞、KY-50细胞
JeKo 1 Cells;背景说明:一位套细胞淋巴瘤患者的巨细胞变种显示白血病转变,从其外周血单核细胞出发建立了MCL细胞株JeKo-1。 JeKo-1细胞EB病毒阴性,并表达一种B细胞表型的IgM。 细胞过表达cyclin D1, Bcl-2, c-Myc 及 Rb 蛋白。 Bcl-1/J(H)基因重排得到了PCR证实。 JeKo-1细胞在SCID小鼠中高成瘤。 [PubMed: 9753063];传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HPAF-2细胞、TE-8细胞、hOMF细胞
BT-549人乳腺管癌复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:上皮细胞样
【悬浮型细胞的传代操作】1)吸出细胞培养,放入离心管中,离心1000rpm 5分钟;2)吸掉上清,加入适量之新鲜培养基,混和均匀后,依稀释比例转移至新的培养瓶中,以正常培养条件培养。【注意事项】1)严格的无菌操作;2)适度消化:消化的时间受消化的种类、配制时间、加入培养瓶中的量等诸多因素的影响,消化过程中应该注意培养细胞形态的变化,一旦胞质回缩,连接变松散,或有成片浮起的迹象就要立即终止消化。附:EDA(0.02%四乙二钠)消化配方:EDA 0.20g,NaCl 8.00g,KCl 0.20g,KH2PO4 0.02g,葡萄糖 2.00g,0.5%红4ml,加入蒸馏水定容至1000ml。10磅20minGAO压灭菌,使用时调节PH值到7.4。注意EDA不能被血清中和,使用后培养瓶要彻底清洗,否则再培养时细胞容易脱壁。
HEK 293FT Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:CBRH7919细胞、HBE细胞、IAR 20细胞
BS-C-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:U87-MG细胞、RCC-4细胞、H-1693细胞
RCC 7860 Cells;背景说明:该细胞源自一位原发性肾透明细胞癌患者。该细胞有微绒毛和桥粒,能在软琼脂上生长。此细胞生成一种PTH(甲状旁腺素)样的多肽,与乳癌和肺癌中生成的肽相似,其N端序列与PTH相似,具有PTH样活性,分子量为6000D。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:COLO-205细胞、WEHI231细胞、CAKI 2细胞
OsA-CL Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TE11细胞、TE6细胞、NRK 52E细胞
K299 Cells;背景说明:间变性大细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CL34细胞、H-2347细胞、NK-92细胞
ARH 77 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:UMUC-14细胞、MDA-453细胞、HET1A细胞
NTERA-2 cl.D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM02219细胞、HMC3细胞、B16/F1细胞
H-2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:PK 15细胞、NuTu-19细胞、HCC-1569细胞
SKO3 Cells;背景说明:SK-OV-3由G.Trempe和L.J.Old在1973年从卵巢肿瘤病人的腹水分离得到。 此细胞对肿瘤坏死因子和几种细胞毒性药物包括白喉毒素、顺铂和阿霉素均耐受。 在裸鼠中致瘤,且形成与卵巢原位癌一致的中度分化的腺癌。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:143 B细胞、NCIH460细胞、CV-1 in Origin Simian-7细胞
H2073 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:253J B-V细胞、CSQT-2细胞、McA-RH8994细胞
P3X63 AG 8.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C33细胞、P-2003细胞、PANC-10-05细胞
Duke University 145 Cells;背景说明:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,酸性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:M619细胞、D-283 Med细胞、Japanese Tissue Culture-39细胞
WSU-DLCL-2 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:UWB1.289+BRCA1细胞、MuM-2C细胞、H.Ep.-2细胞
SUM102PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-920细胞、TE 354.T细胞、Chang liver细胞
4T1 Cells;背景说明:4T1是从410.4瘤株中未经诱变筛得的6-鸟嘌噙抗性细胞株。当注射到BALB/c小鼠中时,4T1自发产生高转移肿瘤,可转移到肺,肝,淋巴结和大脑,同时在注射部位形成始发灶。诱导转移时不需要摘除始发灶。4T1细胞在BALB/c小鼠中的生长与转移特性与人体中的乳腺癌十分相近。这种肿瘤是人VI期乳腺癌的动物模型。4T1-诱导的肿瘤在手术后及未手术情况下转移的动力学相近,可以用作手术后及未手术模型。跟其他肿瘤模型相比,由于4T1的抗6-鸟嘌噙特性,微小的转移细胞团(少到仅仅1个)也可以在许多远端器官中检测到。没必要数淋巴结或称重器官。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BSC40细胞、T173细胞、SK_HEP1细胞
DV90 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SRA01/04 (HLE)细胞、RenCa细胞、373 MG细胞
BC-025 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ramos G6.C10细胞、Human Microglia Clone 3细胞、NCI-H748细胞
BT-549人乳腺管癌复苏细胞保种中心|带STR证书
A-PTC Cells(提供STR鉴定图谱)
Abcam MCF-7 PGF KO Cells(提供STR鉴定图谱)
B'SYS CHO Nav1.5 Duo Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRO504 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHC043 Cells(提供STR鉴定图谱)
CE-1 Cells(提供STR鉴定图谱)
DA01205 Cells(提供STR鉴定图谱)
EURACi004-A Cells(提供STR鉴定图谱)
GM07321 Cells(提供STR鉴定图谱)
Karpas-299 Cells;背景说明:间变性大细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HCM细胞、SHIN-3细胞、Rat Lung Epithelial-6-T-antigen Negative细胞
SUM-52 Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:AG 9细胞、B16/F0细胞、HCC 2185细胞
Tb 1 Lu (NBL-12) Cells;背景说明:肺;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:VMM-5A细胞、SU-DHL-2细胞、RASMCs细胞
HFL 1 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:MIA Paca2细胞、Panc 04.03细胞、HCC-1937细胞
SF 539 Cells;背景说明:胶质瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU739细胞、HTR-8/SV-neo细胞、NH6细胞
KU 812 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代。3天内可长满;生长特性:悬浮生长;形态特性:骨髓母细胞;相关产品有:A9 (Hamprecht)细胞、SK-MEL-28细胞、SKUT-1细胞
AR58A314.1 Cells(提供STR鉴定图谱)
FU-MMT-1 Cells;背景说明:子宫肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KG-1a细胞、H-522细胞、SK-Mel 2细胞
IOSE 80 Cells;背景说明:卵巢;上皮细胞;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2330细胞、NALM-6细胞、FHC细胞
C28/I2 Cells;背景说明:软骨;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CHO/dhFr-细胞、Hos TE-85细胞、526 mel细胞
PANC0403 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:L-540细胞、RGE细胞、A704细胞
HEC-1A Cells;背景说明:这株细胞及其亚株HEC-1-B是H.Kuramoto及其同事1968年从一位IA期子宫内膜癌患者身上分离得到的。PAF可以诱导其c-fos的表达。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Oka-C1细胞、SK Col 1细胞、OEC33细胞
HT55 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SUM 159PT细胞、UM-UC-14细胞、ML-2细胞
H2330 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MeT-5A细胞、OEC19细胞、hTERTHME1细胞
G519 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI H747细胞、OVCAR-432细胞、GTL 16细胞
GM09911 Cells(提供STR鉴定图谱)
HAP1 BPTF (-) 2 Cells(提供STR鉴定图谱)
SL1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Cates-1B细胞、NCI-H2347细胞、TFK1细胞
Human Embryonic Kidney 293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:My-La 2059细胞、NKL细胞、NKL细胞
LN299 Cells;背景说明:胶质瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Rat1细胞、ROS 17/28细胞、EB-1细胞
CCD-1095Sk Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SCC 9细胞、hFOB 1.19细胞、HCO细胞
hEM15A Cells;背景说明:子宫内膜间质细胞;永生化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H2591细胞、U-251_MG细胞、M-14细胞
TE 32.T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:SF 767细胞、WEHI-3B细胞、C1498细胞
LNCaP subline C4-2 Cells;背景说明:前列腺癌;左锁骨上淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Swiss3T3细胞、SK_HEP1细胞、CHO/dhFr-细胞
SUPB15 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SN12C-PM6细胞、HRPEpiC细胞、A 2058细胞
HG03368 Cells(提供STR鉴定图谱)
ICRmt-1 Cells(提供STR鉴定图谱)
LNCaP-CS10 Cells(提供STR鉴定图谱)
NCI-H3030 Cells(提供STR鉴定图谱)
PathHunter DLD1 MTNR1A Total GPCR Internalization Cells(提供STR鉴定图谱)
Ubigene HEK293 S1PR3 KO Cells(提供STR鉴定图谱)
WIBRe001-A-35 Cells(提供STR鉴定图谱)
HG01527 Cells(提供STR鉴定图谱)
HuTu-80 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Alpha Mouse Liver 12细胞、3T3-L1 ad细胞、Vero from pool #76细胞
Colo699 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-2330细胞、H-661细胞、HCT116/L细胞
M109 Cells;背景说明:肺癌;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JAR细胞、RGC5细胞、Hs895.T细胞
P3/NSI/1-AG4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SW 1088细胞、Alexander细胞、CAL-148细胞
RM1 Cells;背景说明:前列腺癌;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH208细胞、RAMSCs细胞、CFPAC-1细胞
OVCA433 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:EC-9706细胞、JCA-1细胞、NCI-H2029细胞
8305-C Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CDC/EU.HMEC-1细胞、TGHAVSMC细胞、MCF12F细胞
Rca-B Cells;背景说明:鳞癌;SD大鼠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GT39细胞、MDA-MB 231细胞、NCI-H1792细胞
CRFK Cells;背景说明:肾;自发永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TCC-PAN2细胞、JVM3细胞、NCI.H522细胞
TMK-1 Cells;背景说明:胃癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MGHU3细胞、SF 295细胞、BOWES细胞
OVCAR 5 Cells;背景说明:卵巢癌;腹水转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Nb-2细胞、MTC-TT细胞、SW527细胞
OVCAR3 Cells;背景说明:该细胞1982年由T.C. Hamilton等建系,源自一位60卵巢腺癌的腹水,是卵巢癌抗药性研究的模型。;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BLO-11细胞、SNB.19细胞、NCI-H711细胞
Hs822T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BL6细胞、HR-1细胞、HLE细胞
HKBML Cells;背景说明:脑;淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HL-1细胞、8226细胞、SW 962细胞
CaSki Cells;背景说明:这株细胞是从小肠肠系膜转移灶的细胞中建立的。 据报道,它含有完整的HPV-16(每个细胞大约600个拷贝)和HPV-18相关序列。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HSF2细胞、EL 4细胞、KMS26细胞
SaOS-1 Cells(提供STR鉴定图谱)
Hs729 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Panc 08.13细胞、HONE1细胞、MC3T3-E细胞
SK-Mel 1 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在1×105-2×105,每周补液2-3次。;生长特性:悬浮生长;形态特性:球形的;相关产品有:Hs 683细胞、KYSE 140细胞、Murine Leydig Tumor Cell line-1细胞
HCC-9724 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LN299细胞、L-6 myoblast细胞、697细胞
JVM3 Cells;背景说明:慢性髓白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI-SNU-761细胞、MDA-MB-231细胞、SH-SY5Y细胞
GM-215 Cells;背景说明:肺;自发永生;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2286细胞、KP-N-YN细胞、MonoMac 6细胞
HCMEC(BL12-H) Cells;背景说明:结肠微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR10细胞、NCI-H1404细胞、AMO-1细胞
B16 F1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:JROECL33细胞、H-1703细胞、LC-MS细胞
U266B1 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:OV433细胞、EO771细胞、OV1063细胞
S3-HeLa Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hi-five细胞、RPTEC/TERT 1细胞、HNTEC细胞
LA 4 Cells;背景说明:肺癌;A/He;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:T/G HA-VSMC细胞、CHO细胞、H-1781细胞
SUM 102 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Swiss-3T3细胞、H647ell细胞、RPMI8226细胞
PANC-08-13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SUM102细胞、CCD19Lu细胞、LL/2 (LLC1)细胞
OCI-AML-3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs 606.T细胞、H4细胞、KYSE 70细胞
MRAEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Rat Basophilic Leukemia-1细胞、H-35细胞、COV-362细胞
BT-549人乳腺管癌复苏细胞保种中心|带STR证书
BayGenomics ES cell line RRD163 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XF065 Cells(提供STR鉴定图谱)
DBA A.Sp Cells(提供STR鉴定图谱)
MLB13MYC clone 17 Cells(提供STR鉴定图谱)
T3-3H1 Cells(提供STR鉴定图谱)
LUEi005-B Cells(提供STR鉴定图谱)
" "PubMed=7902062
de la Torre M., Hao X.-Y., Larsson R., Nygren P., Tsuruo T., Mannervik B., Bergh J.
Characterization of four doxorubicin adapted human breast cancer cell lines with respect to chemotherapeutic drug sensitivity, drug resistance associated membrane proteins and glutathione transferases.
Anticancer Res. 13:1425-1430(1993)
DOI=10.1016/B978-0-12-333530-2.50009-5
Leibovitz A.
Cell lines from human breast.
(In book chapter) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.161-184; Academic Press; New York; USA (1994)
PubMed=21552935; DOI=10.3892/ijo.7.5.1079
Nangia-Makker P., Thompson E., Hogan C., Ochieng J., Raz A.
Induction of tumorigenicity by galectin-3 in a nontumorigenic human breast-carcinoma cell-line.
Int. J. Oncol. 7:1079-1087(1995)
PubMed=9561029
Warfield P.R., Nangia-Makker P., Raz A., Ochieng J.
Adhesion of human breast carcinoma to extracellular matrix proteins is modulated by galectin-3.
Invasion Metastasis 17:101-112(1997)
PubMed=9671407; DOI=10.1038/sj.onc.1201814
Sweeney K.J., Swarbrick A., Sutherland R.L., Musgrove E.A.
Lack of relationship between CDK activity and G1 cyclin expression in breast cancer cells.
Oncogene 16:2865-2878(1998)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=10862037; DOI=10.1002/1098-2264(200007)28:3<308::AID-GCC9>3.0.CO;2-B
Kytola S., Rummukainen J., Nordgren A., Karhu R., Farnebo F., Isola J.J., Larsson C.
Chromosomal alterations in 15 breast cancer cell lines by comparative genomic hybridization and spectral karyotyping.
Genes Chromosomes Cancer 28:308-317(2000)
PubMed=10969801
Forozan F., Mahlamaki E.H., Monni O., Chen Y.-D., Veldman R., Jiang Y., Gooden G.C., Ethier S.P., Kallioniemi A.H., Kallioniemi O.-P.
Comparative genomic hybridization analysis of 38 breast cancer cell lines: a basis for interpreting complementary DNA microarray data.
Cancer Res. 60:4519-4525(2000)
PubMed=11343771; DOI=10.1016/S0165-4608(00)00387-3
Rummukainen J., Kytola S., Karhu R., Farnebo F., Larsson C., Isola J.J.
Aberrations of chromosome 8 in 16 breast cancer cell lines by comparative genomic hybridization, fluorescence in situ hybridization, and spectral karyotyping.
Cancer Genet. Cytogenet. 126:1-7(2001)
PubMed=15153330; DOI=10.1593/neo.3292; PMCID=PMC1502105
Watts G.S., Oshiro M.M., Junk D.J., Wozniak R.J., Watterson S.J., Domann F.E., Futscher B.W.
The acetyltransferase p300/CBP-associated factor is a p53 target gene in breast tumor cells.
Neoplasia 6:187-194(2004)
PubMed=15677628; DOI=10.1093/carcin/bgi032
Gorringe K.L., Chin S.-F., Pharoah P.D.P., Staines J.M., Oliveira C., Edwards P.A.W., Caldas C.
Evidence that both genetic instability and selection contribute to the accumulation of chromosome alterations in cancer.
Carcinogenesis 26:923-930(2005)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=16397213; DOI=10.1158/0008-5472.CAN-05-2853
Elstrodt F., Hollestelle A., Nagel J.H.A., Gorin M., Wasielewski M., van den Ouweland A.M.W., Merajver S.D., Ethier S.P., Schutte M.
BRCA1 mutation analysis of 41 human breast cancer cell lines reveals three new deleterious mutants.
Cancer Res. 66:41-45(2006)
PubMed=16541312; DOI=10.1007/s10549-006-9186-z
Wasielewski M., Elstrodt F., Klijn J.G.M., Berns E.M.J.J., Schutte M.
Thirteen new p53 gene mutants identified among 41 human breast cancer cell lines.
Breast Cancer Res. Treat. 99:97-101(2006)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=17157791; DOI=10.1016/j.ccr.2006.10.008; PMCID=PMC2730521
Neve R.M., Chin K., Fridlyand J., Yeh J., Baehner F.L., Fevr T., Clark L., Bayani N., Coppe J.-P., Tong F., Speed T., Spellman P.T., DeVries S., Lapuk A., Wang N.J., Kuo W.-L., Stilwell J.L., Pinkel D., Albertson D.G., Waldman F.M., McCormick F., Dickson R.B., Johnson M.D., Lippman M.E., Ethier S.P., Gazdar A.F., Gray J.W.
A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes.
Cancer Cell 10:515-527(2006)
PubMed=18516279; DOI=10.1016/j.molonc.2007.02.004; PMCID=PMC2391005
Kenny P.A., Lee G.Y., Myers C.A., Neve R.M., Semeiks J.R., Spellman P.T., Lorenz K., Lee E.H., Barcellos-Hoff M.H., Petersen O.W., Gray J.W., Bissell M.J.
The morphologies of breast cancer cell lines in three-dimensional assays correlate with their profiles of gene expression.
Mol. Oncol. 1:84-96(2007)
PubMed=18277095; DOI=10.4161/cbt.7.5.5712
Berglind H., Pawitan Y., Kato S., Ishioka C., Soussi T.
Analysis of p53 mutation status in human cancer cell lines: a paradigm for cell line cross-contamination.
Cancer Biol. Ther. 7:699-708(2008)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=19582160; DOI=10.1371/journal.pone.0006146; PMCID=PMC2702084
Kao J., Salari K., Bocanegra M., Choi Y.-L., Girard L., Gandhi J., Kwei K.A., Hernandez-Boussard T., Wang P., Gazdar A.F., Minna J.D., Pollack J.R.
Molecular profiling of breast cancer cell lines defines relevant tumor models and provides a resource for cancer gene discovery.
PLoS ONE 4:E6146-E6146(2009)
PubMed=19727395; DOI=10.1371/journal.pone.0006888; PMCID=PMC2731225
Wadlow R.C., Wittner B.S., Finley S.A., Bergquist H., Upadhyay R., Finn S.P., Loda M., Mahmood U., Ramaswamy S.
Systems-level modeling of cancer-fibroblast interaction.
PLoS ONE 4:E6888-E6888(2009)
DOI=10.25904/1912/1434
Morrison B.J.
Breast cancer stem cells: tumourspheres and implications for therapy.
Thesis PhD (2010); Griffith University; Brisbane; Australia
PubMed=19593635; DOI=10.1007/s10549-009-0460-8
Hollestelle A., Nagel J.H.A., Smid M., Lam S., Elstrodt F., Wasielewski M., Ng S.S., French P.J., Peeters J.K., Rozendaal M.J., Riaz M., Koopman D.G., ten Hagen T.L.M., de Leeuw B.H.C.G.M., Zwarthoff E.C., Teunisse A.F.A.S., van der Spek P.J., Klijn J.G.M., Dinjens W.N.M., Ethier S.P., Clevers H.C., Jochemsen A.G., den Bakker M.A., Foekens J.A., Martens J.W.M., Schutte M.
Distinct gene mutation profiles among luminal-type and basal-type breast cancer cell lines.
Breast Cancer Res. Treat. 121:53-64(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20679594; DOI=10.1093/jnci/djq279; PMCID=PMC2935474
Gazdar A.F., Girard L., Lockwood W.W., Lam W.L., Minna J.D.
Lung cancer cell lines as tools for biomedical discovery and research.
J. Natl. Cancer Inst. 102:1310-1321(2010)
PubMed=21778573; DOI=10.3233/BD-2010-0307; PMCID=PMC3532890
Chavez K.J., Garimella S.V., Lipkowitz S.
Triple negative breast cancer cell lines: one tool in the search for better treatment of triple negative breast cancer.
Breast Dis. 32:35-48(2010)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=23151021; DOI=10.1186/1471-2164-13-619; PMCID=PMC3546428
Grigoriadis A., Mackay A., Noel E., Wu P.-J., Natrajan R., Frankum J., Reis-Filho J.S., Tutt A.
Molecular characterisation of cell line models for triple-negative breast cancers.
BMC Genomics 13:619.1-619.14(2012)
PubMed=23601657; DOI=10.1186/bcr3415; PMCID=PMC3672661
Riaz M., van Jaarsveld M.T.M., Hollestelle A., Prager-van der Smissen W.J.C., Heine A.A.J., Boersma A.W.M., Liu J.-J., Helmijr J.C.A., Ozturk B., Smid M., Wiemer E.A.C., Foekens J.A., Martens J.W.M.
miRNA expression profiling of 51 human breast cancer cell lines reveals subtype and driver mutation-specific miRNAs.
Breast Cancer Res. 15:R33.1-R33.17(2013)
PubMed=23637631; DOI=10.1371/journal.pgen.1003464; PMCID=PMC3636093
Giacomini C.P., Sun S., Varma S., Shain A.H., Giacomini M.M., Balagtas J.M.S., Sweeney R.T., Lai E., Del Vecchio C.A., Forster A.D., Clarke N., Montgomery K.D., Zhu S., Wong A.J., van de Rijn M., West R.B., Pollack J.R.
Breakpoint analysis of transcriptional and genomic profiles uncovers novel gene fusions spanning multiple human cancer types.
PLoS Genet. 9:E1003464-E1003464(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24094812; DOI=10.1016/j.ccr.2013.08.020; PMCID=PMC3931310
Timmerman L.A., Holton T., Yuneva M., Louie R.J., Padro M., Daemen A., Hu M., Chan D.A., Ethier S.P., van 't Veer L.J., Polyak K., McCormick F., Gray J.W.
Glutamine sensitivity analysis identifies the xCT antiporter as a common triple-negative breast tumor therapeutic target.
Cancer Cell 24:450-465(2013)
PubMed=24162158; DOI=10.1007/s10549-013-2743-3; PMCID=PMC3832776
Prat A., Karginova O., Parker J.S., Fan C., He X.-P., Bixby L.M., Harrell J.C., Roman E., Adamo B., Troester M.A., Perou C.M.
Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes.
Breast Cancer Res. Treat. 142:237-255(2013)
PubMed=24176112; DOI=10.1186/gb-2013-14-10-r110; PMCID=PMC3937590
Daemen A., Griffith O.L., Heiser L.M., Wang N.J., Enache O.M., Sanborn Z., Pepin F., Durinck S., Korkola J.E., Griffith M., Hur J.S., Huh N., Chung J., Cope L., Fackler M.J., Umbricht C.B., Sukumar S., Seth P., Sume V.P., Jakkula L.R., Lu Y.-L., Mills G.B., Cho R.J., Collisson E.A., van 't Veer L.J., Spellman P.T., Gray J.W.
Modeling precision treatment of breast cancer.
Genome Biol. 14:R110.1-R110.14(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25892236; DOI=10.1016/j.celrep.2015.03.050; PMCID=PMC4425736
Lawrence R.T., Perez E.M., Hernandez D., Miller C.P., Haas K.M., Irie H.Y., Lee S.-I., Blau C.A., Villen J.
The proteomic landscape of triple-negative breast cancer.
Cell Rep. 11:630-644(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28287265; DOI=10.1021/acs.jproteome.6b00470; PMCID=PMC5557415
Yen T.-Y., Bowen S., Yen R., Piryatinska A., Macher B.A., Timpe L.C.
Glycoproteins in claudin-low breast cancer cell lines have a unique expression profile.
J. Proteome Res. 16:1391-1400(2017)
PubMed=28889351; DOI=10.1007/s10549-017-4496-x
Saunus J.M., Smart C.E., Kutasovic J.R., Johnston R.L., Kalita-de Croft P., Miranda M., Rozali E.N., Vargas A.C., Reid L.E., Lorsy E., Cocciardi S., Seidens T., McCart Reed A.E., Dalley A.J., Wockner L.F., Johnson J., Sarkar D., Askarian-Amiri M.E., Simpson P.T., Khanna K.K., Chenevix-Trench G., Al-Ejeh F., Lakhani S.R.
Multidimensional phenotyping of breast cancer cell lines to guide preclinical research.
Breast Cancer Res. Treat. 167:289-301(2018)
PubMed=30613774; DOI=10.1126/sciadv.aau7314; PMCID=PMC6314821
Vande Voorde J., Ackermann T., Pfetzer N., Sumpton D., Mackay G., Kalna G., Nixon C., Blyth K., Gottlieb E., Tardito S.
Improving the metabolic fidelity of cancer models with a physiological cell culture medium.
Sci. Adv. 5:eaau7314.1-eaau7314.14(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"