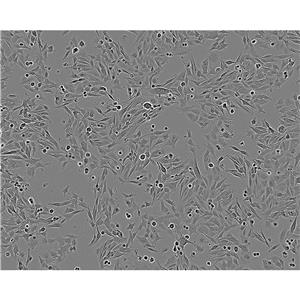
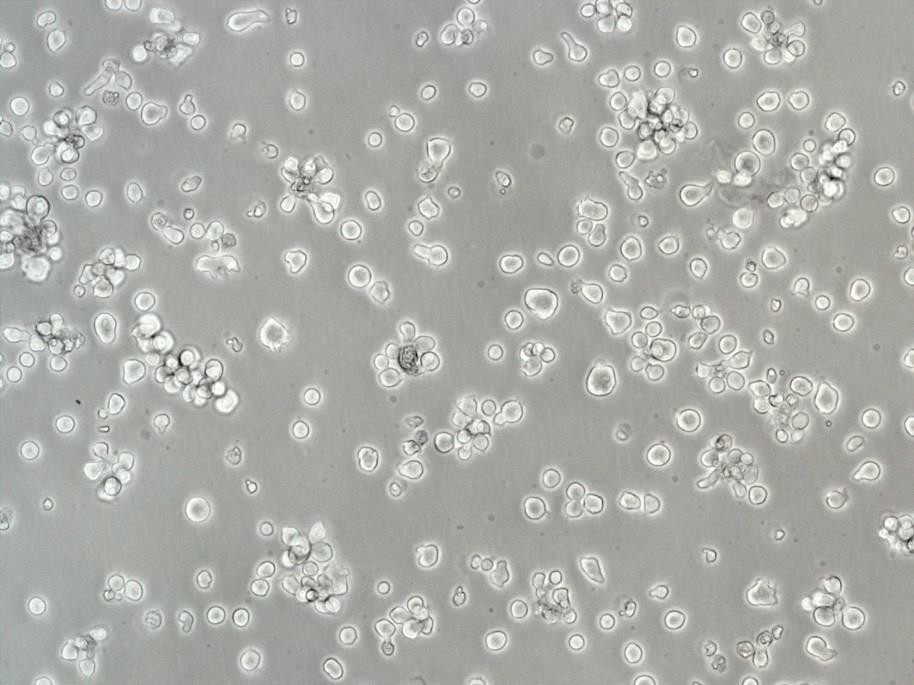
"A-375人恶性黑色素瘤复苏细胞保种中心|带STR证书
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
正确的细胞复苏需知事项:细胞冻存HAO了,接下来要注意什么问题呢?没错,就是记得到时间了,拿出来复苏。那么,细胞复苏的过程中又有哪些该注意的事项呢?细胞活力和形态检查的作用何在?活力检查——千万不要使用不健康的细胞,可能有污染(真菌、支原体等),如果发现有污染毫不犹豫的丢弃!形态检查——检查细胞的固有形态和生长行为。冻存细胞:补充新的培养——在您开始冻存细胞的前一天补充新的培养。在细胞长至70%单层时收获细胞,计数活细胞数,用冻存调整细胞密度~5 x106 s/ml (根据不同的细胞类型调整);冻存——用冻存洗细胞并用冻存重悬细胞,有不同类型的冻存,根据细胞类型选择Zui合适的冻存(常用的冻存成分有):5-10% DMSO——注意确保DMSO不含有其他的毒性物质;5-15%甘油;如果细胞在无血清培养基内生长,应在50%条件培养基内(细胞在无血清培养基内生长24小时)内冻存和复苏。在冻存管上标记HAO细胞类型,日期,冻存人等信息,并保证每冻存管不超过1.5ml。放入罐之前记录冻存管的数量和位置。以Zui快的速度转移冻存管知罐内,因此,此步骤ZuiHAO使用干冰,或者把冻存管浸入装有的小盒内。此外还要注意,在冻存管上没有足够的空间记录细胞的详细信息,做HAO记录是非常非常重要的!还有一个Zui重要的,一定要在异地的罐内保存同样的一份细胞,以免其中的一个罐出现问题!细胞正确的复苏方式和正确的冻存方式同样重要,熟记以下要点:当从罐内取出细胞时,有可能会出现冻存管破裂的情况,使用保护面罩和防护服十分必要;其实,细胞复苏只是一个简单的实验,不过这其中却不可避免有一些需要注意的细节,不然,也不一定会尽如人意。例如说,人身健康方面:一定要记得做HAO防冻工作,戴上护目镜;尽量降低DMSO对细胞的损伤等等。
换液周期:每周2-3次
KMS18 Cells;背景说明:浆细胞骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:NS20-Y细胞、UCLA NPA-87-1细胞、JHH-7细胞
RPMI8226/S Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HFL-1细胞、SMA-560细胞、BEL7404细胞
HC11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MM6细胞、HEC-251细胞、NUGC4细胞
A-375人恶性黑色素瘤复苏细胞保种中心|带STR证书
背景信息:A375源自一位54岁女性,是Giard DJ等人建立的一系列细胞株中的一株。该细胞可在免疫抑制小鼠上成瘤,在琼脂上形成克隆。
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
C-6 Cells;背景说明:胶质细胞株C6是由Benda等用N-nitrosomethylurea诱导的大鼠胶质瘤克隆,并经过一系列的体外培养和动物传代交替后建成的。 当细胞从低密度生长到满瓶时,S-100产量增加10倍。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:MRC9细胞、JIMT细胞、GM-637细胞
GP2d Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CCD19Lu细胞、GM2131细胞、GM637细胞
NCI-H1693 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MARC 145细胞、RCK8细胞、Hs888Lu细胞
SK-N-BE(1) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AZ-521细胞、NOR-10细胞、RAMOS2G64C10细胞
A-375人恶性黑色素瘤复苏细胞保种中心|带STR证书
物种来源:人源、鼠源等其它物种来源
形态特性:上皮细胞样
我常看到一个比较复杂的四步消化法,这个挺有意思,我也是第一次听说,应该是网友自己发展出来的方法,很有参考价值。但我估计这个方法可能对付少数非常怪异的细胞才需要这么复杂的程序。我目前养过的近300株细胞里面,还没遇到这么怪异的。比如Caco2其实是很HAO消化的细胞,不需要胰酶孵育即可,只是有点成片分布。不要以为成片分布是自己没养HAO,这个视细胞而定。如果和标准形态不一致,那可能是自己没消化HAO导致的,但如果消化方法正确,仍然成片分布,甚至完全吹打成单细胞悬,贴壁后仍然成片分布,这是细胞的性,是因为贴壁过程中重新聚集了。这个时候你拼了命要去让它均匀分布,你的细胞之后会对你越来越不HAO;比较难消化的细胞(润洗方法5min还不能消化),就以colon canr为例,比如HC15, LS411和KM12,这样的细胞一般需要用少量胰酶孵育,但也不需要很多,一般100 mm dish,一次Zui多加入500ul,就足够了,一般我加300ul。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。一些正常细胞也会有难消化的时候,比如sDC细胞,但也只要用少量胰酶孵育,3min左右即可看到成片沙状移动。
B16BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEI193细胞、Rat Basophilic Leukemia-1细胞、MSF细胞
NCIH1963 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:C1498细胞、NR-8383细胞、ES2细胞
UMNSAH-DF 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:EOL1细胞、SK.MEL.2细胞、H2052_MM细胞
CX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MIA-Pa-Ca-2细胞、BV-2细胞、GM03320细胞
QGP-1 Cells;背景说明:胰腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BALB 3T3 clone A31细胞、HSC-1细胞、JB6 Cl 30细胞
SKNO-1 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HEC-151细胞、IEC-18细胞、C32 [Human melanoma]细胞
CL1-5 Cells;背景说明:肺腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HuH-1细胞、HEp-2细胞、SW 626细胞
KMS-18 Cells;背景说明:浆细胞骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:MALME.3M细胞、TCam 2细胞、HH [Human lymphoma]细胞
KMB17 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Verda reno细胞、CNE-2Z细胞、801-D细胞
SF 767 Cells;背景说明:脑瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TSUpr1细胞、ESC细胞、PE/CA-PJ-34细胞
LLC-MK-2 Cells;背景说明:胚胎;肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A-9细胞、SN12CPM6细胞、NCI-H1734细胞
CX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MIA-Pa-Ca-2细胞、BV-2细胞、GM03320细胞
MAVER-1 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代;2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:AML-193细胞、NCI-H2170细胞、SNU-520细胞
ANA1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:C-127细胞、KBM7细胞、H-2081细胞
SKMEL-24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:MDA-134细胞、AE 1201细胞、MV4II细胞
ESC-410 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HOS-143B细胞、FOXNY细胞、HANK-1细胞
NRK clone 52E Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:PBL细胞、ARO81细胞、NIH:OVCAR-5细胞
A-375人恶性黑色素瘤复苏细胞保种中心|带STR证书
1321N1-UCP2-KO-c5 Cells(提供STR鉴定图谱)
Abcam HeLa DNMT3A KO Cells(提供STR鉴定图谱)
AG19507 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRF083 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG260 Cells(提供STR鉴定图谱)
C0108 Cells(提供STR鉴定图谱)
CW10144 Cells(提供STR鉴定图谱)
DA04948 Cells(提供STR鉴定图谱)
FY-hES-31 Cells(提供STR鉴定图谱)
LTEP-sm 1 Cells;背景说明:小细胞肺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Mono-Mac-6细胞、UPCI-SCC-090细胞、SNU-475细胞
MC116 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:Hs839.T细胞、SCaBER细胞、IHHA-1细胞
SUDHL-16 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:F-81细胞、Porcine Kidney-13细胞、NCIH1781细胞
Baby Hamster Kidney from litter No. 21 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代,每周换液1-2次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:PCI-4B细胞、KATO III细胞、GFP-Olig2细胞
MDCC-MSB1 Cells;背景说明:淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:GA10细胞、CHO Lec1细胞、KE-37细胞
H-295R Cells;背景说明:肾上腺皮质癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU-182细胞、HCMEC细胞、UCH1细胞
140-1 Cells(提供STR鉴定图谱)
NKM1 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:ECC 10细胞、RD-2细胞、766T细胞
BJA-B-1 Cells;背景说明:Burkitt's淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:GES1细胞、OVCAR-10细胞、SLMT-1细胞
DHL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KS-1 [Human glioblastoma]细胞、Human Microvascular Endothelial Cell line-1细胞、HCC0070细胞
CTSC-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HFF细胞、Nb2-11细胞、SU-DHL-5细胞
Jurkat Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:Pt-K1细胞、ACC2细胞、mRMEC细胞
L-cells Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:UPCI-SCC-090细胞、HCT 116细胞、Hopkins-92细胞
IOSE 80 Cells;背景说明:卵巢;上皮细胞;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2330细胞、NALM-6细胞、FHC细胞
RT-112 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C32-mel细胞、HeLa/DDP细胞、RCC10细胞
GM14311 Cells(提供STR鉴定图谱)
HAP1 GGA1 (-) 5 Cells(提供STR鉴定图谱)
HSC6 Cells;背景说明:口腔鳞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HOP 92细胞、Dami细胞、SNU620细胞
EL4 Cells;背景说明:EL4是从用9,10-二甲基-1,2-并蒽在C57BL小鼠中诱导的淋巴瘤中建立的。 能抗0.1 mM 化可的松,对20 mcg/ml PHA敏感。 还有一个亚株(EL4.IL-2, ATCC TIB-181)可以生成高水平的IL-2。 检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:MDCK.2细胞、SK-MEL3细胞、SV40-HCEC细胞
GEO Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1373细胞、BSC-40细胞、OCI-LY-7细胞
TGHAVSMC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC70细胞、Hs839.T细胞、Raji细胞
16HBE14o- Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Panc 3.27细胞、SGC7901细胞、HeLa/S3细胞
KCL-22 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Central Adrenergic TH-expressing a细胞、MKN28细胞、SaOS细胞
HT-29 Cells;背景说明:该细胞是1964年由FoghJ用移植培养方法和含15%FBS的F12培养液从原发性肿瘤分离的。近来,已建株的培养细胞用含血清的McCoy's5a培养基培养。该细胞系在裸鼠中成瘤,也能在类固醇处理的地鼠中成瘤。该细胞可合成IgA、CEA、TGFβ结合蛋白和黏液素;表达尿激酶受体,但没有检测到血浆酶原活性;不表达CD4,但细胞表面表达半乳糖神经酰胺(HIV的可能替代受体)。该细胞系癌基因c-myc、K-ras、H-ras、N-ras、Myb、sis、fos阳性;p53基因过表达,并且在273位密码子处发;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HS578细胞、HBL-1 [Human diffuse large B-cell lymphoma]细胞、HcerEpic细胞
BEL/FU Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KOSC-2 cl3-43细胞、HBL1细胞、RT-BM细胞
HNO124 Cells(提供STR鉴定图谱)
J-87 Cells(提供STR鉴定图谱)
MCF10A_MTAP_15 Cells(提供STR鉴定图谱)
ND15074 Cells(提供STR鉴定图谱)
PR00124 Cells(提供STR鉴定图谱)
Ubigene A-549 STAT5A KO Cells(提供STR鉴定图谱)
v39.7 Cells(提供STR鉴定图谱)
HAP1 SNX10 (-) 1 Cells(提供STR鉴定图谱)
S3-HeLa Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hi-five细胞、RPTEC/TERT 1细胞、HNTEC细胞
ZYM-DIEC02 Cells;背景说明:小肠上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HPaSteC(HPSC)细胞、RPPVEC细胞、NL20SV细胞
MCA-38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:MILE SVEN 1细胞、MOLT 3细胞、OCI Ly10细胞
SK-N-AS Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:FDCP-1细胞、NCI N87细胞、CCRF细胞
CW-2 Cells;背景说明:来源于结肠癌。 CEA阳性,移植于裸鼠可成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCIH2066细胞、C-4 I细胞、Rat Chondrosarcoma Swarm细胞
CW-2 Cells;背景说明:来源于结肠癌。 CEA阳性,移植于裸鼠可成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCIH2066细胞、C-4 I细胞、Rat Chondrosarcoma Swarm细胞
SPC-A1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H548细胞、NCIH1793细胞、HMEC-1细胞
HMy2.CIR Cells;背景说明:B细胞;EBV转染;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:P3X63 Ag8.653细胞、OCI/AML3细胞、MC-38细胞
no.11 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCE-2 [50.B1]细胞、THC-8307细胞、HCA-7细胞
NCI-H2087 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:悬浮生长,有少数细胞疏松贴壁;形态特性:上皮样;相关产品有:SW962细胞、2B4细胞、KYSE30细胞
D 407 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HCT 15细胞、293 HEK细胞、ECC12细胞
CX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MIA-Pa-Ca-2细胞、BV-2细胞、GM03320细胞
SF763 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H510A细胞、MONO-MAC 6细胞、PBMC细胞
NCTC-1469 Cells;背景说明:1952年建系,源于正常C3H/An小鼠的肝脏组织,表达H-2K抗原,鼠痘病毒阴性。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:淋巴母细胞;相关产品有:COLO-357细胞、Jeko1细胞、FRhK4细胞
NTERA2D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MARC145细胞、MGH-U3细胞、H2195细胞
SK-BR-3-Luc [JCRB] Cells(提供STR鉴定图谱)
NR 8383 Cells;背景说明:NR8383(正常大鼠,1983年8月3日)来源于肺灌洗时的正常大鼠肺泡巨噬细胞。细胞在gerbil肺细胞连续培养液存在下培养了大约8-9个月。随后,不再需要外源生长因子。通过有限稀释法从单个细胞克隆并亚克隆NR8383细胞,并三次用软琼脂亚克隆。细胞表现出巨噬细胞的特性,吞噬酵母多糖和铜绿,非特异性脂酶活性,Fc受体,氧化降解;分泌IL-1,TNFbeta和IL-6,可重复地响应外源生长因子。NR8383细胞响应博莱霉素,分泌TGFbeta前体。在博莱霉素刺激下,TGFbe;传代方法:1:2传代;生长特性:半贴壁生长;形态特性:巨噬细胞;相关产品有:RH-30细胞、SF 539细胞、PLC8024细胞
HUC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NCI-H187细胞、Astrocyte type I clone细胞、TE-12细胞
MDA 231-LM2-4175 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:University of Michigan-Urothelial Carcinoma-14细胞、Ca761细胞、H719细胞
Hs822T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BL6细胞、HR-1细胞、HLE细胞
TO 175.T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:RAW264.7细胞、HCC70细胞、SCL2细胞
P3X63Ag8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEK293A细胞、SCC-7细胞、SNU407细胞
SUM-159-PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Sp2/0-Ag-14细胞、SW1271细胞、OVCAR4细胞
NCIH69 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:悬浮生长,聚团;形态特性:聚团悬浮;相关产品有:RD 2细胞、H-1963细胞、ASPC1细胞
Panc-8_13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HUV-EC-C细胞、KMST6细胞、NCIH838细胞
ARO-81 Cells;背景说明:甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CT-26细胞、Tj-905细胞、A204细胞
CBRH-7919 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MH-S细胞、Colo16细胞、REC1细胞
3AO Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:TH1细胞、MHCC-LM3细胞、NCI-H102细胞
Hs27 Cells;背景说明:包皮;成纤维细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs742T细胞、Hep2细胞、Earles's cells细胞
OV90 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:P30-OHK细胞、Michigan Cancer Foundation-12A细胞、Kit225细胞
A-375人恶性黑色素瘤复苏细胞保种中心|带STR证书
BayGenomics ES cell line CSJ176 Cells(提供STR鉴定图谱)
BayGenomics ES cell line TEA030 Cells(提供STR鉴定图谱)
CCRC-M12 Cells(提供STR鉴定图谱)
M14-11 Cells(提供STR鉴定图谱)
RG7/11.1 Cells(提供STR鉴定图谱)
SACK-Xs Lig-13 Cells(提供STR鉴定图谱)
" "PubMed=833871; DOI=10.1093/jnci/58.2.209
Fogh J., Wright W.C., Loveless J.D.
Absence of HeLa cell contamination in 169 cell lines derived from human tumors.
J. Natl. Cancer Inst. 58:209-214(1977)
DOI=10.1007/BF00199208
Bruggen J., Sorg C., Macher E.
Membrane associated antigens of human malignant melanoma V: Serological typing of cell lines using antisera from nonhuman primates.
Cancer Immunol. Immunother. 5:53-62(1978)
PubMed=77569; DOI=10.1111/j.1399-0039.1978.tb01259.x
Espmark J.A., Ahlqvist-Roth L., Sarne L., Persson A.
Tissue typing of cells in culture. III. HLA antigens of established human cell lines. Attempts at typing by the mixed hemadsorption technique.
Tissue Antigens 11:279-286(1978)
PubMed=375235; DOI=10.1073/pnas.76.3.1288; PMCID=PMC383236
Sherwin S.A., Sliski A.H., Todaro G.J.
Human melanoma cells have both nerve growth factor and nerve growth factor-specific receptors on their cell surfaces.
Proc. Natl. Acad. Sci. U.S.A. 76:1288-1292(1979)
PubMed=22282976; DOI=10.1093/carcin/1.1.21
Day R.S. 3rd, Ziolkowski C.H.J., Scudiero D.A., Meyer S.A., Mattern M.R.
Human tumor cell strains defective in the repair of alkylation damage.
Carcinogenesis 1:21-32(1980)
DOI=10.1007/BF00205883
Bruggen J., Macher E., Sorg C.
Expression of surface antigens and its relation to parameters of malignancy in human malignant melanoma.
Cancer Immunol. Immunother. 10:121-127(1981)
PubMed=6954533; DOI=10.1073/pnas.79.7.2194; PMCID=PMC346157
Westin E.H., Gallo R.C., Arya S.K., Eva A., Souza L.M., Baluda M.A., Aaronson S.A., Wong-Staal F.
Differential expression of the amv gene in human hematopoietic cells.
Proc. Natl. Acad. Sci. U.S.A. 79:2194-2198(1982)
PubMed=6220172
Dracopoli N.C., Fogh J.
Polymorphic enzyme analysis of cultured human tumor cell lines.
J. Natl. Cancer Inst. 70:469-476(1983)
PubMed=6500159; DOI=10.1159/000163283
Gershwin M.E., Lentz D., Owens R.B.
Relationship between karyotype of tissue culture lines and tumorigenicity in nude mice.
Exp. Cell Biol. 52:361-370(1984)
PubMed=6584666; DOI=10.1093/jnci/72.4.913
Kozlowski J.M., Hart I.R., Fidler I.J., Hanna N.
A human melanoma line heterogeneous with respect to metastatic capacity in athymic nude mice.
J. Natl. Cancer Inst. 72:913-917(1984)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=1832891; DOI=10.1016/0277-5379(91)90277-K
Hansson J., Fichtinger-Schepman A.M.J., Edgren M.R., Ringborg U.
Comparative study of two human melanoma cell lines with different sensitivities to mustine and cisplatin.
Eur. J. Cancer 27:1039-1045(1991)
PubMed=9670966; DOI=10.4049/jimmunol.161.2.877
Bettinotti M.P., Kim C.J., Lee K.-H., Roden M., Cormier J.N., Panelli M.C., Parker K.K., Marincola F.M.
Stringent allele/epitope requirements for MART-1/Melan A immunodominance: implications for peptide-based immunotherapy.
J. Immunol. 161:877-889(1998)
PubMed=9973934; DOI=10.1016/S0165-4608(98)00122-8
Nelson M.A., Ariza M.E., Yang J.-M., Thompson F.H., Taetle R., Trent J.M., Wymer J., Massey-Brown K.S., Broome-Powell M., Easton J., Lahti J.M., Kidd V.J.
Abnormalities in the p34cdc2-related PITSLRE protein kinase gene complex (CDC2L) on chromosome band 1p36 in melanoma.
Cancer Genet. Cytogenet. 108:91-99(1999)
PubMed=10497214; DOI=10.1074/jbc.274.40.28505
Ariza M.E., Broome-Powell M., Lahti J.M., Kidd V.J., Nelson M.A.
Fas-induced apoptosis in human malignant melanoma cell lines is associated with the activation of the p34(cdc2)-related PITSLRE protein kinases.
J. Biol. Chem. 274:28505-28513(1999)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=14871852; DOI=10.1158/0008-5472.CAN-03-2209
Hogan K.T., Coppola M.A., Gatlin C.L., Thompson L.W., Shabanowitz J., Hunt D.F., Engelhard V.H., Ross M.M., Slingluff C.L. Jr.
Identification of novel and widely expressed cancer/testis gene isoforms that elicit spontaneous cytotoxic T-lymphocyte reactivity to melanoma.
Cancer Res. 64:1157-1163(2004)
PubMed=15009714; DOI=10.1046/j.0022-202X.2004.22243.x; PMCID=PMC2586668
Tsao H., Goel V., Wu H., Yang G., Haluska F.G.
Genetic interaction between NRAS and BRAF mutations and PTEN/MMAC1 inactivation in melanoma.
J. Invest. Dermatol. 122:337-341(2004)
PubMed=15467732; DOI=10.1038/sj.onc.1208152
Tanami H., Imoto I., Hirasawa A., Yuki Y., Sonoda I., Inoue J., Yasui K., Misawa-Furihata A., Kawakami Y., Inazawa J.
Involvement of overexpressed wild-type BRAF in the growth of malignant melanoma cell lines.
Oncogene 23:8796-8804(2004)
PubMed=17308088; DOI=10.1158/0008-5472.CAN-06-3311
Shields J.M., Thomas N.E., Cregger M., Berger A.J., Leslie M., Torrice C., Hao H.-L., Penland S., Arbiser J.L., Scott G.A., Zhou T., Bar-Eli M., Bear J.E., Der C.J., Kaufmann W.K., Rimm D.L., Sharpless N.E.
Lack of extracellular signal-regulated kinase mitogen-activated protein kinase signaling shows a new type of melanoma.
Cancer Res. 67:1502-1512(2007)
PubMed=18172304; DOI=10.1158/0008-5472.CAN-07-1939
Sabatino M., Zhao Y.-D., Voiculescu S., Monaco A., Robbins P.F., Karai L., Nickoloff B.J., Maio M., Selleri S., Marincola F.M., Wang E.
Conservation of genetic alterations in recurrent melanoma supports the melanoma stem cell hypothesis.
Cancer Res. 68:122-131(2008)
PubMed=19727395; DOI=10.1371/journal.pone.0006888; PMCID=PMC2731225
Wadlow R.C., Wittner B.S., Finley S.A., Bergquist H., Upadhyay R., Finn S.P., Loda M., Mahmood U., Ramaswamy S.
Systems-level modeling of cancer-fibroblast interaction.
PLoS ONE 4:E6888-E6888(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=21343389; DOI=10.1158/0008-5472.CAN-10-2958; PMCID=PMC3070783
Rose A.E., Poliseno L., Wang J.-H., Clark M., Pearlman A., Wang G.-M., Vega y Saenz de Miera E.C., Medicherla R., Christos P.J., Shapiro R., Pavlick A., Darvishian F., Zavadil J., Polsky D., Hernando E., Ostrer H., Osman I.
Integrative genomics identifies molecular alterations that challenge the linear model of melanoma progression.
Cancer Res. 71:2561-2571(2011)
PubMed=21424129; DOI=10.3892/or.2011.1220
Manca A., Sini M.C., Izzo F., Ascierto P.A., Tatangelo F., Botti G., Gentilcore G., Capone M., Mozzillo N., Rozzo C., Cossu A., Tanda F., Palmieri G.
Induction of arginosuccinate synthetase (ASS) expression affects the antiproliferative activity of arginine deiminase (ADI) in melanoma cells.
Oncol. Rep. 25:1495-1502(2011)
PubMed=21673604; DOI=10.1097/CMR.0b013e32834495c3; PMCID=PMC3131479
Orgaz J.L., Benguria A., Sanchez-Martinez C., Ladhani O., Volpert O.V., Jimenez B.
Changes in the gene expression profile of A375 human melanoma cells induced by overexpression of multifunctional pigment epithelium-derived factor.
Melanoma Res. 21:285-297(2011)
PubMed=21857157; DOI=10.4161/cc.10.17.17068; PMCID=PMC5479465
Caputo E., Maiorana L., Vasta V., Pezzino F.M., Sunkara S., Wynne K., Elia G., Marincola F.M., McCubrey J.A., Libra M., Travali S., Kane M.
Characterization of human melanoma cell lines and melanocytes by proteome analysis.
Cell Cycle 10:2924-2936(2011)
PubMed=22178978; DOI=10.1016/j.freeradbiomed.2011.11.019
Swalwell H., Latimer J., Haywood R.M., Birch-Machin M.A.
Investigating the role of melanin in UVA/UVB- and hydrogen peroxide-induced cellular and mitochondrial ROS production and mitochondrial DNA damage in human melanoma cells.
Free Radic. Biol. Med. 52:626-634(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23039341; DOI=10.1186/1476-4598-11-75; PMCID=PMC3554420
Byron S.A., Loch D.C., Wellens C.L., Wortmann A., Wu J.-Y., Wang J., Nomoto K., Pollock P.M.
Sensitivity to the MEK inhibitor E6201 in melanoma cells is associated with mutant BRAF and wildtype PTEN status.
Mol. Cancer 11:75.1-75.15(2012)
PubMed=24581590; DOI=10.1016/j.jdermsci.2014.01.006
Gehrke S., Otsuka A., Huber R., Meier B., Kistowska M., Fenini G., Cheng P., Dummer R., Kerl K., Contassot E., French L.E.
Metastatic melanoma cell lines do not secrete IL-1beta but promote IL-1beta production from macrophages.
J. Dermatol. Sci. 74:167-169(2014)
PubMed=25056119; DOI=10.1158/0008-5472.CAN-14-1232-T; PMCID=PMC4167745
Watson I.R., Li L.-R., Cabeceiras P.K., Mahdavi M., Gutschner T., Genovese G., Wang G.-C., Fang Z.-N., Tepper J.M., Stemke-Hale K., Tsai K.Y., Davies M.A., Mills G.B., Chin L.
The RAC1 P29S hotspot mutation in melanoma confers resistance to pharmacological inhibition of RAF.
Cancer Res. 74:4845-4852(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26405815; DOI=10.1371/journal.pone.0138210; PMCID=PMC4583389
Capaldo B.J., Roller D.G., Axelrod M.J., Koeppel A.F., Petricoin E.F. 3rd, Slingluff C.L. Jr., Weber M.J., Mackey A.J., Gioeli D., Bekiranov S.
Systems analysis of adaptive responses to MAP kinase pathway blockade in BRAF mutant melanoma.
PLoS ONE 10:E0138210-E0138210(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26673621; DOI=10.18632/oncotarget.6548; PMCID=PMC4823068
Roller D.G., Capaldo B.J., Bekiranov S., Mackey A.J., Conaway M.R., Petricoin E.F. 3rd, Gioeli D., Weber M.J.
Combinatorial drug screening and molecular profiling reveal diverse mechanisms of intrinsic and adaptive resistance to BRAF inhibition in V600E BRAF mutant melanomas.
Oncotarget 7:2734-2753(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29275043; DOI=10.1016/j.jprot.2017.12.013
Liberato T., Pessotti D.S., Fukushima I., Kitano E.S., Serrano S.M.T., Zelanis A.
Signatures of protein expression revealed by secretome analyses of cancer associated fibroblasts and melanoma cell lines.
J. Proteomics 174:1-8(2018)
PubMed=29492214; DOI=10.18632/oncotarget.23989; PMCID=PMC5823576
Sini M.C., Doneddu V., Paliogiannis P., Casula M., Colombino M., Manca A., Botti G., Ascierto P.A., Lissia A., Cossu A., Palmieri G.
Genetic alterations in main candidate genes during melanoma progression.
Oncotarget 9:8531-8541(2018)
PubMed=29605720; DOI=10.1016/j.neo.2018.02.009; PMCID=PMC5915992
Mologni L., Costanza M., Sharma G.G., Viltadi M., Massimino L., Citterio S., Purgante S., Raman H., Pirola A., Zucchetti M., Piazza R., Gambacorti-Passerini C.
Concomitant BCORL1 and BRAF mutations in vemurafenib-resistant melanoma cells.
Neoplasia 20:467-477(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"