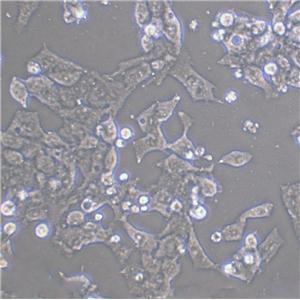
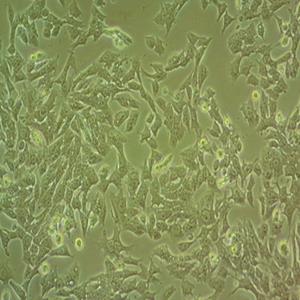
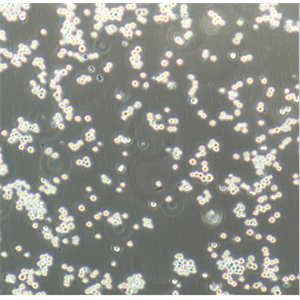
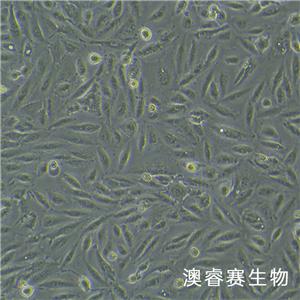
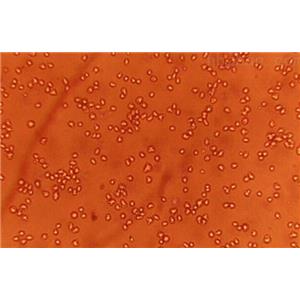
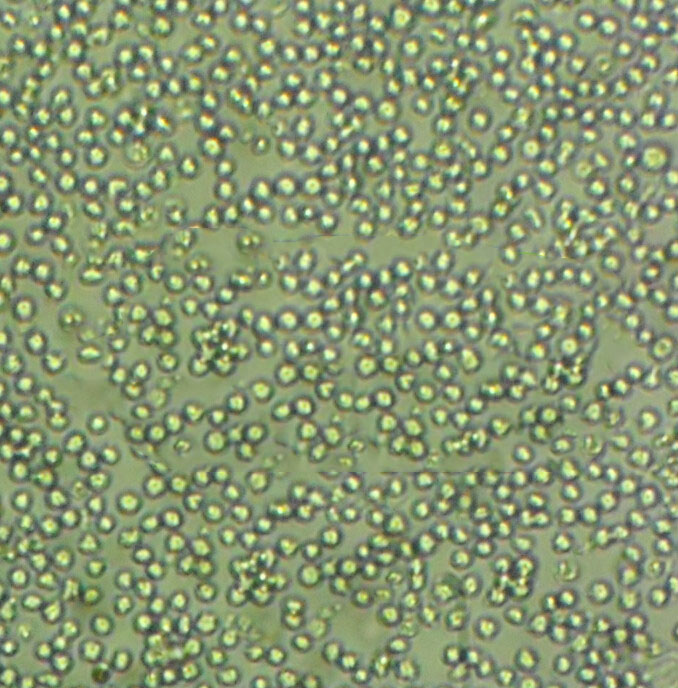
"A-498人肾癌细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
MC3T3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3-NS1/1-Ag4-1细胞、Panc 02细胞、COLO 201细胞
NCI H345 Cells;背景说明:小细胞肺癌;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TOV112D细胞、Earles's cells细胞、PATU-S细胞
VeroC1008 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Bronchial Epithelium transformed with Ad12-SV40 2B细胞、CEMO-1细胞、EST81细胞
背景信息:最初是从一名患有肾透明细胞癌(Clear Cell Renal Cell Carcinoma, ccRCC)的患者的肾脏肿瘤组织中分离并培养的。是一种人肾癌细胞系是由Aaronson·S建立,源自一位52岁患有肾癌的女性病人。
A-498人肾癌细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
SV40 MES 13 Cells;背景说明:肾小球系膜;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DITNC1细胞、OCI-Ly 10细胞、HCC-95细胞
TE-5 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Leydig细胞、KU1919细胞、T.T细胞
DBTRG-05MG Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:EBTr细胞、HB611细胞、NCI-SNU-216细胞
3T3-L1 CAR SNRK KO Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
A-498人肾癌细胞全年复苏|已有STR图谱
形态特性:上皮细胞样
正确的细胞复苏需知事项:细胞冻存HAO了,接下来要注意什么问题呢?没错,就是记得到时间了,拿出来复苏。那么,细胞复苏的过程中又有哪些该注意的事项呢?细胞活力和形态检查的作用何在?活力检查——千万不要使用不健康的细胞,可能有污染(真菌、支原体等),如果发现有污染毫不犹豫的丢弃!形态检查——检查细胞的固有形态和生长行为。冻存细胞:补充新的培养——在您开始冻存细胞的前一天补充新的培养。在细胞长至70%单层时收获细胞,计数活细胞数,用冻存调整细胞密度~5 x106 s/ml (根据不同的细胞类型调整);冻存——用冻存洗细胞并用冻存重悬细胞,有不同类型的冻存,根据细胞类型选择Zui合适的冻存(常用的冻存成分有):5-10% DMSO——注意确保DMSO不含有其他的毒性物质;5-15%甘油;如果细胞在无血清培养基内生长,应在50%条件培养基内(细胞在无血清培养基内生长24小时)内冻存和复苏。在冻存管上标记HAO细胞类型,日期,冻存人等信息,并保证每冻存管不超过1.5ml。放入罐之前记录冻存管的数量和位置。以Zui快的速度转移冻存管知罐内,因此,此步骤ZuiHAO使用干冰,或者把冻存管浸入装有的小盒内。此外还要注意,在冻存管上没有足够的空间记录细胞的详细信息,做HAO记录是非常非常重要的!还有一个Zui重要的,一定要在异地的罐内保存同样的一份细胞,以免其中的一个罐出现问题!细胞正确的复苏方式和正确的冻存方式同样重要,熟记以下要点:当从罐内取出细胞时,有可能会出现冻存管破裂的情况,使用保护面罩和防护服十分必要;其实,细胞复苏只是一个简单的实验,不过这其中却不可避免有一些需要注意的细节,不然,也不一定会尽如人意。例如说,人身健康方面:一定要记得做HAO防冻工作,戴上护目镜;尽量降低DMSO对细胞的损伤等等。
PANC-10-05 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SNU-878细胞、TGBC11TKB细胞、B16 BL6细胞
J774 A.1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SNU-1040细胞、F-81细胞、MCA205细胞
HPAF Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs 729细胞、SKMEL-24细胞、SNK1细胞
KLN 205 Cells;背景说明:肺;鳞癌细胞;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA-MB-175细胞、COLO357细胞、Hep3B细胞
LOUNH91 Cells;背景说明:肺鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CCD-112 CoN细胞、HSC-T6细胞、Vx-2细胞
LU451 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:J82 COT细胞、NCI-H1238细胞、MRC 5细胞
SK_HEP1 Cells;背景说明:SK-HEP-1细胞系已被鉴定为内皮来源。该细胞系为异倍体女性人(XX),染色体在亚三倍体范围内。在裸鼠中,它能形成与肝癌相一致的大细胞癌;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:WIL2 S细胞、Panc 03.27细胞、Hk-2细胞
H-838 Cells;背景说明:该细胞于1984年建系,源于一位59岁患有非小细胞肺癌的白人男性吸烟者,从患者淋巴结转移灶分离而来。;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CCLP-1细胞、ACC3细胞、IMR 90细胞
SK MEL 5 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:OVCA 420细胞、AKR细胞、Case 3细胞
PANC1005 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-2198细胞、N-87细胞、MOLM-16细胞
Hs675 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:H-1755细胞、Calf Pulmonary Artery 47细胞、Panc 1细胞
SUNE1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM06141B细胞、NCIH1930细胞、NCIH446细胞
UMNSAH/DF-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:NCM-460细胞、NCIH1666细胞、H-1993细胞
U 138 MG Cells;背景说明:星形细胞瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A375-MEL细胞、R-1059-D细胞、32D-Cl3细胞
NALM6 Cells;背景说明:急性B淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Ramos (RA)细胞、IPEC-J2细胞、CTV1细胞
HONE1 Cells;背景说明:鼻咽癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:T2 (174 x CEM.T2)细胞、TR-146细胞、Rin-M-5F细胞
HOS-MNNG Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GB1细胞、HUC细胞、H187细胞
Abcam A-549 HLA-DRA KO Cells(提供STR鉴定图谱)
Abcam THP-1 SUV39H1 KO Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSD390 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRT125 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC293 Cells(提供STR鉴定图谱)
CHL AR-16 Cells(提供STR鉴定图谱)
DA02209 Cells(提供STR鉴定图谱)
DD1493 Cells(提供STR鉴定图谱)
GM01928 Cells(提供STR鉴定图谱)
RGC-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:B16F0细胞、Human Epidermoid carcinoma #2细胞、A 72细胞
HITT15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CNE-1细胞、Lu-65细胞、HR-1细胞
NHA Cells;背景说明:星形胶质 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MPP-89细胞、CNE细胞、NIE 115细胞
COV-362 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:BAEC细胞、MKN45细胞、PLB 985细胞
HCC2157 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮生长;形态特性:上皮样;相关产品有:IMR-32细胞、Panc10.05细胞、OV433细胞
E.G7-OVA Cells;背景说明:详见相关文献介绍;传代方法:细胞密度保持在1×105—1×106 cells/ml,每周换液2—3次 ;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:EOC20细胞、CAKI 1细胞、AD-293细胞
TE7 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1417细胞、CPA 47细胞、UM-UC-14细胞
Clone Y-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C-Lu65细胞、H1666_DA细胞、CCLP1细胞
A-498人肾癌细胞全年复苏|已有STR图谱
HD-LM-2 Cells;背景说明:霍奇金淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:EOC 20细胞、GM04679细胞、FU-MMT-1细胞
K-1735 Cells;背景说明:黑色素瘤; C3H/HeN;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:786.O细胞、Calu 6细胞、HSAS3细胞
KMM-1 Cells;背景说明:骨髓瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MCF12F细胞、RPMI 2650细胞、CCRFCEM细胞
MRAEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LNCaP C4-2B细胞、LS-123细胞、RAMSCs细胞
Tu686 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:UCLA-RO 81细胞、SUM102细胞、RPMI8402细胞
A2780S Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:OE-33细胞、3T3 clone A31细胞、WSU-DLCL2细胞
H676B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CEM T4细胞、AN3细胞、ROS1728细胞
GM10044 Cells(提供STR鉴定图谱)
HAP1 BTRC (-) 2 Cells(提供STR鉴定图谱)
PG-LH7 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RSMC细胞、T/GHA-VSMC细胞、CAL 78细胞
University of Arizona Cell Culture-893 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:GM2132细胞、SKBr3细胞、Sp2/O细胞
NCIH1703 Cells;背景说明:该细胞1987年建系,源自一位54岁患有非小细胞肺癌的白人男性,该患者为吸烟者。;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NK-10A细胞、Hs 683细胞、A-172MG细胞
NEC-8 Cells;背景说明:睾丸胚胎性癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TE-10细胞、Panc2_03细胞、KTA7细胞
Anip973 Cells;背景说明:肺腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WI-38细胞、COLO-320-DM细胞、MDCKII细胞
BIC1 Cells;背景说明:食管腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1048细胞、TR 146细胞、AHH-1细胞
GOS3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH660细胞、H-28细胞、LAN-1细胞
ASH3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:University of Michigan-Renal Carcinoma-2细胞、MM1细胞、NCTC-929细胞
HG03476 Cells(提供STR鉴定图谱)
IDE8 Cells(提供STR鉴定图谱)
Lovo-175X2 Cells(提供STR鉴定图谱)
NCI-H711 Cells(提供STR鉴定图谱)
PathHunter U2OS IkappaB Degradation Cells(提供STR鉴定图谱)
Ubigene 4T1 Spp1 KO Cells(提供STR鉴定图谱)
UNESP-MM1 Cells(提供STR鉴定图谱)
HG01705 Cells(提供STR鉴定图谱)
CAL 12T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HOS-TE85细胞、SW-1990细胞、MES-SA/Dx-5细胞
NH6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:MonoMac6细胞、WERI细胞、HDQ-P1细胞
SUP-B1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RPMI 7951细胞、Hep-G2细胞、SKLMS1细胞
293FT Cells;背景说明:该细胞稳定表达SV40大T抗原,并且促进最适病毒产物的产生。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:圆形;相关产品有:Panc327细胞、BC3 H1细胞、C4-2 B细胞
KOSC2 Cells;背景说明:详见相关文献介绍;传代方法:2.2 x 10^4 cells/ml ;生长特性:贴壁生长;形态特性:上皮样;相关产品有:TALL-104细胞、SU-DHL2细胞、HGF-1细胞
KOSC2 Cells;背景说明:详见相关文献介绍;传代方法:2.2 x 10^4 cells/ml ;生长特性:贴壁生长;形态特性:上皮样;相关产品有:TALL-104细胞、SU-DHL2细胞、HGF-1细胞
W133 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HN 4细胞、UPCISCC090细胞、Sp2/0细胞
HPAF II Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-134-VI细胞、Caki-1细胞、NUGC2细胞
AML 12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Kit225 K6细胞、NCI-H1882细胞、NCIN87细胞
HCT-GEO Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SF 126细胞、H460细胞、NALM6细胞
KMST-6 Cells;背景说明:胚成纤维细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DF-1细胞、TK+/- (clone 3.7.2C)细胞、NCIH441细胞
KP-N-NS Cells;背景说明:源自脑转移灶的肾上腺神经母细胞瘤。;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CV 1细胞、NTERA-2cl.D1细胞、OVCAR420细胞
PK15 Cells;背景说明:PK-15细胞建系于1955(Stice,E)。是PK-1a细胞的克隆系。该细胞系可用于多种病毒的增值及特性研究。另外,电镜观察发现,PK-15细胞内有C-型病毒颗粒存在,是研究C-型病毒的材料。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:WM 2664细胞、HEC151细胞、Mel RM细胞
Nthy-ori 3.1 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CL 1-5细胞、LLC-PK(1)细胞、IPEC1细胞
H-378 Cells;背景说明:小细胞肺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW839细胞、NCI-H87细胞、Alpha Mouse Liver 12细胞
SBC-3/CDDP Cells(提供STR鉴定图谱)
HCC 94 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:624mel细胞、SK-MEL 28细胞、hTERT RPE-1细胞
Tu-212 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:SNU-475细胞、NCI-H526细胞、V79-4细胞
CMT 64 Cells;背景说明:肺腺癌;雌性;C57;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TGBC11T细胞、M20 [Human melanoma]细胞、NEC8细胞
Roswell Park Memorial Institute 1846 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PLC PRF 5细胞、HuT-102细胞、GM02219细胞
UCLA NPA-87-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SNU-16细胞、MHCC97-L细胞、HCC78细胞
B16/BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ketr 3细胞、Huh-7.5细胞、RWPE2细胞
Panc-327 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCEC-12细胞、SN12C细胞、SNU-C2B细胞
ChaGo K-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:UCH1细胞、HIT clone T15细胞、KYSE 510细胞
A-498人肾癌细胞全年复苏|已有STR图谱
RH-30 Cells;背景说明:肺泡横纹肌肉瘤;骨髓转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IAR-20细胞、SK-N-BE2C细胞、CMT 93细胞
ChaGo K-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:UCH1细胞、OE-33细胞、PL11细胞
526mel Cells;背景说明:黑色素瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MC3T3-E1(C4)细胞、T9细胞、P3-X63-Ag8细胞
SCC-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCIH1048细胞、A-10细胞、451-LU细胞
OAC-P4C Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Dysplastic Oral Keratinocyte细胞、Case 3细胞、293E细胞
L-929 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NW-MEL-38细胞、JB-6 Cl 30细胞、NCI-H719细胞
BayGenomics ES cell line CSG132 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRT510 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC508 Cells(提供STR鉴定图谱)
JS2 TLR4-/- WT Cells(提供STR鉴定图谱)
PCRP-ZBTB3-2E10 Cells(提供STR鉴定图谱)
mAb 35 Cells(提供STR鉴定图谱)
" "PubMed=571047
Fogh J.
Cultivation, characterization, and identification of human tumor cells with emphasis on kidney, testis, and bladder tumors.
Natl. Cancer Inst. Monogr. 49:5-9(1978)
PubMed=375235; DOI=10.1073/pnas.76.3.1288; PMCID=PMC383236
Sherwin S.A., Sliski A.H., Todaro G.J.
Human melanoma cells have both nerve growth factor and nerve growth factor-specific receptors on their cell surfaces.
Proc. Natl. Acad. Sci. U.S.A. 76:1288-1292(1979)
PubMed=450131; DOI=10.1038/279797a0
Day R.S. 3rd, Ziolkowski C.H.J.
Human brain tumour cell strains with deficient host-cell reactivation of N-methyl-N'-nitro-N-nitrosoguanidine-damaged adenovirus 5.
Nature 279:797-799(1979)
PubMed=6244232
Williams R.D.
Human urologic cancer cell lines.
Invest. Urol. 17:359-363(1980)
PubMed=22282976; DOI=10.1093/carcin/1.1.21
Day R.S. 3rd, Ziolkowski C.H.J., Scudiero D.A., Meyer S.A., Mattern M.R.
Human tumor cell strains defective in the repair of alkylation damage.
Carcinogenesis 1:21-32(1980)
PubMed=6935474; DOI=10.1093/jnci/66.2.239
Wright W.C., Daniels W.P., Fogh J.
Distinction of seventy-one cultured human tumor cell lines by polymorphic enzyme analysis.
J. Natl. Cancer Inst. 66:239-247(1981)
PubMed=7459858
Rousset M., Zweibaum A., Fogh J.
Presence of glycogen and growth-related variations in 58 cultured human tumor cell lines of various tissue origins.
Cancer Res. 41:1165-1170(1981)
PubMed=6954533; DOI=10.1073/pnas.79.7.2194; PMCID=PMC346157
Westin E.H., Gallo R.C., Arya S.K., Eva A., Souza L.M., Baluda M.A., Aaronson S.A., Wong-Staal F.
Differential expression of the amv gene in human hematopoietic cells.
Proc. Natl. Acad. Sci. U.S.A. 79:2194-2198(1982)
PubMed=6401685; DOI=10.1007/BF02617989
Halton D.M., Peterson W.D. Jr., Hukku B.
Cell culture quality control by rapid isoenzymatic characterization.
In Vitro 19:16-24(1983)
PubMed=6825208; DOI=10.1093/carcin/4.2.199
Yarosh D.B., Foote R.S., Mitra S., Day R.S. 3rd
Repair of O6-methylguanine in DNA by demethylation is lacking in Mer- human tumor cell strains.
Carcinogenesis 4:199-205(1983)
PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2041050; DOI=10.1093/jnci/83.11.757
Monks A., Scudiero D.A., Skehan P., Shoemaker R.H., Paull K.D., Vistica D.T., Hose C.D., Langley J., Cronise P., Vaigro-Wolff A., Gray-Goodrich M., Campbell H., Mayo J.G., Boyd M.R.
Feasibility of a high-flux anticancer drug screen using a diverse panel of cultured human tumor cell lines.
J. Natl. Cancer Inst. 83:757-766(1991)
PubMed=8493574; DOI=10.1126/science.8493574
Latif F., Tory K., Gnarra J.R., Yao M., Duh F.-M., Orcutt M.L., Stackhouse T., Kuzmin I.A., Modi W., Geil L., Schmidt L.S., Zhou F.-W., Li H., Wei M.H., Chen F., Glenn G., Choyke P., Walther M.M., Weng Y.-K., Duan D.-S.R., Dean M., Glavac D., Richards F.M., Crossey P.A., Ferguson-Smith M.A., Le Paslier D., Chumakov I., Cohen D., Chinault A.C., Maher E.R., Linehan W.M., Zbar B., Lerman M.I.
Identification of the von Hippel-Lindau disease tumor suppressor gene.
Science 260:1317-1320(1993)
PubMed=7915601; DOI=10.1038/ng0594-85
Gnarra J.R., Tory K., Weng Y.-K., Schmidt L.S., Wei M.H., Li H., Latif F., Liu S., Chen F., Duh F.-M., Lubensky I.A., Duan D.-S.R., Florence C., Pozzatti R., Walther M.M., Bander N.H., Grossman H.B., Brauch H., Pomer S., Brooks J.D., Isaacs W.B., Lerman M.I., Zbar B., Linehan W.M.
Mutations of the VHL tumour suppressor gene in renal carcinoma.
Nat. Genet. 7:85-90(1994)
PubMed=9488600; DOI=10.1007/s002800050744
Ma J.-G., Maliepaard M., Nooter K., Boersma A.W.M., Verweij J., Stoter G., Schellens J.H.M.
Synergistic cytotoxicity of cisplatin and topotecan or SN-38 in a panel of eight solid-tumor cell lines in vitro.
Cancer Chemother. Pharmacol. 41:307-316(1998)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=10723130; DOI=10.1038/sj.onc.1203449
Alimov A., Kost-Alimova M., Liu J., Li C.-D., Bergerheim U.S.R., Imreh S., Klein G., Zabarovsky E.R.
Combined LOH/CGH analysis proves the existence of interstitial 3p deletions in renal cell carcinoma.
Oncogene 19:1392-1399(2000)
PubMed=10929426; DOI=10.1007/s002400000103
Shintaku I., Kawagoe N., Yutani S., Hoshi S., Orikasa S., Yoshizumi O., Itoh K.
Expression of the SART1 tumor rejection antigen in renal cell carcinoma.
Urol. Res. 28:178-184(2000)
PubMed=11146448; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1034>3.0.CO;2-S
Kondo K.-i., Yao M., Kobayashi K., Ota S., Yoshida M., Kaneko S., Baba M., Sakai N., Kishida T., Kawakami S., Uemura H., Nagashima Y., Nakatani Y., Hosaka M.
PTEN/MMAC1/TEP1 mutations in human primary renal-cell carcinomas and renal carcinoma cell lines.
Int. J. Cancer 91:219-224(2001)
PubMed=15585611; DOI=10.1158/1078-0432.CCR-04-0072
Tykodi S.S., Warren E.H., Thompson J.A., Riddell S.R., Childs R.W., Otterud B.E., Leppert M.F., Storb R., Sandmaier B.M.
Allogeneic hematopoietic cell transplantation for metastatic renal cell carcinoma after nonmyeloablative conditioning: toxicity, clinical response, and immunological response to minor histocompatibility antigens.
Clin. Cancer Res. 10:7799-7811(2004)
PubMed=15604581; DOI=10.1159/000081597
Ikemoto S., Sugimura K., Yoshida N., Kuratsukuri K., Wada S., Nakatani T.
Comparative antitumor activity of 5-fluorouracil and 5'-deoxy-5-fluorouridine in combination with interferon-alpha in renal cell carcinoma cell lines.
Urol. Int. 73:348-353(2004)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=17409424; DOI=10.1158/0008-5472.CAN-06-4571
Furge K.A., Chen J.-D., Koeman J., Swiatek P.J., Dykema K., Lucin K., Kahnoski R., Yang X.-M.J., Teh B.T.
Detection of DNA copy number changes and oncogenic signaling abnormalities from gene expression data reveals MYC activation in high-grade papillary renal cell carcinoma.
Cancer Res. 67:3171-3176(2007)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=22949125; DOI=10.1002/ijc.27822
Pawlowski R., Muhl S.M., Sulser T., Krek W., Moch H., Schraml P.
Loss of PBRM1 expression is associated with renal cell carcinoma progression.
Int. J. Cancer 132:E11-E17(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26759240; DOI=10.1158/0008-5472.CAN-15-2201
Nakanishi Y., Walter K., Spoerke J.M., O'Brien C., Huw L.Y., Hampton G.M., Lackner M.R.
Activating mutations in PIK3CB confer resistance to PI3K inhibition and define a novel oncogenic role for p110beta.
Cancer Res. 76:1193-1203(2016)
PubMed=26972028; DOI=10.1016/j.jprot.2016.03.008
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Identification of glycosylphosphatidylinositol-anchored proteins and omega-sites using TiO2-based affinity purification followed by hydrogen fluoride treatment.
J. Proteomics 139:77-83(2016)
PubMed=27141528; DOI=10.1016/j.dib.2016.04.001; PMCID=PMC4838930
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Data for identification of GPI-anchored peptides and omega-sites in cancer cell lines.
Data Brief 7:1302-1305(2016)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=27993170; DOI=10.1186/s12943-016-0565-8; PMCID=PMC5168717
Brodaczewska K.K., Szczylik C., Fiedorowicz M., Porta C., Czarnecka A.M.
Choosing the right cell line for renal cell cancer research.
Mol. Cancer 15:83.1-83.15(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28489074; DOI=10.1038/ncomms15165; PMCID=PMC5436135
Sinha R., Winer A.G., Chevinsky M.S., Jakubowski C., Chen Y.-B., Dong Y.-Y., Tickoo S.K., Reuter V.E., Russo P., Coleman J.A., Sander C., Hsieh J.J.-D., Hakimi A.A.
Analysis of renal cancer cell lines from two major resources enables genomics-guided cell line selection.
Nat. Commun. 8:15165.1-15165.10(2017)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"