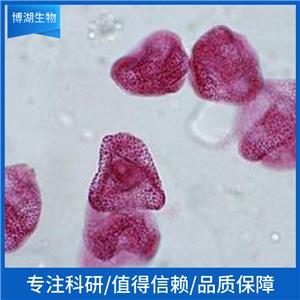
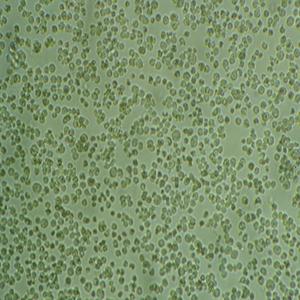
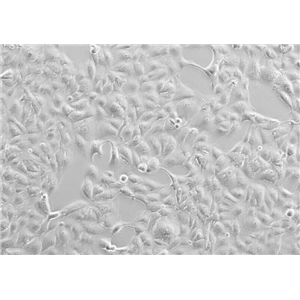
"MOLT-4人急性淋巴母细胞白血病细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:悬浮生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
HEK-293F Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;悬浮生长;形态特性:上皮细胞样;相关产品有:Jurkat E6细胞、FTC-133细胞、COLO 320DM细胞
32D:cl3 Cells;背景说明:骨髓淋巴瘤;C3H/HeJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Farage OL细胞、LS174细胞、HepG2/C3A细胞
LS411N Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-510细胞、GM2219C细胞、MC-CAR细胞
背景信息:MOLT-4与MOLT-3来源于一名19岁的男性急性淋巴细胞性白血病的复发患者,该患者前期接受过多种药物联合化疗。MOLT-4细胞系为T淋巴细胞起源,p53基因的第248位密码子有一个G→A突变,不表达p53,不表达免疫球蛋白或EB病毒;可产生高水平的末端脱氧核糖转移酶;表达CD1(49%),CD2(35%),CD3A(26%)B(33%)C(34%),CD4(55%),CD5(72%),CD6(22%),CD7(77%)。
MOLT-4人急性淋巴母细胞白血病细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
HN 4 Cells;背景说明:喉鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NTERA2细胞、EFM-192A细胞、NCI-H1915细胞
NB19 Cells;背景说明:神经母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IHC-ST1细胞、U-251 MG细胞、NCI-H1869细胞
HBVP Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH1793细胞、Panc-8_13细胞、Reuber H35细胞
11F4 Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
MOLT-4人急性淋巴母细胞白血病细胞全年复苏|已有STR图谱
形态特性:淋巴母细胞样
正确的细胞复苏需知事项:细胞冻存HAO了,接下来要注意什么问题呢?没错,就是记得到时间了,拿出来复苏。那么,细胞复苏的过程中又有哪些该注意的事项呢?细胞活力和形态检查的作用何在?活力检查——千万不要使用不健康的细胞,可能有污染(真菌、支原体等),如果发现有污染毫不犹豫的丢弃!形态检查——检查细胞的固有形态和生长行为。冻存细胞:补充新的培养——在您开始冻存细胞的前一天补充新的培养。在细胞长至70%单层时收获细胞,计数活细胞数,用冻存调整细胞密度~5 x106 s/ml (根据不同的细胞类型调整);冻存——用冻存洗细胞并用冻存重悬细胞,有不同类型的冻存,根据细胞类型选择Zui合适的冻存(常用的冻存成分有):5-10% DMSO——注意确保DMSO不含有其他的毒性物质;5-15%甘油;如果细胞在无血清培养基内生长,应在50%条件培养基内(细胞在无血清培养基内生长24小时)内冻存和复苏。在冻存管上标记HAO细胞类型,日期,冻存人等信息,并保证每冻存管不超过1.5ml。放入罐之前记录冻存管的数量和位置。以Zui快的速度转移冻存管知罐内,因此,此步骤ZuiHAO使用干冰,或者把冻存管浸入装有的小盒内。此外还要注意,在冻存管上没有足够的空间记录细胞的详细信息,做HAO记录是非常非常重要的!还有一个Zui重要的,一定要在异地的罐内保存同样的一份细胞,以免其中的一个罐出现问题!细胞正确的复苏方式和正确的冻存方式同样重要,熟记以下要点:当从罐内取出细胞时,有可能会出现冻存管破裂的情况,使用保护面罩和防护服十分必要;其实,细胞复苏只是一个简单的实验,不过这其中却不可避免有一些需要注意的细节,不然,也不一定会尽如人意。例如说,人身健康方面:一定要记得做HAO防冻工作,戴上护目镜;尽量降低DMSO对细胞的损伤等等。
L cell Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C38细胞、CHO-K1细胞、Hep-G2细胞
NCI-H498 Cells;背景说明:结肠腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:RPE1-hTERT细胞、B16 subline B78细胞、22Rv-1细胞
159PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H2122细胞、SuDHL 8细胞、CCD-1112sk细胞
MAntle cell VERona-1 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代;2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:CORL88细胞、PASMCS细胞、Tu686细胞
HAC-84 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Panc-3_27细胞、KNS62细胞、Hs-729-T细胞
Tn5 B1-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CORL51细胞、UCLA-RO 81细胞、B-3细胞
MDA-MB-134 Cells;背景说明:该细胞1973年由R. Cailleau建系,源自74岁乳腺导管癌女性患者的胸腔积液,细胞生长缓慢,松散贴壁,生长过程中会脱落到培养基,不会汇合,过表达FGF受体;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:OVCA432细胞、Tu212细胞、624-mel细胞
HO1-N1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SU-DHL-8细胞、Dakiki细胞、YH-13细胞
MH-S Cells;背景说明:巨噬细胞;雄性;SV40转化;BALB/cJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Stanford University-Diffuse Histiocytic Lymphoma-1细胞、PC12(高分化)细胞、KE39细胞
MDA MB 175 VII Cells;背景说明:该细胞源自一位54岁患有乳腺导管癌白人女性的胸腔积液。;传代方法:1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:Ontario Cancer Institute-Acute Myeloid Leukemia-5细胞、H-1623细胞、TO 175.T细胞
LTEPa2 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU-484细胞、PTK-1细胞、MADB106细胞
OPM-2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:NK-92MI细胞、OVCAR-420细胞、MSC细胞
SNU620 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H196细胞、NCI-H1838细胞、DU-145细胞
Transformed Human Liver Epithelial-2 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OKT3细胞、MOLM-13细胞、RAW264细胞
GM15452 Cells;背景说明:1957年,PuckTT从成年中国仓鼠卵巢的活检组织建立了CHO细胞,CHO-K1是CHO的一个亚克隆。CHO-K1的生长需要脯酸。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs 343.T细胞、Human Hepatocyte Line 5细胞、COLO 320细胞
Panc 8.13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BC-3H-1细胞、TOV-112D细胞、Medical Research Council cell strain-5细胞
NUGC3 Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:VMRC-RCZ细胞、SU-DHL6细胞、16-HBEo细胞
Abcam HEK293T BAMBI KO Cells(提供STR鉴定图谱)
AG09129 Cells(提供STR鉴定图谱)
BayGenomics ES cell line LRY069 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XC262 Cells(提供STR鉴定图谱)
Bristol 8 Cells(提供STR鉴定图谱)
COLO 55 Cells(提供STR鉴定图谱)
DA03918 Cells(提供STR鉴定图谱)
DA04775 Cells(提供STR鉴定图谱)
FS-22 Cells(提供STR鉴定图谱)
MDA-453 Cells;背景说明:该细胞系由CailleauR在1976年从一名48岁的患有转移性乳腺癌的白人女性的心包渗出液中分离建立的。该细胞表达FGF的受体。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:JCA1细胞、CHP-100细胞、Caco-2/BBe 1细胞
343MG Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Fox/NY细胞、ECC-12细胞、BCP-1细胞
HT-1197 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代,2天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:766T细胞、GA-10 (Clone 4)细胞、HCT-8/V细胞
WM239 Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM03320D细胞、SNU-423细胞、TU 686细胞
HEK-AD293 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:KASUMI1细胞、ROS1728细胞、LA-N-5细胞
SN-12C Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BRL 3A细胞、KM.12细胞、MCF7细胞
H1048 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:IGROV1细胞、HT55细胞、NL20细胞
H4-II-E Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hmy.2 CIR细胞、PANC 203细胞、624细胞
MOLT-4人急性淋巴母细胞白血病细胞全年复苏|已有STR图谱
NCI-1155 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:GM05372细胞、MCF7-GFP细胞、102PT细胞
P3/NSI/1-AG4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SW 1088细胞、Alexander细胞、CAL-148细胞
H-1694 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:HEL 92.1.7细胞、526-mel细胞、SKMEL-2细胞
SW626 Cells;背景说明:卵巢癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:XWLC-05细胞、VMRC-LCD细胞、HCV29细胞
NOR10 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-SNU-387细胞、SL-29细胞、UM-UC14细胞
Madin Darby Bovine Kidney Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:LU65细胞、JB6 [Mouse]细胞、H1623细胞
HT3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:FDC-P1细胞、HEK293EBNA细胞、OK细胞
GM21124 Cells(提供STR鉴定图谱)
HAP1 LRP1 (-) 3 Cells(提供STR鉴定图谱)
HTR8/SVneo Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:COS7细胞、P19细胞、Hela-Ap-1细胞
TE-32 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:P3-Jiyoye细胞、hTERT-RPE细胞、EVSAT细胞
Stanford University-Diffuse Histiocytic Lymphoma-6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:Tsup-1细胞、P3X63Ag8细胞、HTori-3.1细胞
Caco 2 Cells;背景说明:细胞株分离自一个原发性结肠癌。当细胞长满时,表现出典型的肠细胞分化的特征。Caco-2细胞表达维生素A酸结合蛋白I和视黄醇结合蛋白II,角蛋白阳性。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:HEM-L细胞、hSCC-25细胞、FRO细胞
PC9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CNE1细胞、HME-1细胞、SU-DHL-6细胞
NCIH647 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Kit 225-K6细胞、Tca-83细胞、CX1细胞
LS180 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW982细胞、MDA-MB453细胞、Hs 294T细胞
FLC7 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM00346B细胞、OVCAR8细胞、Nb2细胞
HPSI0316i-xaqm_4 Cells(提供STR鉴定图谱)
K562-AVB3 Cells(提供STR鉴定图谱)
Meg-J Cells(提供STR鉴定图谱)
NH50204 Cells(提供STR鉴定图谱)
QQ0527 Cells(提供STR鉴定图谱)
Ubigene Hep G2 GLUL KO Cells(提供STR鉴定图谱)
XTUU 139/30 Cells(提供STR鉴定图谱)
HG01921 Cells(提供STR鉴定图谱)
T/GHA-VSMC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SGC-7901细胞、NCIH441细胞、KOSC2细胞
ACC-2 Cells;背景说明:涎腺腺样囊性癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH226细胞、U373 MG细胞、Pt K1 (NBL-3)细胞
HN6 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HPB-ALL细胞、NOZ细胞、MOPC细胞
SK-RC-42 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LoMeT-ccRcc细胞、U87细胞、Stanford University-Diffuse Histiocytic Lymphoma-6细胞
MC/CAR Cells;背景说明:B淋巴;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A-2058细胞、A 72细胞、WEHI 3B细胞
MC/CAR Cells;背景说明:B淋巴;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A-2058细胞、A 72细胞、WEHI 3B细胞
SW982 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-436细胞、HEI-193细胞、HS 1.Tes细胞
Ontario Cancer Institute-Acute Myeloid Leukemia-5 Cells;背景说明:急性髓系白血病细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:H-676B细胞、Centre Antoine Lacassagne-148细胞、RIN-m 14B细胞
EoL-1-cell Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:BRL细胞、H-1048细胞、SPCA-1细胞
SCCVII/St Cells;背景说明:鳞状细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Panc-327细胞、LNCaP-ATCC细胞、LS 180细胞
MC116 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:Hs839.T细胞、SCaBER细胞、IHHA-1细胞
MB39 Cells;背景说明:脑瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Mc Ardle 7777细胞、JROECL 19细胞、NIH-3T3-L1细胞
Ontario Cancer Institute-Acute Myeloid Leukemia-4 Cells;背景说明:急性髓系白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs 294T细胞、U-118MG细胞、HNE-1细胞
BE(2)M17 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Mhh-Call 2细胞、MPP-89细胞、Normal Rat, August 3, 1983细胞
KPNRTBM1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:32D clone3细胞、H 9细胞、CP-70细胞
SQ-38 Cells(提供STR鉴定图谱)
GMK,BSC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE 85 ClF-5细胞、HEK-AD293细胞、HARA-B细胞
Porcine Kidney-15 Cells;背景说明:PK-15细胞建系于1955(Stice,E)。是PK-1a细胞的克隆系。该细胞系可用于多种病毒的增值及特性研究。另外,电镜观察发现,PK-15细胞内有C-型病毒颗粒存在,是研究C-型病毒的材料。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:PLA801C细胞、LS-1034细胞、8305C_1细胞
A204 Cells;背景说明:在裸鼠中成瘤。;传代方法:1:6-1:10传代;每周2-3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HuT 102细胞、KY-270细胞、TK10细胞
COLO-679 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:TE-6细胞、H283细胞、3T3-442A细胞
MGHU1 Cells;背景说明:膀胱癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WM2664细胞、MDA-MB-415细胞、RC-K8细胞
FM88 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BaF3细胞、NCTC-1469细胞、SKO007细胞
SCC 9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MLE-12细胞、SK-MEL-24细胞、GM03671C细胞
293H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SMC-1细胞、HEK (AD293)细胞、MSB-1细胞
MOLT-4人急性淋巴母细胞白血病细胞全年复苏|已有STR图谱
DHL6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:KASUMI6细胞、C22细胞、LM(TK-)细胞
EAhy926 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Hs.27细胞、HANK-1细胞、mREC细胞
NCI-H2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:NCI-H102细胞、OUMS27细胞、H522细胞
HBE 135-E6/E7 Cells;背景说明:支气管上皮细胞;HPV16转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FHL-124细胞、CEM T4细胞、NTERA-2 cl.D1细胞
Glioma 261 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:FL83B细胞、NWA细胞、UPCI-SCC-90细胞
L 132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MG63细胞、Caki-2细胞、NCIH548细胞
BayGenomics ES cell line GST107 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XB216 Cells(提供STR鉴定图谱)
CG7C7 Cells(提供STR鉴定图谱)
MANDM2-10A7 Cells(提供STR鉴定图谱)
Royan N43 Cells(提供STR鉴定图谱)
TRL 12-15 Cells(提供STR鉴定图谱)
" "PubMed=6181032; DOI=10.1016/0198-8859(82)90037-4
Rabinowitz R., Weinstock J., Margalioth E.J., Ben-Bassat H., Schlesinger M.
Antigens specific for human T-lymphocytes detected by xenoantisera to HD-MAR cells: their differential expression on various T-cell lines.
Hum. Immunol. 4:219-228(1982)
PubMed=6954533; DOI=10.1073/pnas.79.7.2194; PMCID=PMC346157
Westin E.H., Gallo R.C., Arya S.K., Eva A., Souza L.M., Baluda M.A., Aaronson S.A., Wong-Staal F.
Differential expression of the amv gene in human hematopoietic cells.
Proc. Natl. Acad. Sci. U.S.A. 79:2194-2198(1982)
PubMed=6198871; DOI=10.18926/AMO/32400
Oda T., Watanabe S., Nakamura T.
Type C virus particles produced in human T-cell lines derived from acute lymphoblastic leukemia and a leukemic T-lymphoid malignancy.
Acta Med. Okayama 37:529-533(1983)
PubMed=6200433; DOI=10.1007/BF00364762
Brodsky F.M.
A matrix approach to human class II histocompatibility antigens: reactions of four monoclonal antibodies with the products of nine haplotypes.
Immunogenetics 19:179-194(1984)
PubMed=6582512; DOI=10.1073/pnas.81.2.568; PMCID=PMC344720
Mattes M.J., Cordon-Cardo C., Lewis J.L. Jr., Old L.J., Lloyd K.O.
Cell surface antigens of human ovarian and endometrial carcinoma defined by mouse monoclonal antibodies.
Proc. Natl. Acad. Sci. U.S.A. 81:568-572(1984)
PubMed=2985879; DOI=10.1016/0145-2126(85)90084-0
Drexler H.G., Gaedicke G., Minowada J.
Isoenzyme studies in human leukemia-lymphoma cell lines -- 1 carboxylic esterase.
Leuk. Res. 9:209-229(1985)
PubMed=3159941; DOI=10.1016/0145-2126(85)90134-1
Drexler H.G., Gaedicke G., Minowada J.
Isoenzyme studies in human leukemia-lymphoma cell lines -- III Beta-hexosaminidase (E.C. 3.2.1.30).
Leuk. Res. 9:549-559(1985)
PubMed=3874327; DOI=10.1016/0145-2126(85)90133-x
Drexler H.G., Gaedicke G., Minowada J.
Isoenzyme studies in human leukemia-lymphoma cells lines -- II. Acid phosphatase.
Leuk. Res. 9:537-548(1985)
PubMed=2846092; DOI=10.1182/blood.V72.5.1755.1755
Greenberg J.M., Gonzalez-Sarmiento R., Arthur D.C., Wilkowski C.W., Streifel B.J., Kersey J.H.
Immunophenotypic and cytogenetic analysis of Molt-3 and Molt-4: human T-lymphoid cell lines with rearrangement of chromosome 7.
Blood 72:1755-1760(1988)
PubMed=3335022
Alley M.C., Scudiero D.A., Monks A., Hursey M.L., Czerwinski M.J., Fine D.L., Abbott B.J., Mayo J.G., Shoemaker R.H., Boyd M.R.
Feasibility of drug screening with panels of human tumor cell lines using a microculture tetrazolium assay.
Cancer Res. 48:589-601(1988)
PubMed=2140233; DOI=10.1111/j.1440-1827.1990.tb01549.x
Nakano A., Harada T., Morikawa S., Kato Y.
Expression of leukocyte common antigen (CD45) on various human leukemia/lymphoma cell lines.
Acta Pathol. Jpn. 40:107-115(1990)
PubMed=2144611; DOI=10.1128/mcb.10.10.5502-5509.1990; PMCID=PMC361264
Cheng J., Haas M.
Frequent mutations in the p53 tumor suppressor gene in human leukemia T-cell lines.
Mol. Cell. Biol. 10:5502-5509(1990)
PubMed=1765142; DOI=10.1016/0014-5793(91)81402-t
Orita S., Saiga A., Takagi S., Tanaka T., Okumura K., Aono Y., Hinuma Y., Igarashi H.
A novel alternatively spliced viral mRNA transcribed in cells infected with human T cell leukemia virus type 1 is mainly responsible for expressing p21X protein.
FEBS Lett. 295:127-134(1991)
PubMed=2041050; DOI=10.1093/jnci/83.11.757
Monks A., Scudiero D.A., Skehan P., Shoemaker R.H., Paull K.D., Vistica D.T., Hose C.D., Langley J., Cronise P., Vaigro-Wolff A., Gray-Goodrich M., Campbell H., Mayo J.G., Boyd M.R.
Feasibility of a high-flux anticancer drug screen using a diverse panel of cultured human tumor cell lines.
J. Natl. Cancer Inst. 83:757-766(1991)
CLPUB00447
Mulivor R.A., Suchy S.F.
1992/1993 catalog of cell lines. NIGMS human genetic mutant cell repository. 16th edition. October 1992.
(In misc. document) Institute for Medical Research (Camden, N.J.) NIH 92-2011; pp.1-918; National Institutes of Health; Bethesda; USA (1992)
PubMed=8316623; DOI=10.2307/3578190
Evans H.H., Ricanati M., Horng M.-F., Jiang Q.-Y., Mencl J., Olive P.L.
DNA double-strand break rejoining deficiency in TK6 and other human B-lymphoblast cell lines.
Radiat. Res. 134:307-315(1993)
PubMed=8127147
Heyman M., Grander D., Brondum-Nielsen K., Cederblad B., Liu Y., Xu B., Einhorn S.
Interferon system defects in malignant T-cells.
Leukemia 8:425-434(1994)
PubMed=8547074; DOI=10.1111/j.1365-2141.1995.tb05302.x
Siebert R., Willers C.P., Schramm A., Fossa A., Dresen I.M.G., Uppenkamp M.J., Nowrousian M.R., Seeber S., Opalka B.
Homozygous loss of the MTS1/p16 and MTS2/p15 genes in lymphoma and lymphoblastic leukaemia cell lines.
Br. J. Haematol. 91:350-354(1995)
DOI=10.11418/jtca1981.15.4_211
Matsuo Y., Okochi A., Ariyasu T., Iimura E., Ohno T.
Identification of cell lines with variable numbers of tandem repeat (VNTR) amplified by polymerase chain reaction.
Tissue Cult. Res. Commun. 15:211-219(1996)
PubMed=8558913
Morita S., Tsuchiya S., Fujie H., Itano M., Ohashi Y., Minegishi M., Imaizumi M., Endo M., Takano N., Konno T.
Cell surface c-kit receptors in human leukemia cell lines and pediatric leukemia: selective preservation of c-kit expression on megakaryoblastic cell lines during adaptation to in vitro culture.
Leukemia 10:102-105(1996)
PubMed=8641406; DOI=10.1111/j.1600-0609.1996.tb00721.x
Borgonovo-Brandter L., Heyman M., Rasool O., Liu Y., Grander D., Einhorn S.
p16INK4/p15INK4B gene inactivation is a frequent event in malignant T-cell lines.
Eur. J. Haematol. 56:313-318(1996)
PubMed=8957066; DOI=10.1111/j.1349-7006.1996.tb03112.x; PMCID=PMC5920998
Kawasaki N., Matsuo Y., Yoshino T., Yanai H., Oka T., Teramoto N., Liu C., Kondo E., Minowada J., Akagi T.
Metastatic potential of lymphoma/leukemia cell lines in SCID mice is closely related to expression of CD44.
Jpn. J. Cancer Res. 87:1070-1077(1996)
PubMed=9510473; DOI=10.1111/j.1349-7006.1998.tb00476.x; PMCID=PMC5921588
Hosoya N., Hangaishi A., Ogawa S., Miyagawa K., Mitani K., Yazaki Y., Hirai H.
Frameshift mutations of the hMSH6 gene in human leukemia cell lines.
Jpn. J. Cancer Res. 89:33-39(1998)
PubMed=9685479; DOI=10.1093/nar/26.16.3651; PMCID=PMC147775
Hultdin M., Gronlund E., Norrback K.-F., Eriksson-Lindstrom E., Just T., Roos G.
Telomere analysis by fluorescence in situ hybridization and flow cytometry.
Nucleic Acids Res. 26:3651-3656(1998)
PubMed=9787181; DOI=10.1182/blood.V92.9.3410
Sakai A., Thieblemont C., Wellmann A., Jaffe E.S., Raffeld M.
PTEN gene alterations in lymphoid neoplasms.
Blood 92:3410-3415(1998)
PubMed=10071127; DOI=10.1016/S0145-2126(98)00146-5
Kawamura M., Ohnishi H., Guo S.-X., Sheng X.-M., Minegishi M., Hanada R., Horibe K., Hongo T., Kaneko Y., Bessho F., Yanagisawa M., Sekiya T., Hayashi Y.
Alterations of the p53, p21, p16, p15 and RAS genes in childhood T-cell acute lymphoblastic leukemia.
Leuk. Res. 23:115-126(1999)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=10739008; DOI=10.1016/S0145-2126(99)00182-4
Inoue K., Kohno T., Takakura S., Hayashi Y., Mizoguchi H., Yokota J.
Frequent microsatellite instability and BAX mutations in T cell acute lymphoblastic leukemia cell lines.
Leuk. Res. 24:255-262(2000)
PubMed=11021758; DOI=10.1038/sj.leu.2401891
Majka M., Rozmyslowicz T., Honczarenko M.J., Ratajczak J., Wasik M.A., Gaulton G.N., Ratajczak M.Z.
Biological significance of the expression of HIV-related chemokine coreceptors (CCR5 and CXCR4) and their ligands by human hematopoietic cell lines.
Leukemia 14:1821-1832(2000)
DOI=10.1016/B978-0-12-221970-2.50457-5
Drexler H.G.
The leukemia-lymphoma cell line factsbook.
(In book) ISBN 9780122219702; pp.1-733; Academic Press; London; United Kingdom (2001)
PubMed=11226526; DOI=10.1016/S0145-2126(00)00121-1
Inoue K., Kohno T., Takakura S., Hayashi Y., Mizoguchi H., Yokota J.
Corrigendum to: Frequent microsatellite instability and BAX mutations in T cell acute lymphoblastic leukemia cell lines Leukemia Research 24 (2000), 255-262.
Leuk. Res. 25:275-278(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=11986953; DOI=10.1038/sj.leu.2402485
O'Donnell P.H., Guo W.-X., Reynolds C.P., Maurer B.J.
N-(4-hydroxyphenyl)retinamide increases ceramide and is cytotoxic to acute lymphoblastic leukemia cell lines, but not to non-malignant lymphocytes.
Leukemia 16:902-910(2002)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=15472075; DOI=10.1126/science.1102160
Weng A.P., Ferrando A.A., Lee W., Morris J.P. 4th, Silverman L.B., Sanchez-Irizarry C., Blacklow S.C., Look A.T., Aster J.C.
Activating mutations of NOTCH1 in human T cell acute lymphoblastic leukemia.
Science 306:269-271(2004)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=15901131; DOI=10.1016/j.prp.2005.01.002
Murai Y., Hayashi S., Takahashi H., Tsuneyama K., Takano Y.
Correlation between DNA alterations and p53 and p16 protein expression in cancer cell lines.
Pathol. Res. Pract. 201:109-115(2005)
PubMed=16408098; DOI=10.1038/sj.leu.2404081
Quentmeier H., MacLeod R.A.F., Zaborski M., Drexler H.G.
JAK2 V617F tyrosine kinase mutation in cell lines derived from myeloproliferative disorders.
Leukemia 20:471-476(2006)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=17117183; DOI=10.1038/sj.bjc.6603447; PMCID=PMC2360743
Beesley A.H., Palmer M.-L., Ford J., Weller R.E., Cummings A.J., Freitas J.R., Firth M.J., Perera K.U., de Klerk N.H., Kees U.R.
Authenticity and drug resistance in a panel of acute lymphoblastic leukaemia cell lines.
Br. J. Cancer 95:1537-1544(2006)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
DOI=10.4172/2157-7145.S2-005
Fang R.-X., Shewale J.G., Nguyen V.T., Cardoso H., Swerdel M.R., Hart R.P., Furtado M.R.
STR profiling of human cell lines: challenges and possible solutions to the growing problem.
J. Forensic Res. 2 Suppl. 2:5-5(2011)
PubMed=20922763; DOI=10.1002/pbc.22801; PMCID=PMC3005554
Kang M.H., Smith M.A., Morton C.L., Keshelava N., Houghton P.J., Reynolds C.P.
National Cancer Institute pediatric preclinical testing program: model description for in vitro cytotoxicity testing.
Pediatr. Blood Cancer 56:239-249(2011)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=24762992; DOI=10.7534/j.issn.1009-2137.2014.02.004
Ma X.-C., Liu C.-Y., Sun X.-J., He J.-J., Wan S.-G., Sun W.-L.
Genetic characteristics of human acute lymphoblastic leukemia cell line Molt-4.
Zhongguo Shi Yan Xue Ye Xue Za Zhi 22:280-284(2014)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26295583; DOI=10.1371/journal.pone.0135958; PMCID=PMC4546578
Vidyasekar P., Shyamsunder P., Arun R., Santhakumar R., Kapadia N.K., Kumar R., Verma R.S.
Genome wide expression profiling of cancer cell lines cultured in microgravity reveals significant dysregulation of cell cycle and microRNA gene networks.
PLoS ONE 10:E0135958-E0135958(2015)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28569787; DOI=10.1038/cddis.2017.249; PMCID=PMC5520907
Kasai S., Furuichi Y., Ando N., Kagami K., Abe M., Nakane T., Goi K., Inukai T., Saitoh S., Ohno S., Okazaki S., Nagano O., Saya H., Sugita K.
Inflammatory mediator ultra-low-molecular-weight hyaluronan triggers necrosis of B-precursor leukemia cells with high surface CD44 expression.
Cell Death Dis. 8:e2857.1-e2857.11(2017)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31160637; DOI=10.1038/s41598-019-44491-x; PMCID=PMC6547646
Quentmeier H., Pommerenke C., Dirks W.G., Eberth S., Koeppel M., MacLeod R.A.F., Nagel S., Steube K.G., Uphoff C.C., Drexler H.G.
The LL-100 panel: 100 cell lines for blood cancer studies.
Sci. Rep. 9:8218-8218(2019)"