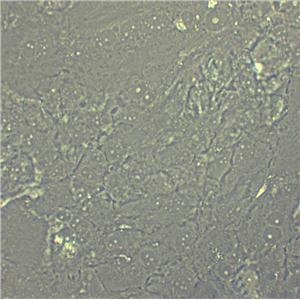
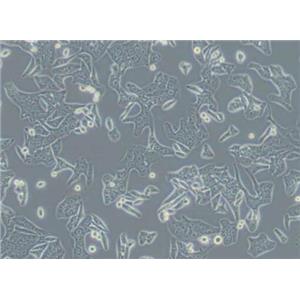
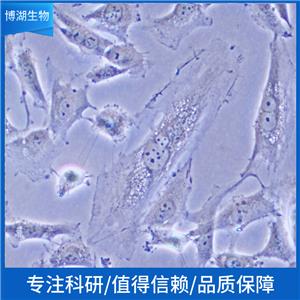
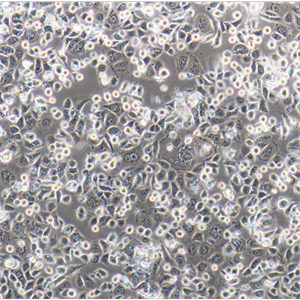
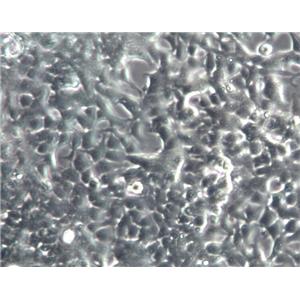
"LoVo人结肠癌细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
LC-2/ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE-85 clone 5细胞、SDBMSC细胞、AG 9细胞
NCI-SNU-601 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:U87 MG细胞、NIH3T3-L1细胞、H2591细胞
95C Cells;背景说明:肺巨细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BT474细胞、Lu-99A细胞、UO.31细胞
背景信息:LoVo建自1971年,从诊断为结肠腺癌的56岁男性白人的一个转移到左锁骨上区的肿瘤结节建系。癌基因C-myc,K-ras,H-ras,N-ras,Myb,sis和fos的表达呈阳性。癌基因N-myc的表达未做检测。它在裸鼠中能成瘤。与107细胞联合皮下接种5只裸鼠21天后全部成瘤。表达肿瘤特异的核基质蛋白蛋白CC-3和CC-4。
LoVo人结肠癌细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
ATCC细胞库(American Type Culture Colection),该中心一直致力于细胞分类、鉴定和保藏工作。ATCC是全球最大的生物资源保藏中心,ATCC通过行业标准产品、服务和创新解决方案支持全球学术、政府、生物技术、制药、食品、农业和工业领域的科学进步。ATCC提供的服务和定制解决方案包括细胞和微生物培养、鉴定、生物衍生物的开发和生产、性能测试和生物资源保藏服务。美国国家标准协会(ANSI)认可了ATCC标准开发组织,并制定了标准协议,以确保生物材料的可靠性和可重复性。ATCC的使命是为了获取、鉴定、保存、开发、标准化和分发生物资源和生物信息,以提高和应用生物科学知识。
PSN1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SVEC4-10细胞、TOV21G细胞、DiFi细胞
NCI-747 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Ramos-2G6-4C10细胞、CHG5细胞、FRO81-2细胞
C3H/10T1/2 clone 8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE 32细胞、PIG1细胞、RPMI-8402细胞
9D4.9.1 Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
LoVo人结肠癌细胞全年复苏|已有STR图谱
形态特性:上皮细胞样
细胞株(系)的使用,为医学研究和测试工作带来了大的方便。但细胞的传代是有限制的,长期连续传代的细胞,不仅消耗大量的人力和物力,而且细胞的生长与形态等会有一定退变或转化,因而细胞失去原有的遗传性,有时还会由于细胞污染而造成传代中断,种子丢失。因此,在实际工作中常需冻存一定数量的细胞,以备替换使用。细胞冷冻与复苏是细胞培养 室的常规工作和通用技术。目前,细胞冻存Zui常用的技术是冷冻保存法,主要采用加适量保护剂的缓慢冷冻法冻存细胞。细胞在不加任何保护剂的情况下直接冷冻,细胞内外的水分会很快形成冰晶,从而引起一系列不良反应。如细胞脱水使局部电解质浓度增GAO,pH值改变,部分蛋白质由于上述原因而变性,引起细胞内部空间结构紊乱,溶酶体膜由此遭到损伤而释放出溶酶体酶,使细胞内结构成分造成破坏,线粒体肿胀,功能丢失,并造成能量代谢障碍。胞膜上的类脂蛋白复合体也易破坏引起细胞膜通透性的改变,使细胞内容物丢失。如果细胞内冰晶形成较多,随冷冻温度的降低,冰晶体积膨胀造成细胞核DNA空间构型发生不可逆的损伤,而致细胞死亡。因此,细胞冷冻技术的关键是尽可能地减少细胞内水分,减少细胞内冰晶的形成。采用甘油或二甲基亚砜作保护剂,这两种物质分子量小,溶解度大,易穿透细胞,可以使冰点下降,提GAO细胞膜对水的通透性,且对细胞无明显毒性。慢速冷冻方法又可使细胞内的水分渗出细胞外,减少胞内形成冰结晶的机会,从而减少冰晶对细胞的损伤。
HT-55 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BT-474细胞、NS1/1-Ag4.1细胞、HLEB-3细胞
H-28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCIH2126细胞、NCI-H2228细胞、UACC-812细胞
BAR-T Cells;背景说明:食管;HGNC-TERT转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK-293F细胞、H22-H8D8细胞、CWR-22rv1细胞
ChaGo-K-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Michigan Cancer Foundation-7细胞、LS123细胞、CAKI1细胞
MAVER Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代;2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:HuH 6细胞、FL-83B细胞、P3.NS-1/1.Ag4.1细胞
H-1385 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:WIL2 Secreting细胞、H125细胞、Bat lung细胞
MARC 145 Cells;背景说明:胚肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CoCL2细胞、CP70细胞、T-T细胞
H-23 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:L 363细胞、SK-N-BE(2C)细胞、NCI-H187细胞
F56 [Human neoplasm] Cells;背景说明:F56是一株人的腺癌细胞系,可用于腺癌发生机制的研究,也可用于抗癌药物的筛选。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:CL-11细胞、PANC-02-03细胞、TE-85 clone 5细胞
Hep 3B Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Factor Dependent Continuous-Paterson 1细胞、SNGM细胞、HEK 293 c18细胞
NG10815 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P30-OHK细胞、B78细胞、IOSE-80细胞
Panc-3_27 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:WiDr细胞、SUIT 2细胞、PC3M-2B4细胞
ChaGo K-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:UCH1细胞、HIT clone T15细胞、KYSE 510细胞
NCI-H2073 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Gerner 7666细胞、HS-68细胞、B35细胞
Pt-K2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDA 231-LM2-4175细胞、HFT-8810细胞、HKF细胞
GT38 Cells;背景说明:胃癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TOG细胞、CoCL3细胞、Mv 1 Lu细胞
NB19-RIKEN Cells;背景说明:神经母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MODE-K细胞、NCI-H2342细胞、IPEC-J2细胞
Abcam HCT 116 KRT19 KO Cells(提供STR鉴定图谱)
AG07613 Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSJ151 Cells(提供STR鉴定图谱)
BayGenomics ES cell line TEA009 Cells(提供STR鉴定图谱)
BJTTHi003-A Cells(提供STR鉴定图谱)
CL1DNEO Cells(提供STR鉴定图谱)
DA03396 Cells(提供STR鉴定图谱)
eCG 50.5 Cells(提供STR鉴定图谱)
GM05209 Cells(提供STR鉴定图谱)
H-920 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:JVM3细胞、SNU475细胞、NCIH2052细胞
NCI747 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:N87细胞、KYSE0520细胞、NCI-H735细胞
V-79-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HBZY 1细胞、C3H/10T1/2 clone 8细胞、A204细胞
High-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH446细胞、CL-40细胞、HCC-1359细胞
OCI-LY-1 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs 445细胞、L6细胞、SMMC7721细胞
H3255 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:RCC-4细胞、KP4细胞、KU-812F细胞
NCI-H2106 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:Institute for Medical Research-32细胞、SK-OV-433细胞、VMM-5A细胞
VeroC1008 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Bronchial Epithelium transformed with Ad12-SV40 2B细胞、CEMO-1细胞、EST81细胞
LoVo人结肠癌细胞全年复苏|已有STR图谱
Rat 2 Cells;背景说明:成纤维细胞;自发永生;Fischer 344;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:AR4IP细胞、TEV-1细胞、253J B-V细胞
AC29 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEC-1-B细胞、H8细胞、GM3190细胞
K-1735 Cells;背景说明:黑色素瘤; C3H/HeN;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:786.O细胞、Calu 6细胞、HSAS3细胞
BAEC Cells;背景说明:主动脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C28/I2细胞、H295细胞、LK2细胞
BEAS-2B Cells;背景说明:从一位非癌个体的正常人支气管上皮病理切片分离出上皮细胞。这些细胞用腺病毒12-SV40病毒杂交病毒感染并克隆。DEAS-2B细胞保留了对血清反应进行鳞关分化的能力,并有用于筛选诱导或影响分化及致癌的化学或生物制剂。细胞角蛋白及SV40抗原染色阳性。;传代方法:消化3-5分钟。1:2。3天内可长满;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:PA-1细胞、143TK-细胞、HCC0078细胞
BEAS-2B Cells;背景说明:从一位非癌个体的正常人支气管上皮病理切片分离出上皮细胞。这些细胞用腺病毒12-SV40病毒杂交病毒感染并克隆。DEAS-2B细胞保留了对血清反应进行鳞关分化的能力,并有用于筛选诱导或影响分化及致癌的化学或生物制剂。细胞角蛋白及SV40抗原染色阳性。;传代方法:消化3-5分钟。1:2。3天内可长满;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:PA-1细胞、143TK-细胞、HCC0078细胞
HDQ-P1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCT.116细胞、A-673细胞、HIEC细胞
HAP1 ABCC1 (-) 3 Cells(提供STR鉴定图谱)
HAP1 SCML2 (-) 2 Cells(提供STR鉴定图谱)
CTV1 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Intestinal Epithelioid Cell line No. 6细胞、NEC细胞、PANC.1细胞
MHCC97H Cells;背景说明:来源于中山医院,用人肝癌细胞株MHCC97-H接种裸鼠,进行3次肺转移筛选,取肺转移瘤建成皮下接种后高度自发性肺转移的肝癌细胞系;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Melanoma 14细胞、M-O7e细胞、H2030细胞
HL-60 Cells;背景说明:该细胞由CollinsSJ从一位患有急性早幼粒细胞性白血病的36岁白人女性的外周血中分离建立;可自发分化,或在盐、次黄嘌呤、佛波醇肉豆蔻酸(PMA,TPA)、DMSO(1%to1.5%)、D和视黄酸的刺激下发生分化;PMA刺激后可分泌TNF-α。该细胞具有吞噬活性和趋化反应,癌基因myc阳性,表达补体受体和FcR。;传代方法:维持细胞浓度在1×105-1×106/ml,每2-3天换液1次。;生长特性:悬浮生长;形态特性:髓母细胞样;相关产品有:Panc-813细胞、CX-1细胞、KNS62细胞
3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Me-Wo细胞、WERIRb1细胞、Line 207细胞
NB9 Cells;背景说明:详见相关文献介绍;传代方法:1:10 1:50每2 - 3周;每周换液2-3次。;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:WM451细胞、A-9细胞、MPC11细胞
NPA'87 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Lewis-Lung细胞、HBZY1细胞、87 MG细胞
TFK1 Cells;背景说明:胆管癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ID8细胞、H520细胞、HCC1143细胞
MFM-223 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKLU-1细胞、KM12-SM细胞、P3X63Ag8-6-5-3细胞
HyCyte H4-II KO-rInsrr Cells(提供STR鉴定图谱)
LC-06-JCK Cells(提供STR鉴定图谱)
MZ-MEL-18 Cells(提供STR鉴定图谱)
OUN-1 Cells(提供STR鉴定图谱)
RPMI-8432 Cells(提供STR鉴定图谱)
Ubigene HEK293 PPA2 KO Cells(提供STR鉴定图谱)
WG3733 Cells(提供STR鉴定图谱)
HARA [Human B-cell IHW] Cells(提供STR鉴定图谱)
GLC82 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCTC 929细胞、Panc5.04细胞、TE-8细胞
Rat Fetal Lung-6 Cells;背景说明:胚肺;成纤维细胞;SD大鼠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JB6 [Mouse]细胞、HUV-EC-C细胞、SK-MEL-2细胞
MM1S Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:混合生长;形态特性:淋巴母细胞样;相关产品有:L-1210细胞、FAT细胞、B5537SKIN细胞
293H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SMC-1细胞、HEK (AD293)细胞、MSB-1细胞
CEM-CCRF (CAMR) Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:H-358细胞、SKM-1细胞、Epstein-Barr-3细胞
CEM-CCRF (CAMR) Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:H-358细胞、SKM-1细胞、Epstein-Barr-3细胞
JKT-1 Cells;背景说明:精原瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEYA8细胞、NCI-SNU-449细胞、HCC0366细胞
RGCs Cells;背景说明:视网膜;神经节 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-MES-1细胞、C81-61细胞、3T6 Swiss Albino细胞
Eph4 1424 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HDMEC细胞、RBMVEC细胞、LC-2-Ad细胞
RBSMC Cells;背景说明:脑动脉;平滑肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LNCaP-C4-2细胞、MDA MB231细胞、HFT 8810细胞
CFSC-8B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L-6 myoblast细胞、HCAEC细胞、M20 [Human melanoma]细胞
CHL-1 Cells;背景说明:详见相关文献介绍;传代方法:1:6—1:10传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCI-128细胞、HEK/EBNA细胞、MGH-U1细胞
RMS13 Cells;背景说明:肺泡横纹肌肉瘤;骨髓转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WM115-mel细胞、P3 NS1 Ag4细胞、SP2细胞
CHO Cells;背景说明:1957年,PuckTT从成年中国仓鼠卵巢的活检组织建立了CHO细胞;广泛用于生物制品的表达。;传代方法:1:3传代,3-4天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MT-2细胞、EVSAT细胞、HECCL-1细胞
Swiss 3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:JROECL 19细胞、624细胞、HNE2细胞
TCC-SUPrGEMCI20 Cells(提供STR鉴定图谱)
OCI-Ly 18 Cells;背景说明:弥漫大B细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:8305C细胞、F81细胞、P-3J细胞
GM637A Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HO-8910 PM细胞、686LN-M4e细胞、Hs 746.T细胞
MCF 7B Cells;背景说明:浸润性导管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:J-111细胞、CCD 1112SK细胞、Hep 2细胞
T_T_ Cells;背景说明:详见相关文献介绍;传代方法:10^5 cells/60mm dish;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BN-CL2细胞、NCIH82细胞、LP1细胞
WIL2S Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TGW细胞、HOC细胞、SF-539细胞
U373-MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Human Melanoma Cell Bowes细胞、HCC1954细胞、Me-Wo细胞
RKO_AS45 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCL-H548细胞、X63-Ag8.653细胞、Saos2细胞
CHL1 Cells;背景说明:详见相关文献介绍;传代方法:1:6—1:10传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MBMEC细胞、NCI-H220细胞、MDA157细胞
LoVo人结肠癌细胞全年复苏|已有STR图谱
HFLS Cells;背景说明:滑膜;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MPC11细胞、HCC-9204细胞、HEM细胞
SK-RC-39 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PE/CA-PJ34细胞、Huh 7.5.1细胞、GM03571细胞
SF17 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC2157细胞、IPLB-SF-21-AE细胞、Mouse Forestomach Carcinoma细胞
GC-2spd(ts) Cells;背景说明:精母细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Lilly Laboratories Cell-Monkey Kidney 2细胞、JM1细胞、BNL-HCC细胞
SK-NSH Cells;背景说明:SK-N-SH细胞系由J.L.Bieder建系,它与SK-N-MC所不同的是倍增时间较长且多巴胺-β-羟基酶水平较高。 SK-N-SH在细胞介导的细胞毒性试验中用作靶细胞系。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:上皮细胞样;相关产品有:BHK 21细胞、EFM-192C细胞、NCI-SNU-475细胞
SUM 102 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK BR 03细胞、H157细胞、SK Mel 24细胞
BayGenomics ES cell line CSH170 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRU529 Cells(提供STR鉴定图谱)
BCL1 clone CW13.20-3B3 Cells(提供STR鉴定图谱)
KKF Cells(提供STR鉴定图谱)
PCRP-ZNF485-1G1 Cells(提供STR鉴定图谱)
NOBEC-1 Cells(提供STR鉴定图谱)
" "PubMed=8464898; DOI=10.1073/pnas.90.7.2842; PMCID=PMC46192
Browning M.J., Krausa P., Rowan A.J., Bicknell D.C., Bodmer J.G., Bodmer W.F.
Tissue typing the HLA-A locus from genomic DNA by sequence-specific PCR: comparison of HLA genotype and surface expression on colorectal tumor cell lines.
Proc. Natl. Acad. Sci. U.S.A. 90:2842-2845(1993)
PubMed=8197130; DOI=10.1073/pnas.91.11.4751; PMCID=PMC43866
Bicknell D.C., Rowan A.J., Bodmer W.F.
Beta 2-microglobulin gene mutations: a study of established colorectal cell lines and fresh tumors.
Proc. Natl. Acad. Sci. U.S.A. 91:4751-4755(1994)
PubMed=7651727
Kastrinakis W.V., Ramchurren N., Rieger K.M., Hess D.T., Loda M., Steele G., Summerhayes I.C.
Increased incidence of p53 mutations is associated with hepatic metastasis in colorectal neoplastic progression.
Oncogene 11:647-652(1995)
PubMed=9000147
Cottu P.-H., Muzeau F., Estreicher A., Flejou J.-F., Iggo R.D., Thomas G., Hamelin R.
Inverse correlation between RER+ status and p53 mutation in colorectal cancer cell lines.
Oncogene 13:2727-2730(1996)
PubMed=9000572
Hoang J.-M., Cottu P.-H., Thuille B., Salmon R.J., Thomas G., Hamelin R.
BAT-26, an indicator of the replication error phenotype in colorectal cancers and cell lines.
Cancer Res. 57:300-303(1997)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::aid-mc5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=9294210; DOI=10.1073/pnas.94.19.10330; PMCID=PMC23362
Ilyas M., Tomlinson I.P.M., Rowan A.J., Pignatelli M., Bodmer W.F.
Beta-catenin mutations in cell lines established from human colorectal cancers.
Proc. Natl. Acad. Sci. U.S.A. 94:10330-10334(1997)
PubMed=9515795
Sparks A.B., Morin P.J., Vogelstein B., Kinzler K.W.
Mutational analysis of the APC/beta-catenin/Tcf pathway in colorectal cancer.
Cancer Res. 58:1130-1134(1998)
PubMed=9715273; DOI=10.1038/sj.onc.1201986
Eshleman J.R., Casey G., Kochera M.E., Sedwick W.D., Swinler S.E., Veigl M.L., Willson J.K.V., Schwartz S., Markowitz S.D.
Chromosome number and structure both are markedly stable in RER colorectal cancers and are not destabilized by mutation of p53.
Oncogene 17:719-725(1998)
PubMed=10674020; DOI=10.1016/S0959-8049(99)00206-3
Ku J.-L., Yoon K.-A., Kim D.-Y., Park J.-G.
Mutations in hMSH6 alone are not sufficient to cause the microsatellite instability in colorectal cancer cell lines.
Eur. J. Cancer 35:1724-1729(1999)
PubMed=10612807; DOI=10.1002/(SICI)1098-2264(200002)27:2<183::aid-gcc10>3.0.CO;2-P; PMCID=PMC4721570
Ghadimi B.M., Sackett D.L., Difilippantonio M.J., Schrock E., Neumann T., Jauho A., Auer G., Ried T.
Centrosome amplification and instability occurs exclusively in aneuploid, but not in diploid colorectal cancer cell lines, and correlates with numerical chromosomal aberrations.
Genes Chromosomes Cancer 27:183-190(2000)
PubMed=10737795; DOI=10.1073/pnas.97.7.3352; PMCID=PMC16243
Rowan A.J., Lamlum H., Ilyas M., Wheeler J.M.D., Straub J., Papadopoulou A., Bicknell D.C., Bodmer W.F., Tomlinson I.P.M.
APC mutations in sporadic colorectal tumors: a mutational 'hotspot' and interdependence of the 'two hits'.
Proc. Natl. Acad. Sci. U.S.A. 97:3352-3357(2000)
PubMed=11226274; DOI=10.1073/pnas.041603298; PMCID=PMC30173
Abdel-Rahman W.M., Katsura K., Rens W., Gorman P.A., Sheer D., Bicknell D.C., Bodmer W.F., Arends M.J., Wyllie A.H., Edwards P.A.W.
Spectral karyotyping suggests additional subsets of colorectal cancers characterized by pattern of chromosome rearrangement.
Proc. Natl. Acad. Sci. U.S.A. 98:2538-2543(2001)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=11526487; DOI=10.1038/sj.onc.1204611
Gayet J., Zhou X.-P., Duval A., Rolland S., Hoang J.-M., Cottu P.-H., Hamelin R.
Extensive characterization of genetic alterations in a series of human colorectal cancer cell lines.
Oncogene 20:5025-5032(2001)
PubMed=11668190; DOI=10.1177/002215540104901105
Quentmeier H., Osborn M., Reinhardt J., Zaborski M., Drexler H.G.
Immunocytochemical analysis of cell lines derived from solid tumors.
J. Histochem. Cytochem. 49:1369-1378(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=12584437; DOI=10.1159/000068544
Melcher R., Koehler S., Steinlein C., Schmid M., Mueller C.R., Luehrs H., Menzel T., Scheppach W., Moerk H., Scheurlen M., Koehrle J., Al-Taie O.
Spectral karyotype analysis of colon cancer cell lines of the tumor suppressor and mutator pathway.
Cytogenet. Genome Res. 98:22-28(2002)
PubMed=12615714
Hempen P.M., Zhang L., Bansal R.K., Iacobuzio-Donahue C.A., Murphy K.M., Maitra A., Vogelstein B., Whitehead R.H., Markowitz S.D., Willson J.K.V., Yeo C.J., Hruban R.H., Kern S.E.
Evidence of selection for clones having genetic inactivation of the activin A type II receptor (ACVR2) gene in gastrointestinal cancers.
Cancer Res. 63:994-999(2003)
PubMed=12661003; DOI=10.1002/gcc.10196
Seitz S., Wassmuth P., Plaschke J., Schackert H.K., Karsten U., Santibanez-Koref M.F., Schlag P.M., Scherneck S.
Identification of microsatellite instability and mismatch repair gene mutations in breast cancer cell lines.
Genes Chromosomes Cancer 37:29-35(2003)
PubMed=15771911; DOI=10.1016/j.cancergencyto.2004.08.023
Melcher R., Maisch S., Koehler S., Bauer M., Steinlein C., Schmid M., Kudlich T., Schauber J., Luehrs H., Menzel T., Scheppach W.
SKY and genetic fingerprinting reveal a cross-contamination of the putative normal colon epithelial cell line NCOL-1.
Cancer Genet. Cytogenet. 158:84-87(2005)
PubMed=15900046; DOI=10.1093/jnci/dji133
Mashima T., Oh-hara T., Sato S., Mochizuki M., Sugimoto Y., Yamazaki K., Hamada J.-i., Tada M., Moriuchi T., Ishikawa Y., Kato Y., Tomoda H., Yamori T., Tsuruo T.
p53-defective tumors with a functional apoptosome-mediated pathway: a new therapeutic target.
J. Natl. Cancer Inst. 97:765-777(2005)
PubMed=16418264; DOI=10.1073/pnas.0510146103; PMCID=PMC1327731
Liu Y., Bodmer W.F.
Analysis of p53 mutations and their expression in 56 colorectal cancer cell lines.
Proc. Natl. Acad. Sci. U.S.A. 103:976-981(2006)
PubMed=16854228; DOI=10.1186/1476-4598-5-29; PMCID=PMC1550420
Bandres Elizalde E.M., Cubedo E., Agirre X., Malumbres R., Zarate R., Ramirez N., Abajo A., Navarro A., Moreno I., Monzo M., Garcia-Foncillas J.
Identification by real-time PCR of 13 mature microRNAs differentially expressed in colorectal cancer and non-tumoral tissues.
Mol. Cancer 5:29.1-29.10(2006)
PubMed=18258742; DOI=10.1073/pnas.0712176105; PMCID=PMC2268141
Emaduddin M., Bicknell D.C., Bodmer W.F., Feller S.M.
Cell growth, global phosphotyrosine elevation, and c-Met phosphorylation through Src family kinases in colorectal cancer cells.
Proc. Natl. Acad. Sci. U.S.A. 105:2358-2362(2008)
PubMed=19927377; DOI=10.1002/gcc.20730; PMCID=PMC2818350
Knutsen T., Padilla-Nash H.M., Wangsa D., Barenboim-Stapleton L., Camps J., McNeil N.E., Difilippantonio M.J., Ried T.
Definitive molecular cytogenetic characterization of 15 colorectal cancer cell lines.
Genes Chromosomes Cancer 49:204-223(2010)
PubMed=19941903; DOI=10.1016/j.jviromet.2009.11.022
Karger A., Bettin B., Lenk M., Mettenleiter T.C.
Rapid characterisation of cell cultures by matrix-assisted laser desorption/ionisation mass spectrometric typing.
J. Virol. Methods 164:116-121(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20570890; DOI=10.1158/0008-5472.CAN-10-0192; PMCID=PMC2943514
Janakiraman M., Vakiani E., Zeng Z.-S., Pratilas C.A., Taylor B.S., Chitale D., Halilovic E., Wilson M., Huberman K., Ricarte Filho J.C.M., Persaud Y., Levine D.A., Fagin J.A., Jhanwar S.C., Mariadason J.M., Lash A., Ladanyi M., Saltz L.B., Heguy A., Paty P.B., Solit D.B.
Genomic and biological characterization of exon 4 KRAS mutations in human cancer.
Cancer Res. 70:5901-5911(2010)
PubMed=20606684; DOI=10.1038/sj.bjc.6605780; PMCID=PMC2920028
Bracht K., Nicholls A.M., Liu Y., Bodmer W.F.
5-fluorouracil response in a large panel of colorectal cancer cell lines is associated with mismatch repair deficiency.
Br. J. Cancer 103:340-346(2010)
PubMed=20831567; DOI=10.1111/j.1582-4934.2010.01170.x; PMCID=PMC3918049
Ma Y.-L., Zhang P., Wang F., Moyer M.P., Yang J.-J., Liu Z.-H., Peng J.-Y., Chen H.-Q., Zhou Y.-K., Liu W.-J., Qin H.-L.
Human embryonic stem cells and metastatic colorectal cancer cells shared the common endogenous human microRNA-26b.
J. Cell. Mol. Med. 15:1941-1954(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=24042735; DOI=10.1038/oncsis.2013.35; PMCID=PMC3816225
Ahmed D., Eide P.W., Eilertsen I.A., Danielsen S.A., Eknaes M., Hektoen M., Lind G.E., Lothe R.A.
Epigenetic and genetic features of 24 colon cancer cell lines.
Oncogenesis 2:e71.1-e71.8(2013)
PubMed=24755471; DOI=10.1158/0008-5472.CAN-14-0013
Mouradov D., Sloggett C., Jorissen R.N., Love C.G., Li S., Burgess A.W., Arango D., Strausberg R.L., Buchanan D., Wormald S., O'Connor L., Wilding J.L., Bicknell D.C., Tomlinson I.P.M., Bodmer W.F., Mariadason J.M., Sieber O.M.
Colorectal cancer cell lines are representative models of the main molecular subtypes of primary cancer.
Cancer Res. 74:3238-3247(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25926053; DOI=10.1038/ncomms8002
Medico E., Russo M., Picco G., Cancelliere C., Valtorta E., Corti G., Buscarino M., Isella C., Lamba S., Martinoglio B., Veronese S., Siena S., Sartore-Bianchi A., Beccuti M., Mottolese M., Linnebacher M., Cordero F., Di Nicolantonio F., Bardelli A.
The molecular landscape of colorectal cancer cell lines unveils clinically actionable kinase targets.
Nat. Commun. 6:7002.1-7002.10(2015)
PubMed=25944804; DOI=10.1158/1078-0432.CCR-14-2457
Bazzocco S., Dopeso H., Carton-Garcia F., Macaya I., Andretta E., Chionh F., Rodrigues P., Garrido M., Alazzouzi H., Nieto R., Sanchez A., Schwartz S. Jr., Bilic J., Mariadason J.M., Arango D.
Highly expressed genes in rapidly proliferating tumor cells as new targets for colorectal cancer treatment.
Clin. Cancer Res. 21:3695-3704(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26537799; DOI=10.1074/mcp.M115.051235; PMCID=PMC4762531
Holst S., Deuss A.J.M., van Pelt G.W., van Vliet S.J., Garcia-Vallejo J.J., Koeleman C.A.M., Deelder A.M., Mesker W.E., Tollenaar R.A.E.M., Rombouts Y., Wuhrer M.
N-glycosylation profiling of colorectal cancer cell lines reveals association of fucosylation with differentiation and caudal type homebox 1 (CDX1)/villin mRNA expression.
Mol. Cell. Proteomics 15:124-140(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28683746; DOI=10.1186/s12943-017-0691-y; PMCID=PMC5498998
Berg K.C.G., Eide P.W., Eilertsen I.A., Johannessen B., Bruun J., Danielsen S.A., Bjornslett M., Meza-Zepeda L.A., Eknaes M., Lind G.E., Myklebost O., Skotheim R.I., Sveen A., Lothe R.A.
Multi-omics of 34 colorectal cancer cell lines -- a resource for biomedical studies.
Mol. Cancer 16:116.1-116.16(2017)
PubMed=28854368; DOI=10.1016/j.celrep.2017.08.010; PMCID=PMC5583477
Roumeliotis T.I., Williams S.P., Goncalves E., Alsinet C., Del Castillo Velasco-Herrera M., Aben N., Ghavidel F.Z., Michaut M., Schubert M., Price S., Wright J.C., Yu L., Yang M., Dienstmann R., Guinney J.H., Beltrao P., Brazma A., Pardo M., Stegle O., Adams D.J., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Choudhary J.S.
Genomic determinants of protein abundance variation in colorectal cancer cells.
Cell Rep. 20:2201-2214(2017)
PubMed=29101300; DOI=10.15252/msb.20177701; PMCID=PMC5731344
Frejno M., Zenezini Chiozzi R., Wilhelm M., Koch H., Zheng R.-S., Klaeger S., Ruprecht B., Meng C., Kramer K., Jarzab A., Heinzlmeir S., Johnstone E., Domingo E., Kerr D.J., Jesinghaus M., Slotta-Huspenina J., Weichert W., Knapp S., Feller S.M., Kuster B.
Pharmacoproteomic characterisation of human colon and rectal cancer.
Mol. Syst. Biol. 13:951-951(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"