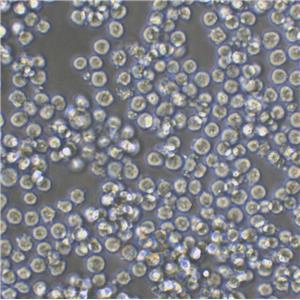
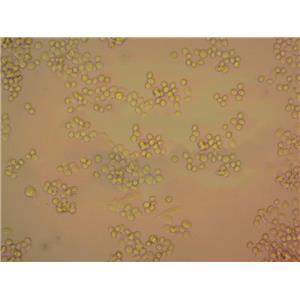
"ES-2人卵巢透明细胞癌细胞全年复苏|已有STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的应用:1)免疫组化研究2)RNA干扰研究3)药物作用研究4)慢病毒转染研究等其它应用。细胞系通常用于实验研究,如增殖、迁移、侵袭等。细胞系在多个领域的研究中被广泛应用,包括基础医学、临床试验、药物筛选和分子生物学研究。这些研究不仅在中国,也在日本、美国和欧洲等多个国家和地区进行。
换液周期:每周2-3次
GM15452 Cells;背景说明:1957年,PuckTT从成年中国仓鼠卵巢的活检组织建立了CHO细胞,CHO-K1是CHO的一个亚克隆。CHO-K1的生长需要脯酸。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs 343.T细胞、Human Hepatocyte Line 5细胞、COLO 320细胞
P3HR1-BL Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:Pan02细胞、MCA-205细胞、SK-ES-1细胞
GM04678 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:P388细胞、L cells (TK-)细胞、GCT细胞
背景信息:ES-2细胞系源于一位47岁黑人女性的临床卵巢透明细胞癌手术标本。该细胞对中低剂量的阿霉素,顺铂,双乙基亚硝脲,表鬼臼毒素吡喃葡糖苷等化疗药物有一定耐药性。该细胞少量表达糖蛋白P。
ES-2人卵巢透明细胞癌细胞全年复苏|已有STR图谱
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
细胞培养应用于分子生物学、细胞生物学、遗传学、免疫学、肿瘤学及病毒学等领域,细胞培养是指将细胞从动物或植物体内取出,然后在适宜的人工环境中生长的过程。细胞可以在培养前直接从组织中取出并通过酶或机械方法进行解离,也可以来源于已建立的细胞系。传代培养是指当细胞生长至高密度时,将其分殖至新的培养瓶中,以维持其生长和增殖。贴壁细胞是指在培养基表面附着生长的细胞,悬浮细胞是指在培养基中悬浮生长的细胞,不依附于培养皿表面。半贴壁半悬浮细胞同时具备贴壁细胞和悬浮细胞的特点,通常在培养基中部分附着生长,部分悬浮于培养基上。
KM H-2 Cells;背景说明:霍奇金淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI-H1838细胞、Kit 225细胞、AZ521细胞
MCMEC Cells;背景说明:心脏微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IB-RS2细胞、HUT28细胞、PC-3/M细胞
NTera2 cl.D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PaTu8988s细胞、253J B-V细胞、CoC1细胞
A1-425 Cells(提供STR鉴定图谱)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
物种来源:人源、鼠源等其它物种来源
ES-2人卵巢透明细胞癌细胞全年复苏|已有STR图谱
形态特性:上皮细胞样
细胞冻存复苏材料与方法步骤:常用的细胞冷冻贮存器为贮存器,规格有35L和50L两种。使用时要注意以下几点:(1)一般两周需充一次,至少一个月充一次。温度达-196℃,使用时注意勿让溅到皮肤上,以免引起冻伤。(2)容器为双层结构,中间为真空层,瓶口有双层焊接处,应防止焊接部裂开。(3)在装入时,要注意缓慢小心,并用厚纸卷筒或制漏斗作引导,使直达瓶底,如有专用灌注装置则更HAO。若为初次使用,加时更要缓慢,以免温度骤降而使容器损坏。细胞冻存时常备的材料有:0.25%胰蛋白酶,含10%~20%的血清培养,DMSO(分析纯)或无色新鲜甘油(121°C蒸气GAO压消毒),2mL安瓿(或专用细胞冻存管)、吸管、离心管、喷灯、纱布袋(或冻存管架)等。主要操作步骤为:(1)选择处于对数生长期的细胞,在冻存前一天ZuiHAO换。将多个培养瓶中的细胞培养 去掉,用0.25%胰蛋白酶消化。适时去掉胰蛋白酶,加入少量新培养。用吸管吸取培养反复吹打瓶壁上的细胞,使其成为均匀分散的细胞悬。悬浮生产细胞则不要消化处理。然后将细胞收集于离心管中离心(1000r/min,10分钟)。(2)去上清,加入含20%小牛血清的完全培养基,于4℃预冷15分钟后,逐滴加入已无菌的DMSO或甘油,用吸管轻轻吹打使细胞均匀,细胞浓度为3×106~1×107/mL之间。(3)将上述细胞分装于安瓿或专用冷冻塑料管中,安瓿装1~1.5mL在火焰喷灯上封口,封口处要完全封闭,圆滑无勾。冷冻管要将盖子盖紧,并标记HAO细胞名称和冻存日期,同时作HAO登记(日期、细胞种类及代次、冻存支数)。(4)将装HAO细胞的安瓿或冻存管装入沙布袋内;置于容器颈口处存放过夜,次日转入中。采用控制降温速度的方法也可采用下列步骤:先将安瓿置入4℃冰箱中2~3小时,再移至冰箱冷冻室内3~4小时,再吊入容器颈气态部分存放2小时,Zui后沉入中。细胞冻存在中可以长期保存,但为妥善起见,冻存半年后,ZuiHAO取出一只安瓿细胞复苏培养,观察生长情况,然后再继续冻存。
MonoMac 6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:MDA-MB-231-luc细胞、LA795细胞、AMC-HN-8细胞
H-1395 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;5-6天传代一次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:A549-Taxol细胞、NCI-322细胞、UO-31细胞
H1975 Cells;背景说明:这株细胞于1988年七月建株。组织提供者是一位非吸烟人士。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:GM0637细胞、Spodoptera frugiperda clone 9细胞、Human Epidermoid carcinoma #2细胞
Kuramochi Cells;背景说明:详见相关文献介绍;传代方法:1:10传代;每周换液2-3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCI-AML-4细胞、OP9细胞、SNU-C1细胞
EJ 138 Cells;背景说明:膀胱癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IEC18细胞、MV(4;11)细胞、MSCs(mUCMSCs)细胞
SUDHL-6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MCF-10 Attached细胞、University of Michigan-Renal Carcinoma-2细胞、RGC-5细胞
MonoMac 6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:MDA-MB-231-luc细胞、LA795细胞、AMC-HN-8细胞
DMS 79 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:SH-SY5Y Parental细胞、Cates-1B细胞、HN6细胞
GL261 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:GM02131细胞、SK-N-MC细胞、HuH-1细胞
NOZC-1 Cells;背景说明:患者有癌性腹膜炎。细胞为中等分化的管状胆囊癌。会分泌AFP和CEA。倍增时间48小时,板植率14-19%。细胞可在裸鼠中成瘤,形态与原发肿瘤相似。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:F442A细胞、MFE-280细胞、Hs 636.T细胞
S-16 Cells;背景说明:Schwann细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs578Bst细胞、Vero 76 clone E-6细胞、QGY 7701细胞
210RCY3-Ag1.2.3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C32 [Human melanoma]细胞、QSG-7701细胞、NCIH650细胞
Mink Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ROS 17/28细胞、SUIT2细胞、Human Microglia Clone 3细胞
VB Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:NCI-H526细胞、H-2452细胞、CCC-HEH-2细胞
TE-12 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKNBE细胞、U2-OS细胞、SuDHL 1细胞
SKRC-52 Cells;背景说明:肾癌;纵隔膜转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:VA-ES-BJ细胞、Ly19细胞、Sci-1细胞
CAL-27 Cells;背景说明:该细胞1982年由J. Gioanni建系,源自一位56岁白人男性的舌头中倍的病变部位,角蛋白强阳性;传代方法:1:6传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:CAL-51细胞、My-La 2059细胞、NCI-747细胞
Abcam A-549 CD14 KO Cells(提供STR鉴定图谱)
Abcam Raji UPP1 KO Cells(提供STR鉴定图谱)
BayGenomics ES cell line BGC456 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRS228 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA435 Cells(提供STR鉴定图谱)
chHES-103 Cells(提供STR鉴定图谱)
DA01907 Cells(提供STR鉴定图谱)
EDi017-A Cells(提供STR鉴定图谱)
GM05396 Cells(提供STR鉴定图谱)
Huh-7.5.1 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L-cells细胞、NPA87细胞、SMA-560细胞
P3/ag Cells;背景说明:骨髓瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HECCL-1细胞、NCI-HUT-292细胞、B-cell with DC Morphology细胞
HCC-1937 Cells;背景说明:这株细胞1995年10月13日最初来源于原发性导管癌, 用了11.5个月建株。肿瘤分类为TNM IIB期, 3级。BRCA1分析表明这株细胞是BRCA1 5382C突变纯合的, 而来源于同一病人的类淋巴母细胞细胞株在这个突变位点上是杂合的。 另两个家庭成员也有这个突变; 一个同卵双生姐妹也患有乳腺癌。这株细胞有一个后天的TP53突变, 而其野生型等位基因丢失; 一个PTEN基因的后天的纯合缺失, 以及多个与乳腺癌发病机理相关的位点上发生的杂合突变。这株细胞Her2-neu和p53表达都呈阴性。;传代方法:1:2传代;4-5天传代一次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:RBMEC细胞、OE21细胞、MDAMB330细胞
LLC-MK2 Cells;背景说明:胚胎;肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Primary Liver Carcinoma/Poliomyelitis Research Foundation/5细胞、PC3细胞、L-M (TK-)细胞
OCILY7 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SNU-520细胞、PT-67细胞、Hs839T细胞
HCC9724 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BIU87细胞、MRAEC细胞、T-47D细胞
PC9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CNE1细胞、HME-1细胞、SU-DHL-6细胞
BRL3A Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:WEHI-164细胞、NTera-2D1细胞、COLO-205细胞
ES-2人卵巢透明细胞癌细胞全年复苏|已有STR图谱
GM-3573 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:CCD18细胞、COLO-394细胞、CCRF-CEM C7细胞
C3H10T1-2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:V79-1细胞、BT 325细胞、Stanford University-Diffuse Histiocytic Lymphoma-5细胞
SKG3A Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:IOSE29细胞、B-3细胞、SW-48细胞
alpha TC1.6 Cells;背景说明:胰岛素瘤;a细胞;C57BL/6xDBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HO1N1细胞、MIN-6细胞、BC-024细胞
Helacyton gartleri Cells;背景说明:HeLa是第一个来自人体组织经连续培养获得的非整倍体上皮样细胞系,它由GeyGO等在1951年从31岁女性黑人的宫颈癌组织建立。经原始组织切片重新观察,Jones等将其诊断为腺癌。已知该细胞系含有人乳头状瘤病毒HPV18序列,需在2级生物安全防护台操作。该细胞角蛋白阳性,p53表达量较低,但表达正常水平的pRB(视网膜母细胞瘤抑制因子)。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCGC细胞、VP229细胞、RBL.2H3细胞
EC-GI Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MV-3细胞、PK13细胞、293-GP细胞
ATDC5 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:L23/P细胞、RGC-6细胞、Calf Pulmonary Artery 47细胞
GM28079 Cells(提供STR鉴定图谱)
HAP1 PCCA (-) 1 Cells(提供STR鉴定图谱)
LTEP-a2 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:S37细胞、Intestinal Porcine Epithelial Cell line-1细胞、WRL68细胞
A375mel Cells;背景说明:A375源自一位54岁女性,是Giard DJ等人建立的一系列细胞株中的一株。该细胞可在免疫抑制小鼠上成瘤,在琼脂上形成克隆。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:SKLMS-1细胞、SGC-7901细胞、JHH2细胞
Hs 729 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:SK Hep1细胞、YES-2细胞、JB-6 Cl 30细胞
UCLA RO-81-A-1 Cells;背景说明:甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:293 EBNA细胞、Molm 13细胞、SUDHL8细胞
SNU878 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L cell细胞、CMECs细胞、A-875细胞
PLMVEC Cells;背景说明:肺微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JB6 clone 30, subclone 7b细胞、HTh-74细胞、L(TK-)细胞
CAL39 Cells;背景说明:外阴鳞癌细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-UT-1细胞、RBE细胞、BT-20细胞
J111 Cells;背景说明:单核细胞白血病;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCIH650细胞、Hs819T细胞、University of Michigan-Urothelial Carcinoma-14细胞
HS181 EGFP-2A-RUNX1c Cells(提供STR鉴定图谱)
KOLF2.1J KIF5A N256S SNV/WT Cells(提供STR鉴定图谱)
MN-R Cells(提供STR鉴定图谱)
NUIGi047-B Cells(提供STR鉴定图谱)
RG-226 Cells(提供STR鉴定图谱)
Ubigene A-549 TAOK2 KO Cells(提供STR鉴定图谱)
V79 A3 Cells(提供STR鉴定图谱)
HCT116-MFSD2B-KO-c17 Cells(提供STR鉴定图谱)
IPLB-SF 21 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HITT15细胞、RIN Cl-5F细胞、CLL-CII细胞
IGR-OV1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1734细胞、EMT6细胞、MFD-1细胞
P560 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HuT-78细胞、H889细胞、OVCAR-10细胞
P3-X63-Ag8-653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H250细胞、JKT1细胞、SJSA细胞
KYSE-510 Cells;背景说明:食管鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR432细胞、KE37细胞、B78细胞
KYSE-510 Cells;背景说明:食管鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR432细胞、KE37细胞、B78细胞
SCLC-21H Cells;背景说明:小细胞肺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SPCA-1细胞、NCIH2107细胞、231-luc细胞
RCC4 Cells;背景说明:肾透明细胞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KM933细胞、NCI-SNU-423细胞、HEK 293-F细胞
MRC-V Cells;背景说明:MRC-5细胞系来自14周龄男性胎儿的正常肺组织,该细胞老化前能传代42~46个倍增时间。;传代方法:1:2-1:5传代;每周1-2次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Human Pancreatic Duct Epithelial细胞、HSC 3细胞、HO8910细胞
Mv 1 Lu (NBL-7) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BCaP 37细胞、H-1944细胞、NCI-UMC-11细胞
KYSE-270 Cells;背景说明:详见相关文献介绍;传代方法:1:5传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MCA 38细胞、NCI-H2108细胞、GM02132C细胞
L cell Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C38细胞、CHO-K1细胞、Hep-G2细胞
HEL 299 Cells;背景说明:红白细胞白血病;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:H2085细胞、MM.1S细胞、L-5178-Y细胞
SF17 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC2157细胞、OCI-Ly3细胞、SK-N-BE-2细胞
huH 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Lewis lung carcinoma细胞、D283 Med细胞、343MG细胞
STBCi305-A Cells(提供STR鉴定图谱)
T-T Cells;背景说明:详见相关文献介绍;传代方法:10^5 cells/60mm dish;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:CWR22-Rv1细胞、32D clone3细胞、SBC-5细胞
Mv.1.Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:YAC细胞、BC-027细胞、LICCF细胞
WEHI-279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P31FUJ细胞、LICCF细胞、PC14细胞
Ball 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:KM-H2细胞、HMEC1细胞、GM03569B细胞
SW982 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-436细胞、HEI-193细胞、HS 1.Tes细胞
ZR75-1 Cells;背景说明:该细胞产生高水平的黏液素MUC-1 mRNA,低水平的MUC-2 mRNA,但不表达MUC-3基因;表达雌激素受体。;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MSB-1细胞、SF126细胞、Large Cell Lung Cancer-103H细胞
MDA330 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Pt K2细胞、PG [Human lung carcinoma]细胞、ROS-17/2.8细胞
F-36P Cells;背景说明:详见相关文献介绍;传代方法:每周2次换液;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hepa 1-6细胞、NB9细胞、PLMVEC细胞
ES-2人卵巢透明细胞癌细胞全年复苏|已有STR图谱
mRMEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:A375mel细胞、SCL-II细胞、SK-GT-2细胞
SUD4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:HNTEC细胞、RPTEC/TERT 1细胞、MOLP-2细胞
FDC-P1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:CMT-93细胞、NCI-H740细胞、KM-H2细胞
Centre Antoine Lacassagne-12T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDA MB 134VI细胞、293细胞、N-Tera-2细胞
A2058 Cells;背景说明:详见相关文献介绍;传代方法:1:6-1:12传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:COV 504细胞、NCI-446细胞、CEM/C1细胞
rRTEC Cells;背景说明:肾小管;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Nthy-ori 3.1细胞、Capan 2细胞、VK2/E6E7细胞
BayGenomics ES cell line RRD071 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XE800 Cells(提供STR鉴定图谱)
DA5C7 Cells(提供STR鉴定图谱)
MK/T-1 Cells(提供STR鉴定图谱)
SZ-179 Cells(提供STR鉴定图谱)
Lu-130 Cells(提供STR鉴定图谱)
" "PubMed=12960427; DOI=10.1091/mbc.E03-05-0279; PMCID=PMC266758
Schaner M.E., Ross D.T., Ciaravino G., Sorlie T., Troyanskaya O.G., Diehn M., Wang Y.C., Duran G.E., Sikic T.L., Caldeira S., Skomedal H., Tu I.-P., Hernandez-Boussard T., Johnson S.W., O'Dwyer P.J., Fero M.J., Kristensen G.B., Borresen-Dale A.-L., Hastie T.J., Tibshirani R., van de Rijn M., Teng N.N.H., Longacre T.A., Botstein D., Brown P.O., Sikic B.I.
Gene expression patterns in ovarian carcinomas.
Mol. Biol. Cell 14:4376-4386(2003)
PubMed=16382445; DOI=10.1002/gcc.20300
Wang Y.C., Juric D., Francisco B., Yu R.X., Duran G.E., Chen G.K., Chen X., Sikic B.I.
Regional activation of chromosomal arm 7q with and without gene amplification in taxane-selected human ovarian cancer cell lines.
Genes Chromosomes Cancer 45:365-374(2006)
PubMed=17671176; DOI=10.1158/0008-5472.CAN-06-4567
Kikuchi R., Tsuda H., Kanai Y., Kasamatsu T., Sengoku K., Hirohashi S., Inazawa J., Imoto I.
Promoter hypermethylation contributes to frequent inactivation of a putative conditional tumor suppressor gene connective tissue growth factor in ovarian cancer.
Cancer Res. 67:7095-7105(2007)
PubMed=18560578; DOI=10.1371/journal.pone.0002425; PMCID=PMC2409963
Faca V.M., Ventura A.P., Fitzgibbon M.P., Pereira-Faca S.R., Pitteri S.J., Green A.E., Ireton R.C., Zhang Q., Wang H., O'Briant K.C., Drescher C.W., Schummer M., McIntosh M.W., Knudsen B.S., Hanash S.M.
Proteomic analysis of ovarian cancer cells reveals dynamic processes of protein secretion and shedding of extra-cellular domains.
PLoS ONE 3:E2425-E2425(2008)
PubMed=18720132; DOI=10.1080/10717540802329183
Serikawa T., Kikuchi H., Oite T., Tanaka K.
Enhancement of gene expression efficiency using cationic liposomes on ovarian cancer cells.
Drug Deliv. 15:523-529(2008)
PubMed=20081105; DOI=10.1210/me.2009-0295; PMCID=PMC2817607
Nagaraja A.K., Creighton C.J., Yu Z.-F., Zhu H.-F., Gunaratne P.H., Reid J.G., Olokpa E., Itamochi H., Ueno N.T., Hawkins S.M., Anderson M.L., Matzuk M.M.
A link between mir-100 and FRAP1/mTOR in clear cell ovarian cancer.
Mol. Endocrinol. 24:447-463(2010)
PubMed=20204287; DOI=10.3892/or_00000728; PMCID=PMC2909445
Langland G.T., Yannone S.M., Langland R.A., Nakao A., Guan Y.-H., Long S.B.T., Vonguyen L., Chen D.J., Gray J.W., Chen F.-Q.
Radiosensitivity profiles from a panel of ovarian cancer cell lines exhibiting genetic alterations in p53 and disparate DNA-dependent protein kinase activities.
Oncol. Rep. 23:1021-1026(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22328975; DOI=10.1158/2159-8290.CD-11-0170; PMCID=PMC3274821
Hanrahan A.J., Schultz N., Westfal M.L., Sakr R.A., Giri D.D., Scarperi S., Janakiraman M., Olvera N., Stevens E.V., She Q.-B., Aghajanian C., King T.A., de Stanchina E., Spriggs D.R., Heguy A., Taylor B.S., Sander C., Rosen N., Levine D.A., Solit D.B.
Genomic complexity and AKT dependence in serous ovarian cancer.
Cancer Discov. 2:56-67(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22705003; DOI=10.1016/j.humpath.2012.03.011
Rahman M., Nakayama K., Rahman M.T., Nakayama N., Ishikawa M., Katagiri A., Iida K., Nakayama S., Otsuki Y., Shih I.-M., Miyazaki K.
Clinicopathologic and biological analysis of PIK3CA mutation in ovarian clear cell carcinoma.
Hum. Pathol. 43:2197-2206(2012)
PubMed=22710073; DOI=10.1016/j.ygyno.2012.06.017; PMCID=PMC3432677
Korch C.T., Spillman M.A., Jackson T.A., Jacobsen B.M., Murphy S.K., Lessey B.A., Jordan V.C., Bradford A.P.
DNA profiling analysis of endometrial and ovarian cell lines reveals misidentification, redundancy and contamination.
Gynecol. Oncol. 127:241-248(2012)
PubMed=23415752; DOI=10.1016/j.molonc.2012.12.007; PMCID=PMC4106023
Stordal B., Timms K., Farrelly A., Gallagher D., Busschots S., Renaud M., Thery J., Williams D., Potter J., Tran T., Korpanty G., Cremona M., Carey M.S., Li J., Li Y., Aslan O., O'Leary J.J., Mills G.B., Hennessy B.T.
BRCA1/2 mutation analysis in 41 ovarian cell lines reveals only one functionally deleterious BRCA1 mutation.
Mol. Oncol. 7:567-579(2013)
PubMed=23839242; DOI=10.1038/ncomms3126; PMCID=PMC3715866
Domcke S., Sinha R., Levine D.A., Sander C., Schultz N.
Evaluating cell lines as tumour models by comparison of genomic profiles.
Nat. Commun. 4:2126.1-2126.10(2013)
PubMed=24023729; DOI=10.1371/journal.pone.0072162; PMCID=PMC3762837
Anglesio M.S., Wiegand K.C., Melnyk N., Chow C., Salamanca C.M., Prentice L.M., Senz J., Yang W., Spillman M.A., Cochrane D.R., Shumansky K., Shah S.P., Kalloger S.E., Huntsman D.G.
Type-specific cell line models for type-specific ovarian cancer research.
PLoS ONE 8:E72162-E72162(2013)
PubMed=25049276; DOI=10.1136/jclinpath-2014-202430
Kwok A.L.M., Wong O.G.-W., Wong E.S.Y., Tsun O.K.-L., Chan K.-K., Cheung A.N.-Y.
Caution over use of ES2 as a model of ovarian clear cell carcinoma.
J. Clin. Pathol. 67:921-922(2014)
PubMed=25230021; DOI=10.1371/journal.pone.0103988; PMCID=PMC4167545
Beaufort C.M., Helmijr J.C.A., Piskorz A.M., Hoogstraat M., Ruigrok-Ritstier K., Besselink N., Murtaza M., van IJcken W.F.J., Heine A.A.J., Smid M., Koudijs M.J., Brenton J.D., Berns E.M.J.J., Helleman J.
Ovarian cancer cell line panel (OCCP): clinical importance of in vitro morphological subtypes.
PLoS ONE 9:E103988-E103988(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26388441; DOI=10.1016/j.cell.2015.08.056; PMCID=PMC4644236
Creixell P., Schoof E.M., Simpson C.D., Longden J., Miller C.J., Lou H.J., Perryman L., Cox T.R., Zivanovic N., Palmeri A., Wesolowska-Andersen A., Helmer-Citterich M., Ferkinghoff-Borg J., Itamochi H., Bodenmiller B., Erler J.T., Turk B.E., Linding R.
Kinome-wide decoding of network-attacking mutations rewiring cancer signaling.
Cell 163:202-217(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28273451; DOI=10.1016/j.celrep.2017.02.028
Medrano M., Communal L., Brown K.R., Iwanicki M., Normand J., Paterson J., Sircoulomb F., Krzyzanowski P.M., Novak M., Doodnauth S.A., Suarez Saiz F.J., Cullis J., Al-awar R., Neel B.G., McPherson J., Drapkin R.I., Ailles L., Mes-Masson A.-M., Rottapel R.
Interrogation of functional cell-surface markers identifies CD151 dependency in high-grade serous ovarian cancer.
Cell Rep. 18:2343-2358(2017)
PubMed=30485824; DOI=10.1016/j.celrep.2018.10.096; PMCID=PMC6481945
Papp E., Hallberg D., Konecny G.E., Bruhm D.C., Adleff V., Noe M., Kagiampakis I., Palsgrove D., Conklin D., Kinose Y., White J.R., Press M.F., Drapkin R.I., Easwaran H., Baylin S.B., Slamon D.J., Velculescu V.E., Scharpf R.B.
Integrated genomic, epigenomic, and expression analyses of ovarian cancer cell lines.
Cell Rep. 25:2617-2633(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"


![ES-2[人卵巢癌细胞]](https://img.chemicalbook.com/SupplyImg/2019-08-28/Large/201908280634521799877.jpg)


