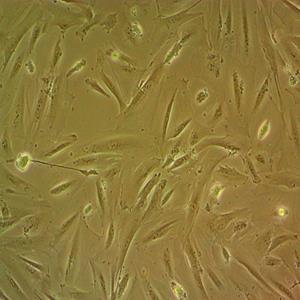
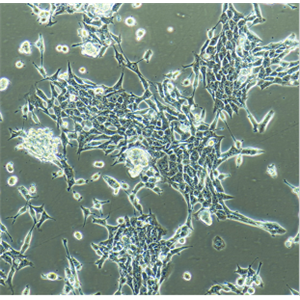
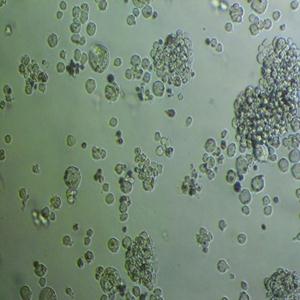
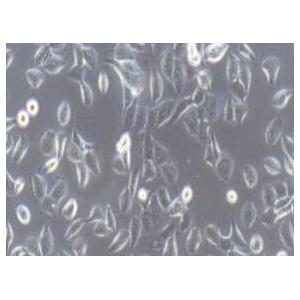
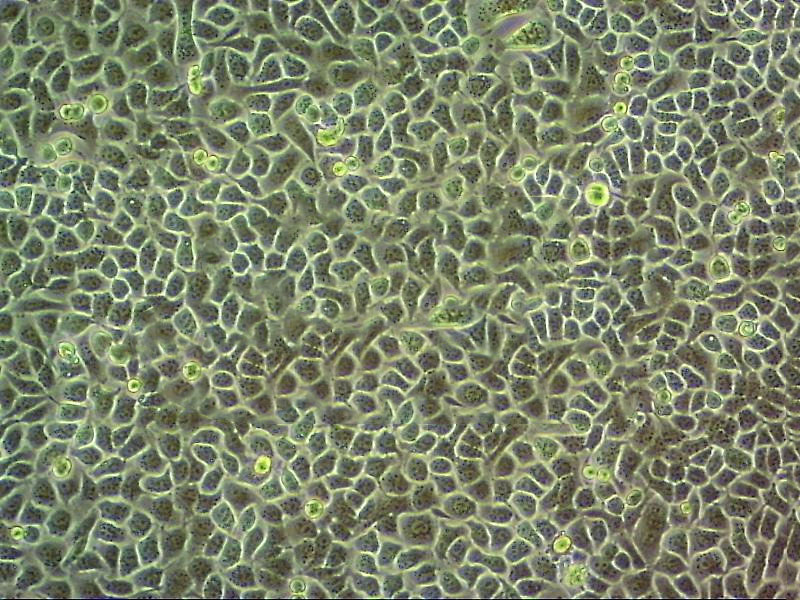
"SUIT-2人胰腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
H1373 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Opossum Kidney细胞、SUP-T1细胞、JEKO细胞
Hs-675-T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:HO8910细胞、C4 I细胞、RPMI-8226S细胞
OVCAR.3 Cells;背景说明:该细胞1982年由T.C. Hamilton等建系,源自一位60卵巢腺癌的腹水,是卵巢癌抗药性研究的模型。;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Vero C1008细胞、SKNEP1细胞、Madin Darby Canine Kidney细胞
SUIT-2人胰腺癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
SUIT-2人胰腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
NCI-H2066 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:Panc_08_13细胞、Hs606T细胞、3T3细胞
Alexander Cells;背景说明:该细胞系分泌乙肝病毒表面抗原(HBsAg)。 此细胞系原先被支原体污染,后用BM-cycline去除支原体;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:IPLB-SF-21-AE细胞、95D细胞、Sp2/0-Ag-14细胞
CCC-HIE-2 Cells;背景说明:胚胎;肠粘膜;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HEK293EBNA细胞、293 H细胞、Gejiu Lung Carcinoma-82细胞
HCC1428 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:贴壁生长,偶尔上皮细胞液泡的形成;形态特性:上皮细胞样;相关产品有:P-388细胞、CTLL-2细胞、NR8383.1细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
贴壁细胞消化传代时通常采用两种方法:一、加入胰酶等细胞脱落后,再加培养基中止胰酶作用,离心传代;二、加入胰酶后,镜下观察待细胞始脱落时,弃胰酶,加培养分瓶。但前者太麻烦,而后者有可能对细胞施加胰酶选择,因为总是贴壁不牢的细胞先脱落,对肿瘤细胞来说,这部分细胞有可能是恶性程度较GAO的细胞亚群。一种简单的消化传代方法。加入PBS洗去血清或加入胰酶先中和血清的作用(30s),弃之,再加入适量胰酶作用10s-40s(根据细胞消化的难易程度),弃之,这样依赖残余的胰酶就可将细胞消化单细胞。对于较难消化的细胞,可以用2%利多卡因消化5-8分钟,然后再弃去,加培养基吹打也可以,对细胞的影响不大。不用PBS也不用Hanks洗,只要把旧培养吸的干净一点,直接加酶消化应该不会有什么问题。弃培养后,用0.04%的EDA冲洗一次,再用1/4v的0.04%的EDA室温孵育5min,弃取大部分EDA,加入与剩余EDA等量的胰酶(预热)总体积1/10v。消化到有细胞脱落。不过有人说EDA对细胞不HAO,有证据吗?培养的BASMC:倒掉旧培养加入少量胰酶冲一下,倒掉再加入0.125-0.25%胰酶约6-10滴或1ml(25ml bole)消化再加入适量新培养基中和,并分瓶这种方法简单、省事;效果很HAO并且不损失细胞!
ChaGo K-1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:UCH1细胞、HIT clone T15细胞、KYSE 510细胞
CW-2 Cells;背景说明:来源于结肠癌。 CEA阳性,移植于裸鼠可成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCIH2066细胞、C-4 I细胞、Rat Chondrosarcoma Swarm细胞
NS653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1869细胞、LAPC-4细胞、PLA-802细胞
hSCC-25 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SF 295细胞、K1735细胞、HDF-a细胞
32Dc3 Cells;背景说明:骨髓淋巴瘤;C3H/HeJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:L-M(TK-)细胞、BMSCs(mBMSCs)细胞、BE(2)M-17细胞
MARC 145 Cells;背景说明:胚肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CoCL2细胞、CP70细胞、T-T细胞
H596 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:GA-10(Clone 4)细胞、CCD 19Lu细胞、Dakiki细胞
SW1222 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HRA 19细胞、Normal Rat Kidney-49F细胞、HCC941122细胞
hA549 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MOLT16细胞、OVCA420细胞、Hs863T细胞
Intestinal Porcine Epithelial Cell line-1 Cells;背景说明:小肠;上皮细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR-5细胞、A-101D细胞、FCCH1018细胞
MXI Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Earles's cells细胞、HET-1A细胞、HepG2-luc细胞
TW-039 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RT-4细胞、102PT细胞、GalK 1细胞
Detroit562 Cells;背景说明:器官:咽头 疾病:癌 取材转移灶:胸水;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NALM-6细胞、OE-33细胞、UCD-MLA-144细胞
Panc3_27 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:PG13细胞、Hs 746.T细胞、SVG p12细胞
NWA Cells;背景说明:这株细胞有EB病毒基因组。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NCI H2106细胞、OCI AML3细胞、Natural Killer-92细胞
Rat Basophilic Leukemia-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Sp2/O-Ag14细胞、PG-LH7细胞、CAL-27细胞
CP-70 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MHCC97-H细胞、SNU-5细胞、UMC-11细胞
4a-3B Cells(提供STR鉴定图谱)
Abcam Jurkat TRPM2 KO Cells(提供STR鉴定图谱)
AMJ2-C20 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRK204 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XL796 Cells(提供STR鉴定图谱)
CAK [Cromileptes] Cells(提供STR鉴定图谱)
DA00561 Cells(提供STR鉴定图谱)
DD2031 Cells(提供STR鉴定图谱)
GM02048 Cells(提供STR鉴定图谱)
NCI-H847 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Namalwa细胞、NCI-H-128细胞、IPLB-SF 21AE细胞
SUIT-2人胰腺癌细胞代次低|培养基|送STR图谱
PC-14 Cells;背景说明:肺腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW 982细胞、DV90细胞、C33A细胞
H1568 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MSTO-211细胞、NCIH1694细胞、H-676细胞
AN-3 Cells;背景说明:AN3CA细胞建系于1964年。它衍生于子宫内膜癌患者淋巴结转移组织,具有癌细胞的基本特性,能在体外长期传代培养,接种实验动物产生明显肿瘤。但细胞的生物学特性及超微结构尚未深入研究,仅发现该细胞系促黑激素合成为阴性。细胞常用于人子宫内膜癌细胞生物学及其相关特性研究。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:U373MG细胞、COLO 320细胞、HCM细胞
NCI-H209 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:悬浮生长,有少数细胞疏松贴壁;形态特性:上皮样;相关产品有:RA.1细胞、H1693细胞、KBM-7细胞
NRK clone 49F Cells;背景说明:肾;成纤维细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H322T细胞、OVCA-420细胞、OE21细胞
41-2 Cells(提供STR鉴定图谱)
RIN-m 14B Cells;背景说明:胰岛素瘤;雄性;NEDH;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW-1783细胞、SHG44细胞、SupB15W细胞
NK92MI Cells;背景说明:NK细胞;淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:KYSE 450细胞、Biologics Standards-Cercopithecus-1细胞、Lu99A细胞
IMR-90 Cells;背景说明:W.W. Nichols及其同事从一位16周女婴的肺中取材,建立了人二倍体成纤维细胞株IMR-90。分裂潜能,病毒感受性和其它性质都得到了充分研究,因而这株细胞可以作为WI-38或其它标准人肺细胞株的替代株。有报道称这株细胞在表现出衰老前可倍增58次。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:LC1细胞、WILL2细胞、MIN6细胞
GM03573A Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Panc 5.04细胞、NOMO1细胞、LLC-PK1细胞
H647 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:DMS-273细胞、FRhK4细胞、Reuber-H-35 hepatoma细胞
CTX TNA2 Cells;背景说明:星形胶质细胞;SV40转化;SD;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JB-6 Cl 30细胞、USMC细胞、C-32细胞
NCIH1944 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:KMM1细胞、H295R-S1细胞、HCC9204细胞
WEHI-231 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H2170细胞、CCC-HPF-1细胞、SW-900细胞
GM13670 Cells(提供STR鉴定图谱)
HAP1 FUNDC2 (-) 5 Cells(提供STR鉴定图谱)
3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Me-Wo细胞、WERIRb1细胞、Line 207细胞
NH6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:MonoMac6细胞、WERI细胞、HDQ-P1细胞
RMC Cells;背景说明:肾系膜;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NBL-2细胞、NCI-H1355细胞、Hs343T细胞
PG-BE1 Cells;背景说明:肺巨细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-1792细胞、SUPT-1细胞、H510A细胞
FHCRC subclone 11 Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:3T3细胞、OCM-1A细胞、ARO81细胞
ECV-304 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:National Medical Center-Glioma 1细胞、F56 [Human neoplasm]细胞、SDBMSC细胞
Clone 15 HL-60 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在1×105-1×106/ml,每2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Hs-578Bst细胞、CW-2细胞、KU-19-19细胞
CMT.64 Cells;背景说明:肺腺癌;雌性;C57;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Mv.1.Lu细胞、NCI-H2135细胞、Mel-RM细胞
HML/SE Cells(提供STR鉴定图谱)
ISMMSi051-A Cells(提供STR鉴定图谱)
MCF-7:CFR Cells(提供STR鉴定图谱)
ND13625 Cells(提供STR鉴定图谱)
PPMI.I.1051.5 Cells(提供STR鉴定图谱)
Ubigene HeLa CD47 KO Cells(提供STR鉴定图谱)
WTC-mEGFP-DSP-cl65 Cells(提供STR鉴定图谱)
HG01379 Cells(提供STR鉴定图谱)
MN 60 Cells;背景说明:B细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI-H719细胞、639-V细胞、MZCRC1细胞
LTEPa2 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU-484细胞、PTK-1细胞、MADB106细胞
no.11 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCE-2 [50.B1]细胞、THC-8307细胞、HCA-7细胞
SKLU-1 Cells;背景说明:该细胞系源于一位60岁的白人女性患者的肺腺癌组织。;传代方法:1:2传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SK-Mel1细胞、WEHI 3细胞、D283-MED细胞
hTERT RPE1 Cells;背景说明:视网膜色素上皮;hTERT永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPMI-8226细胞、MHCC97-H细胞、H-2108细胞
hTERT RPE1 Cells;背景说明:视网膜色素上皮;hTERT永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPMI-8226细胞、MHCC97-H细胞、H-2108细胞
FD-LSC-1 Cells;背景说明:喉鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DI-TNC1细胞、DK-MG细胞、C127细胞
HT-55 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BT-474细胞、NS1/1-Ag4.1细胞、HLEB-3细胞
P3 (Jiyoye) Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:hMSC-BM细胞、CCD-1095Sk细胞、B-95-8细胞
R3/1 Cells;背景说明:肺;Ⅰ 型上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JS1细胞、NK-10A细胞、HSC2细胞
HT 29 Cells;背景说明:该细胞是1964年由FoghJ用移植培养方法和含15%FBS的F12培养液从原发性肿瘤分离的。近来,已建株的培养细胞用含血清的McCoy's5a培养基培养。该细胞系在裸鼠中成瘤,也能在类固醇处理的地鼠中成瘤。该细胞可合成IgA、CEA、TGFβ结合蛋白和黏液素;表达尿激酶受体,但没有检测到血浆酶原活性;不表达CD4,但细胞表面表达半乳糖神经酰胺(HIV的可能替代受体)。该细胞系癌基因c-myc、K-ras、H-ras、N-ras、Myb、sis、fos阳性;p53基因过表达,并且在273位密码子处发;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:TM4细胞、G361-mel细胞、SRA01/04 (HLE)细胞
hTERTHME1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SK-MEL-3细胞、PaCa2细胞、LY-R细胞
SW 1222 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-2029细胞、T. T细胞、CAL 51细胞
H220 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-676细胞、L cells细胞、EL 4细胞
OCILY-19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HLEB3细胞、SUM149细胞、CV-1细胞
SIHN-013/2 Cells(提供STR鉴定图谱)
PANC3.27 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BT-B细胞、AU 565细胞、SNGM细胞
SB Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SU8686细胞、KG-1细胞、Tu 177细胞
H2.35 Cells;背景说明:肝;SV40转化;雌性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LNT-229细胞、CCD1112Sk细胞、Karpas 299细胞
ST-2 Cells;背景说明:基质;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW 756细胞、HEK-293-H细胞、JOSK-M细胞
H1666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长,松散;形态特性:上皮细胞;相关产品有:Hs 888.Lu细胞、H-2122细胞、LA795细胞
Neukoplast Cells;背景说明:NK细胞;淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HKF细胞、Laboratory of Allergic Diseases 2细胞、TOV21G细胞
SUIT-2人胰腺癌细胞代次低|培养基|送STR图谱
HTori3 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM637A细胞、MDA330细胞、P30/Ohkubo细胞
X63 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MPASMC细胞、C8-D1A细胞、SU4细胞
SNK6 Cells;背景说明:NK/T细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:BJA-B1细胞、H69C细胞、CCC-HPF-1细胞
COLO 320 HSR Cells;背景说明:该细胞1984年建系,源自一位33岁患有大肠腺癌男性经5-fu治疗后的腹水。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:详见产品说明书;相关产品有:DR2R1610细胞、MARC145细胞、A375.S2细胞
HCC-2218 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮生长;形态特性:上皮样;相关产品有:WRL 68细胞、A 2780 CP细胞、H3255细胞
HHUA Cells;背景说明:子宫内膜癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U266BL细胞、L(TK-)细胞、KP-4细胞
OUMS23 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TC-1 [Mouse lung]细胞、CAL62细胞、Primary Liver Carcinoma/Poliomyelitis Research Foundation/5细胞
HPDE Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:MDCC MSB1细胞、CA922细胞、CPA 47细胞
BayGenomics ES cell line RRJ162 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XK148 Cells(提供STR鉴定图谱)
FLC clone M2 Cells(提供STR鉴定图谱)
NIT-1 Cells(提供STR鉴定图谱)
WB6b Cells(提供STR鉴定图谱)
MCF10A-EGFR1m-12 Cells(提供STR鉴定图谱)
" "PubMed=11169959; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1049>3.0.CO;2-C
Sirivatanauksorn V., Sirivatanauksorn Y., Gorman P.A., Davidson J.M., Sheer D., Moore P.S., Scarpa A., Edwards P.A.W., Lemoine N.R.
Non-random chromosomal rearrangements in pancreatic cancer cell lines identified by spectral karyotyping.
Int. J. Cancer 91:350-358(2001)
PubMed=11787853; DOI=10.1007/s004280100474
Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=11854916; DOI=10.3748/wjg.v7.i6.855; PMCID=PMC4695609
Liu B., Staren E., Iwamura T., Appert H., Howard J.
Taxotere resistance in SUIT. Taxotere resistance in pancreatic carcinoma cell line SUIT 2 and its sublines.
World J. Gastroenterol. 7:855-859(2001)
PubMed=12800145; DOI=10.1002/gcc.10218
Adelaide J., Huang H.-E., Murati A., Alsop A.E., Orsetti B., Mozziconacci M.-J., Popovici C., Ginestier C., Letessier A., Basset C., Courtay-Cahen C., Jacquemier J., Theillet C., Birnbaum D., Edwards P.A.W., Chaffanet M.
A recurrent chromosome translocation breakpoint in breast and pancreatic cancer cell lines targets the neuregulin/NRG1 gene.
Genes Chromosomes Cancer 37:333-345(2003)
PubMed=19077451; DOI=10.1159/000178871
Harada T., Chelala C., Crnogorac-Jurcevic T., Lemoine N.R.
Genome-wide analysis of pancreatic cancer using microarray-based techniques.
Pancreatology 9:13-24(2009)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"