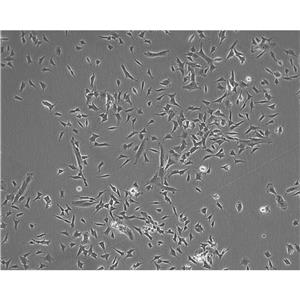
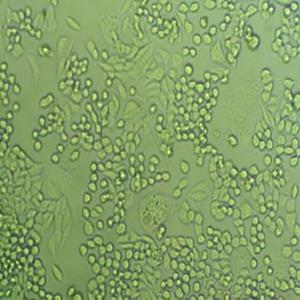
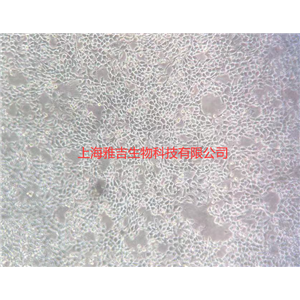
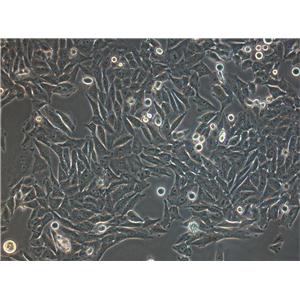
"HCC1143人乳腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
当T25瓶复苏细胞收到货时,请观察好细胞状态后,将T25细胞瓶壁进行75%酒精消毒,将T25瓶置于37度培养箱放置2-4h,以便稳定细胞状态,当细胞密度达80%-90%,即可进行首次传代培养;干冰运输的细胞冻存管收到货后,需立即转入液氮保存或直接进行复苏(第三天换液并检查复苏细胞密度,以便进行下一步)。 能够在实验室条件下进行大量培养和繁殖。这种细胞系在分子生物学和生物技术研究中十分常用。
换液周期:每周2-3次
H-1703 Cells;背景说明:该细胞1987年建系,源自一位54岁患有非小细胞肺癌的白人男性,该患者为吸烟者。;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Porcine Kidney-13细胞、IOSE 80细胞、U343MG细胞
H2196 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LA-N-6(OAN)细胞、MC/CAR细胞、IGR-OV1细胞
RPMI-8226 Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Human Epithelioma-2细胞、CCRF-CEM细胞、Turbot Embryonic Cell line细胞
HCC1143人乳腺癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
绝大部分细胞消化只要用胰酶润洗一遍即可:吸去胰酶后,残留的那些无法计算体积的附着在细胞表面的微量胰酶在37℃一般不到2min足够消化细胞(绝大部分1min不到)。对于这些细胞原则上不要用胰酶孵育细胞,连续这样传代,对细胞伤害很大。简单的程序是PBS润洗吸去,胰酶润洗吸去,然后37℃消化。什么算是消化好了呢?不需要把细胞全部消化成间隔分布很离散的单个圆形才算消化好了,一般你肉眼观察贴壁细胞层,只要能移动了,多半呈沙状移动,其实已经是可以了。一般能移动了,说明细胞与培养基质材料的附着已经消失了,细胞之间的附着也已经消失了,细胞已经独立分布了(虽然没有呈现很广的离散分布)。这个时候应该停止消化,不要等到看到镜下所有细胞都分离得非常好,间隙很大,才停止。细胞系在贴壁的过程中仍然会聚集,这个是贴壁培养的细胞,尤其是肿瘤细胞的一个特性,你可以尝试,准备100%的单个细胞悬液,贴壁后观察细胞,仍然是几个几个细胞聚集在一起。一些悬浮培养细胞也是如此,容易聚集,不要过几个小时就拿出来吹打成单细胞悬液。细胞只要能从基质上脱离下来,即使是成片的(比如Calu-3细胞),吹打不超过20次(一般10次即可),成小规模聚集(10个细胞左右)是正常的,不要再去延长消化时间,等待单细胞悬液出现。比较难消化的细胞:润洗方法5min还不能消化,以结肠癌细胞为例,比如:HCT15、LS411和KM12细胞,胰酶消化,一般10 cm培养皿,一次加入300ul-500ul就足够了。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。一些正常细胞也会有难消化的时候,比如tsDC细胞,用胰酶孵育,3min左右即可看到成片沙状移动。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
HCC1143人乳腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
SU-DHL-10 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:BC-020细胞、Rat-2细胞、NCIH2170细胞
HPASMC Cells;背景说明:肺动脉平滑肌 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW780细胞、Hs-600-T细胞、PAMC82细胞
TALL-104 Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Mino细胞、LTEP-sm 1细胞、HEK-293-EBNA细胞
UPCI-SCC-90 Cells;背景说明:舌鳞癌细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OCI AML4细胞、SU-DHL-10细胞、HCCC-9810细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞复苏相关注意事项:1.取细胞的过程中注意带HAO防冻手套,护目镜。此项尤为重要,细胞冻存管可能漏入,解冻时冻存管中的气温急剧上升,可导致爆炸。2.冻存的问题:冻存的配置已是常识,在这里不作详述,但二甲基亚砜(DMSO )对细胞不是完全无毒副作用,在常温下,二甲基亚砜对细胞的毒副作较大,因此,必须在1-2min内使冻存完全融化。如果复苏温度太慢,会造成细胞的损伤,二甲基亚砜(DMSO)ZuiHAO选择进口产品。3.离心前须加入少量培养。细胞解冻后二甲基亚砜浓度较GAO,注意加入少量培养可稀释其浓度,以减少对细胞的损伤。4.离心问题:目前主要有两种见解。一种是解冻后的细胞悬直接吹打均匀后分装到培养瓶中进行培养,第二天换。因为离心的目的是两个,去除DMSO,去除死细胞,这个是标准流程,但对一般人来说,把握不HAO离心转速和时间,转的不够活细胞沉底的少,细胞就全被扔掉了,转过了活细胞会受压过大,死亡。此外在操作过程中容易污染,所以不推荐。另一种说法为细胞悬中含有二甲基亚砜(DMSO),DMSO对细胞有一定的毒副作用,所以须将离心后的体前倒净,且一定倒干净。我在试验中按照常规的离心分装的方法进行复苏,结果无异常。5.细胞贴壁少的问题:教科书中说明冻存细胞解冻时1ml细胞要加10ml-15ml培养,而在我的试验中的经验总结为培养基越少细胞越容易贴附。6.复苏细胞分装的问题:试验中我的经验总结为复苏1管细胞一般可分装到1-2只培养瓶中,分装过多,细胞浓度过低,不利于细胞的贴壁。7.加培养基的量放入问题:这个量的多少的把握主要涉及到的问题DMSO的浓度,从如果你加培养基的太少,那么DMSO的浓度就会比较大,就会影响细胞生长,从以前的资料来看,DMSO的浓度在小于0.5%的时候对一般细胞没有什么影响,还有一个说法是1%。所以如果你的冻存的浓度是10%DMSO的话那么加10ml以上的培养基就恰HAO稀释到了无害浓度。
C-Li-7 Cells;背景说明:人肝癌细胞株。这株细胞从裸鼠体外移植瘤中建立。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:SW-780细胞、SJSA1细胞、KU 812F细胞
WM-451Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-453细胞、H-1666细胞、LN-18细胞
TOV112D Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MKN7细胞、EB1细胞、HIC细胞
3T3 J2 Cells;背景说明:胚胎;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Henrietta Lacks cells细胞、RPMI 8402细胞、G-401细胞
MV522 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PANC-28细胞、MCF-10A细胞、JROECL 33细胞
LS-180 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO678细胞、ABC1细胞、K1735细胞
MBMEC Cells;背景说明:脑微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CNLMG-B5537SKIN细胞、alphaTC1 Clone 6细胞、UPCI:SCC90细胞
HSF Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:C3H10T1/2 clone8细胞、MGECs细胞、NCIH2106细胞
NE1-E6E7 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SNU668细胞、H-1105细胞、MOLT 4细胞
HTori-3 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LM6细胞、Hs 746T细胞、MUG-Chor1细胞
NCI-H220 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HEC-1A细胞、WM 239细胞、MUTZ1细胞
P36 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:MGSMC细胞、NCIH2227细胞、NHDF细胞
SW-626 Cells;背景说明:卵巢癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Malme-3 M细胞、TALL 1细胞、SK-N-BE(2)细胞
MDA-PCa-2b Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:H-2110细胞、HT144mel细胞、MLMEC细胞
JB6 Cl 30-7b Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:T-ALL-1细胞、Jurkat-77细胞、NCI-H2030细胞
UMC-11 Cells;背景说明:肺肿瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Human Embryo Kidney-2细胞、TSCCa细胞、SNU-620细胞
GNM Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW579细胞、Stanford University Pediatric T-cell line 1细胞、SK.MEL.5细胞
4D20.8 Cells(提供STR鉴定图谱)
Abcam K-562 ACVR2B KO Cells(提供STR鉴定图谱)
AMOC-2 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRK233 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XL920 Cells(提供STR鉴定图谱)
CAL-12T KRAS (G12C/+) Cells(提供STR鉴定图谱)
DA00577 Cells(提供STR鉴定图谱)
DA05387 Cells(提供STR鉴定图谱)
GeneBLAzer ADRA1A-NFAT-bla CHO-K1 Cells(提供STR鉴定图谱)
CESS Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:EFM-19细胞、B16 melanoma细胞、CaSki细胞
HCC1143人乳腺癌细胞代次低|培养基|送STR图谱
HuT 102 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:圆形;淋巴母细胞样;相关产品有:CAKI 1细胞、GM15452细胞、SW954细胞
SUPT-1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:RIN14B细胞、Hs852T细胞、Cates 1B细胞
H-378 Cells;背景说明:小细胞肺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW839细胞、NCI-H87细胞、Alpha Mouse Liver 12细胞
Farage Original Line Cells;背景说明:Farage细胞源于一名患有弥漫性大B细胞淋巴瘤(DLBCL)白人女性的活检淋巴组织,由HBen-Bassat建系。经IL-4处理,该细胞CD21,CD22,CD54和CD58表达上调,而CD21,CD22,andCD38表达下调。;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:IOSE-Van细胞、HO1N1细胞、HEL 299细胞
L cells Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HMC3细胞、M619细胞、MKN 28细胞
1C7 [Mouse hybridoma against M.honghuensis polar filament] Cells(提供STR鉴定图谱)
HPB-ALL Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Eca 109细胞、H3396细胞、PCI-4.2细胞
LLC-MK-2 Cells;背景说明:胚胎;肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A-9细胞、SN12CPM6细胞、NCI-H1734细胞
Hs-606-T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代,2—3天换液一次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:SKES1细胞、NCIH1838细胞、LP-1细胞
B 95.8 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NS20Y细胞、KBM5细胞、HFE-145细胞
RT4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:多角 ;相关产品有:U-118-MG细胞、OLN 93细胞、SVOG细胞
S91 Cells;背景说明:黑色素瘤;雄性;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:EFM192细胞、Walker-256细胞、MDAMB134细胞
MEC1 Cells;背景说明:粘液表皮样癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:J 111细胞、Rat Chondrosarcoma Swarm细胞、SK Col 1细胞
T-47-D Cells;背景说明:浸润性导管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BTI-TN5B1-4细胞、Panc_08_13细胞、KMST6细胞
GM20034 Cells(提供STR鉴定图谱)
HAP1 IRF8 (-) Cells(提供STR鉴定图谱)
ARO 81-1 Cells;背景说明:甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FHCRC-11细胞、MV-3细胞、CNE-2Z细胞
CW2 Cells;背景说明:来源于结肠癌。 CEA阳性,移植于裸鼠可成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Epstein-Barr-2细胞、MJ细胞、HS 683T细胞
HEC1A Cells;背景说明:这株细胞及其亚株HEC-1-B是H.Kuramoto及其同事1968年从一位IA期子宫内膜癌患者身上分离得到的。PAF可以诱导其c-fos的表达。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCIH2029细胞、GES1细胞、Bac12F5细胞
LU65 Cells;背景说明:详见相关文献介绍;传代方法:1:10传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:130 T细胞、H2405细胞、SW 1573细胞
CD18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:LUDLU 1细胞、MARC-145细胞、HCC0827细胞
NCIH1648 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NRK-49F细胞、Hs343T细胞、NS-1细胞
MHCC 97-H Cells;背景说明:来源于中山医院,用人肝癌细胞株MHCC97-H接种裸鼠,进行3次肺转移筛选,取肺转移瘤建成皮下接种后高度自发性肺转移的肝癌细胞系;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NIT-1细胞、SW-626细胞、16HBEo-细胞
HL-7702 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ly3细胞、H508细胞、BPH1细胞
HPS1074 Cells(提供STR鉴定图谱)
JPKO Cells(提供STR鉴定图谱)
MDCC-MSB1 Cells(提供STR鉴定图谱)
ND42223 Cells(提供STR鉴定图谱)
Psi-2 Cells(提供STR鉴定图谱)
Ubigene BEAS-2B SOCS1 KO Cells(提供STR鉴定图谱)
VAMPIRO CHO-K1 Cells(提供STR鉴定图谱)
HD00n Cells(提供STR鉴定图谱)
RBL.2H3 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:BT20细胞、HCC 70细胞、GM346细胞
HEC-251 Cells;背景说明:子宫内膜癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM17346细胞、SV40-MES13细胞、MDA-MB361细胞
SW-1116 Cells;背景说明:CSAp阴性(CSAp-)。 结肠抗原3,阴性。 角蛋白免疫过氧化物酶染色阳性。 癌基因c-myc, K-ras, H-ras, myb, sis 和fos的表达呈阳性。 未检测到癌基因N-myc和N-ras的表达。 表达肿瘤特异的核基质蛋白CC-4,CC-5和CC-6。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CL1细胞、Transformed Human Liver Epithelial-3细胞、MODE-K细胞
hUC-MSC Cells;背景说明:脐带;间充质干 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH378细胞、CHO-ori细胞、MXI细胞
3T3F442A Cells;背景说明:脂肪前体细胞;雄性;Swiss albino;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Microbiological Associates-104细胞、JB-6 Cl 30细胞、NWA细胞
3T3F442A Cells;背景说明:脂肪前体细胞;雄性;Swiss albino;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Microbiological Associates-104细胞、JB-6 Cl 30细胞、NWA细胞
BAC1.2F5 Cells;背景说明:巨噬细胞;SV40转化;BALB/c x A.CA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LAD-2细胞、HBVP细胞、VB细胞
TF-1 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在3×104-5×105 cells/ml;2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:Chang liver细胞、Alexander cells细胞、hTERT RPE1细胞
CCRF CEM Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:ARO81-1细胞、MKN 7细胞、2PK-3细胞
RPE-1 Cells;背景说明:视网膜色素上皮;hTERT永生;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Human Hepatocyte Line 5细胞、H-1793细胞、HFLS细胞
3LL Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HT-22细胞、PA-TU S细胞、SH-SY5Y Parental细胞
3D4/21 Cells;背景说明:肺泡巨噬细胞;SV40转化;Landrace;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:32D CL3细胞、MNNG/HOS Cl #5细胞、TFH细胞
U20-S Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MCF-12F细胞、OPM-2细胞、HuT-102细胞
NCI-SNU-761 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:293细胞、KM12细胞、SNU-182细胞
PL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CCRF-CEM细胞、UACC-893细胞、NTera2 cl.D1细胞
SN12K1 Cells(提供STR鉴定图谱)
QGP 1 Cells;背景说明:胰腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-35细胞、LTEPsm细胞、CATH.a细胞
HOS(TE85) Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HAVSMC细胞、NCI-841细胞、NCIH2347细胞
SW 1116 Cells;背景说明:CSAp阴性(CSAp-)。 结肠抗原3,阴性。 角蛋白免疫过氧化物酶染色阳性。 癌基因c-myc, K-ras, H-ras, myb, sis 和fos的表达呈阳性。 未检测到癌基因N-myc和N-ras的表达。 表达肿瘤特异的核基质蛋白CC-4,CC-5和CC-6。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:WM239A细胞、PF382细胞、MCF-12A细胞
BGC-823 Cells;背景说明:建自一位62岁的胃癌患者;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Pa17C细胞、7404细胞、H-2106细胞
GC-1spg Cells;背景说明:精原细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK.MEL.2细胞、NCI-H1299细胞、YES 2细胞
Calu 6 Cells;背景说明:详见相关文献介绍;传代方法:消化20分钟。1:2。5-6天长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:LAN6细胞、H220细胞、K299细胞
HCC1143人乳腺癌细胞代次低|培养基|送STR图谱
HEK 293-F Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;悬浮生长;形态特性:上皮细胞样;相关产品有:TR 146细胞、Chang liver细胞、RPMI No. 1846细胞
639-V Cells;背景说明:膀胱癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PA-TU-8988S细胞、CAL27细胞、NCI-H1435细胞
COR-L105 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HSC1细胞、NCIH1435细胞、Tca-8113细胞
NCI-H2141 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:KTA-7细胞、Panc2_03细胞、Stanford University-Diffuse Histiocytic Lymphoma-16细胞
MZ-CRC-1 Cells;背景说明:甲状腺髓样癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2030细胞、M-O7e细胞、HO8910细胞
SupB15W Cells;背景说明:详见相关文献介绍;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SNU387细胞、MOPC细胞、PG [Human lung carcinoma]细胞
Hmy.2 CIR Cells;背景说明:B细胞;EBV转染;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:6T-CEM细胞、no-11细胞、SVOG细胞
RPMI1846 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NG-108-15细胞、HIBEC细胞、RINm-5F细胞
BayGenomics ES cell line CSG033 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRT390 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC442 Cells(提供STR鉴定图谱)
JE-2 Cells(提供STR鉴定图谱)
PCRP-USP7-1B6 Cells(提供STR鉴定图谱)
M [Rat] Cells(提供STR鉴定图谱)
" "PubMed=16959974; DOI=10.1126/science.1133427
Sjoblom T., Jones S., Wood L.D., Parsons D.W., Lin J., Barber T.D., Mandelker D., Leary R.J., Ptak J., Silliman N., Szabo S., Buckhaults P., Farrell C., Meeh P., Markowitz S.D., Willis J.E., Dawson D., Willson J.K.V., Gazdar A.F., Hartigan J., Wu L., Liu C.-S., Parmigiani G., Park B.H., Bachman K.E., Papadopoulos N., Vogelstein B., Kinzler K.W., Velculescu V.E.
The consensus coding sequences of human breast and colorectal cancers.
Science 314:268-274(2006)
PubMed=17157791; DOI=10.1016/j.ccr.2006.10.008; PMCID=PMC2730521
Neve R.M., Chin K., Fridlyand J., Yeh J., Baehner F.L., Fevr T., Clark L., Bayani N., Coppe J.-P., Tong F., Speed T., Spellman P.T., DeVries S., Lapuk A., Wang N.J., Kuo W.-L., Stilwell J.L., Pinkel D., Albertson D.G., Waldman F.M., McCormick F., Dickson R.B., Johnson M.D., Lippman M.E., Ethier S.P., Gazdar A.F., Gray J.W.
A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes.
Cancer Cell 10:515-527(2006)
PubMed=17932254; DOI=10.1126/science.1145720
Wood L.D., Parsons D.W., Jones S., Lin J., Sjoblom T., Leary R.J., Shen D., Boca S.M., Barber T.D., Ptak J., Silliman N., Szabo S., Dezso Z., Ustyanksky V., Nikolskaya T., Nikolsky Y., Karchin R., Wilson P.A., Kaminker J.S., Zhang Z.-M., Croshaw R., Willis J.E., Dawson D., Shipitsin M., Willson J.K.V., Sukumar S., Polyak K., Park B.H., Pethiyagoda C.L., Pant P.V.K., Ballinger D.G., Sparks A.B., Hartigan J., Smith D.R., Suh E., Papadopoulos N., Buckhaults P., Markowitz S.D., Parmigiani G., Kinzler K.W., Velculescu V.E., Vogelstein B.
The genomic landscapes of human breast and colorectal cancers.
Science 318:1108-1113(2007)
PubMed=19582160; DOI=10.1371/journal.pone.0006146; PMCID=PMC2702084
Kao J., Salari K., Bocanegra M., Choi Y.-L., Girard L., Gandhi J., Kwei K.A., Hernandez-Boussard T., Wang P., Gazdar A.F., Minna J.D., Pollack J.R.
Molecular profiling of breast cancer cell lines defines relevant tumor models and provides a resource for cancer gene discovery.
PLoS ONE 4:E6146-E6146(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=21778573; DOI=10.3233/BD-2010-0307; PMCID=PMC3532890
Chavez K.J., Garimella S.V., Lipkowitz S.
Triple negative breast cancer cell lines: one tool in the search for better treatment of triple negative breast cancer.
Breast Dis. 32:35-48(2010)
DOI=10.4172/2157-7145.S2-005
Fang R.-X., Shewale J.G., Nguyen V.T., Cardoso H., Swerdel M.R., Hart R.P., Furtado M.R.
STR profiling of human cell lines: challenges and possible solutions to the growing problem.
J. Forensic Res. 2 Suppl. 2:5-5(2011)
PubMed=22414580; DOI=10.1158/0008-5472.CAN-11-3711
Geiger T., Madden S.F., Gallagher W.M., Cox J., Mann M.
Proteomic portrait of human breast cancer progression identifies novel prognostic markers.
Cancer Res. 72:2428-2439(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
PubMed=23151021; DOI=10.1186/1471-2164-13-619; PMCID=PMC3546428
Grigoriadis A., Mackay A., Noel E., Wu P.-J., Natrajan R., Frankum J., Reis-Filho J.S., Tutt A.
Molecular characterisation of cell line models for triple-negative breast cancers.
BMC Genomics 13:619.1-619.14(2012)
PubMed=23601657; DOI=10.1186/bcr3415; PMCID=PMC3672661
Riaz M., van Jaarsveld M.T.M., Hollestelle A., Prager-van der Smissen W.J.C., Heine A.A.J., Boersma A.W.M., Liu J.-J., Helmijr J.C.A., Ozturk B., Smid M., Wiemer E.A.C., Foekens J.A., Martens J.W.M.
miRNA expression profiling of 51 human breast cancer cell lines reveals subtype and driver mutation-specific miRNAs.
Breast Cancer Res. 15:R33.1-R33.17(2013)
PubMed=24094812; DOI=10.1016/j.ccr.2013.08.020; PMCID=PMC3931310
Timmerman L.A., Holton T., Yuneva M., Louie R.J., Padro M., Daemen A., Hu M., Chan D.A., Ethier S.P., van 't Veer L.J., Polyak K., McCormick F., Gray J.W.
Glutamine sensitivity analysis identifies the xCT antiporter as a common triple-negative breast tumor therapeutic target.
Cancer Cell 24:450-465(2013)
PubMed=24162158; DOI=10.1007/s10549-013-2743-3; PMCID=PMC3832776
Prat A., Karginova O., Parker J.S., Fan C., He X.-P., Bixby L.M., Harrell J.C., Roman E., Adamo B., Troester M.A., Perou C.M.
Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes.
Breast Cancer Res. Treat. 142:237-255(2013)
PubMed=24176112; DOI=10.1186/gb-2013-14-10-r110; PMCID=PMC3937590
Daemen A., Griffith O.L., Heiser L.M., Wang N.J., Enache O.M., Sanborn Z., Pepin F., Durinck S., Korkola J.E., Griffith M., Hur J.S., Huh N., Chung J., Cope L., Fackler M.J., Umbricht C.B., Sukumar S., Seth P., Sume V.P., Jakkula L.R., Lu Y.-L., Mills G.B., Cho R.J., Collisson E.A., van 't Veer L.J., Spellman P.T., Gray J.W.
Modeling precision treatment of breast cancer.
Genome Biol. 14:R110.1-R110.14(2013)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25576301; DOI=10.1074/mcp.M114.042812; PMCID=PMC4349985
Bassani-Sternberg M., Pletscher-Frankild S., Jensen L.J., Mann M.
Mass spectrometry of human leukocyte antigen class I peptidomes reveals strong effects of protein abundance and turnover on antigen presentation.
Mol. Cell. Proteomics 14:658-673(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25892236; DOI=10.1016/j.celrep.2015.03.050; PMCID=PMC4425736
Lawrence R.T., Perez E.M., Hernandez D., Miller C.P., Haas K.M., Irie H.Y., Lee S.-I., Blau C.A., Villen J.
The proteomic landscape of triple-negative breast cancer.
Cell Rep. 11:630-644(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28287265; DOI=10.1021/acs.jproteome.6b00470; PMCID=PMC5557415
Yen T.-Y., Bowen S., Yen R., Piryatinska A., Macher B.A., Timpe L.C.
Glycoproteins in claudin-low breast cancer cell lines have a unique expression profile.
J. Proteome Res. 16:1391-1400(2017)
PubMed=28889351; DOI=10.1007/s10549-017-4496-x
Saunus J.M., Smart C.E., Kutasovic J.R., Johnston R.L., Kalita-de Croft P., Miranda M., Rozali E.N., Vargas A.C., Reid L.E., Lorsy E., Cocciardi S., Seidens T., McCart Reed A.E., Dalley A.J., Wockner L.F., Johnson J., Sarkar D., Askarian-Amiri M.E., Simpson P.T., Khanna K.K., Chenevix-Trench G., Al-Ejeh F., Lakhani S.R.
Multidimensional phenotyping of breast cancer cell lines to guide preclinical research.
Breast Cancer Res. Treat. 167:289-301(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"