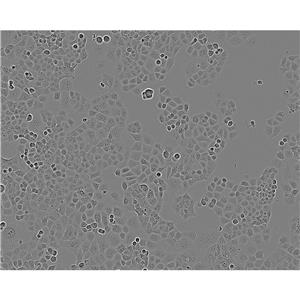
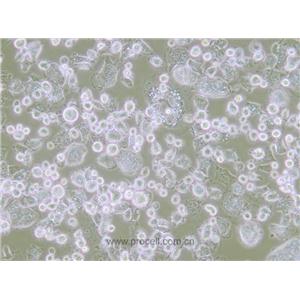
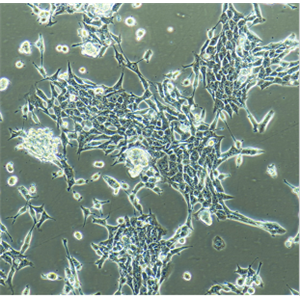
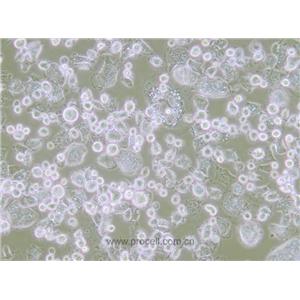
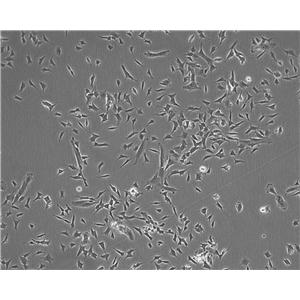
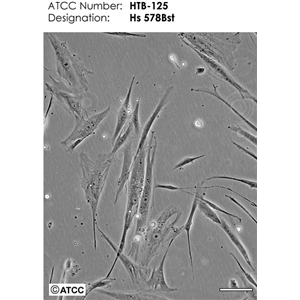
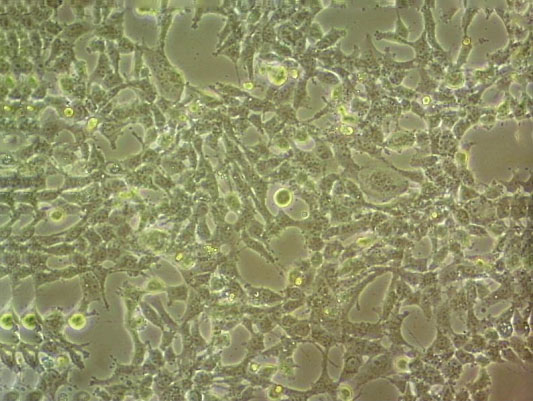
"KYSE-510人食管癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
换液周期:每周2-3次
SHSY-5Y Cells;背景说明:据报道,该细胞的密度可高达1×106cells/cm2,具有中等水平的多巴胺β羟化酶的活性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HEL-1细胞、SKLMS-1细胞、Human Corneal Epithelial cells-Transformed细胞
MDA-MB-415 Cells;背景说明:这株细胞表达WNT7B癌基因。8168088].带瘤患者来自巴拉圭,虽然填报的是白人,但细胞表型存在G6PDA型,显示其属于混血。细胞株形成平展延伸的上皮细胞样,在电镜下呈现结节,伴随着延伸的微管和微板。不容易用胰酶消化。;传代方法:消化5-10分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCI.H23细胞、QSG7701细胞、H-1930细胞
GM2131 Cells;背景说明:B淋巴细胞;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RT-112细胞、P3-X63-Ag8-6-5-3细胞、PIEC细胞
KYSE-510人食管癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
ATCC细胞库(American Type Culture Colection),该中心一直致力于细胞分类、鉴定和保藏工作。ATCC是全球最大的生物资源保藏中心,ATCC通过行业标准产品、服务和创新解决方案支持全球学术、政府、生物技术、制药、食品、农业和工业领域的科学进步。ATCC提供的服务和定制解决方案包括细胞和微生物培养、鉴定、生物衍生物的开发和生产、性能测试和生物资源保藏服务。美国国家标准协会(ANSI)认可了ATCC标准开发组织,并制定了标准协议,以确保生物材料的可靠性和可重复性。ATCC的使命是为了获取、鉴定、保存、开发、标准化和分发生物资源和生物信息,以提高和应用生物科学知识。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
KYSE-510人食管癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
NK 10a Cells;背景说明:1967年,该细胞系KleinE和KleinG建系,源于一名16岁患有Burkitt's淋巴瘤的黑人男性,beta-2-微球蛋白阴性,表达EBNA,VCA,sIg。该细胞携带EB病毒,是一个典型的B淋巴母细胞系,可用于白血病发病机制的研究。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCIH1734细胞、alpha TC1.6细胞、MPP-89细胞
DMS 114 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MDAMB175VII细胞、BrCL12细胞、Roswell Park Memorial Institute 1846细胞
MUTZ3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Vero-76细胞、HCC202细胞、HUCCT1细胞
Bovine ENDometrial cells Cells;背景说明:子宫内膜;上皮细胞;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:N9细胞、MDA-MB-134-VI细胞、CF Pac1细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞传代培养实验:体外培养的原代细胞或细胞株要在体外持续地培养就必须传代,以便获得稳定的细胞株或得到大量的同种细胞,并维持细胞种的延续。培养的细胞形成单层汇合以后,由于密度过大生存空间不足而引起营养枯竭,将培养的细胞分散,从容器中取出,以1:2或1:3以上的比率转移到另外的容器中进行培养,即为传代培养;细胞“一代”指从细胞接种到分离再培养的一段期间,与细胞世代或倍增不同。在一代中,细胞培增3~6次。细胞传代后,一般经过三个阶段:游离期、指数增生期和停止期。常用细胞分裂指数表示细胞增殖的旺盛程度,即细胞群的分裂相数/100个细胞。一般细胞分裂指数介于0.2%~0.5%,肿瘤细胞可达3~5%;细胞接种2~3天分裂增殖旺盛,是活力ZuiHAO时期,称指数增生期(对数生长期),适宜进行各种试验。实验步骤:1.将长成的培养细胞从二氧化碳培养箱中取出,在超净工作台中倒掉瓶内的培养,加入少许消化。(以面盖住细胞为宜),静置5~10分钟。2.在倒置镜下观察被消化的细胞,如果细胞变圆,相互之间不再连接成片,这时应立即在超净台中将消化倒掉,加入3~5ml新鲜培养,吹打,制成细胞悬。3.将细胞悬吸出2ml左右,加到另一个培养瓶中并向每个瓶中分别加3ml左右培养,盖HAO瓶塞,送回二氧化碳培养箱中,继续进行培养。一般情况,传代后的细胞在2小时左右就能附着在培养瓶壁上,2~4天就可在瓶内形成单层,需要再次进行传代。
PCI-4B Cells;背景说明:喉鳞癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GDM-1细胞、NUGC4细胞、HUT125细胞
BT474 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-727细胞、McA-RH8994细胞、MS-751细胞
CCD1095Sk Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H711细胞、ETCC-007细胞、MM134细胞
GM-3573 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:CCD18细胞、COLO-394细胞、CCRF-CEM C7细胞
Ly1 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Intestinal Porcine Epithelial Cell line-1细胞、Sp2/0细胞、NCIH847细胞
HCC1500 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每3-5天换液;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OEC33细胞、HTR-8/SV neo细胞、H1417细胞
BMDC Cells;背景说明:树突状 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KGN细胞、AHH-1细胞、MDA MB 231细胞
Rat-1 Cells;背景说明:成纤维细胞;自发永生;Fischer 344;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM00215A细胞、MDA231细胞、KU-812-F细胞
P-815 Cells;背景说明:肥大细胞瘤;雄性;DBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:P3-X63-Ag8细胞、Human fetal lung fibroblast 1细胞、Panc-327细胞
F98 Cells;背景说明:恶性胶质瘤;雌性;Fischer大鼠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MC-CAR细胞、YES 2细胞、YAC-1细胞
Pro-Lec1.3C Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:3T3 clone A31细胞、UACC 812细胞、NCI-H1770细胞
P30/Ohkubo Cells;背景说明:详见相关文献介绍;传代方法:10^5 cells/60mm dish;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:OVCAR 433细胞、SUM 149细胞、CEM T4细胞
293-F Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;悬浮生长;形态特性:上皮细胞样;相关产品有:PC3M-IE8细胞、JIII细胞、C8161细胞
SCC90 Cells;背景说明:舌鳞癌细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO.205细胞、U-373MG ATCC细胞、W256细胞
H69/P Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:悬浮生长,聚团;形态特性:聚团悬浮;相关产品有:Mo 59J细胞、SNU-1040细胞、HFT-8810细胞
EBTr (NBL-4) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hep3B细胞、MD Anderson-Metastatic Breast-134-VI细胞、HHL5细胞
MHH-CALL-2 Cells;背景说明:急性B淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HIEC细胞、KYSE 270细胞、OCI/AML-3细胞
Abcam A-549 NEU1 KO Cells(提供STR鉴定图谱)
Abgenix 4.288.1 Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSG443 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRU357 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC799 Cells(提供STR鉴定图谱)
CHO-CYP1A2 C2 Cells(提供STR鉴定图谱)
DA02491 Cells(提供STR鉴定图谱)
DA06484 Cells(提供STR鉴定图谱)
GM01053 Cells(提供STR鉴定图谱)
SW 620 Cells;背景说明:SW620是从一个51岁男性白人组织中分离得到。由A.Leibovitz等从一个淋巴结建株。细胞系主要由无绒毛的小园球细胞和双极细胞组成。它仅合成少量癌胚抗原(CEA)且在裸鼠中有高度的致瘤性;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H-650细胞、SKMEL2细胞、Hs695细胞
KYSE-510人食管癌细胞代次低|培养基|送STR图谱
MUTZ-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H1915细胞、NCIH1734细胞、DV-90细胞
U-251MG Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:SW1116细胞、EC-GI-10细胞、P 3 HR 1细胞
GM04679 Cells;背景说明:该细胞源自一名3岁患有Burkitt's淋巴瘤的黑人男孩的B淋巴细胞,EBNA阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HEK-2细胞、CO115细胞、SK-RC-42细胞
MC3T3, E1 subgroup-4 clone Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ketr-3细胞、HAPI细胞、C-6细胞
HSAS4 Cells;背景说明:皮肤成纤维细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A 2058细胞、C8166细胞、MDA-MB-134VI细胞
311-3D4 Cells(提供STR鉴定图谱)
COLO-HSR Cells;背景说明:该细胞1984年建系,源自一位33岁患有大肠腺癌男性经5-fu治疗后的腹水。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:详见产品说明书;相关产品有:H.Ep. #2细胞、HOC1细胞、rRTEC细胞
MOPC Cells;背景说明:少突胶质前体 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:HS-294细胞、GM3570细胞、C57 Mouse Tumor 64细胞
NCI-H1703 Cells;背景说明:该细胞1987年建系,源自一位54岁患有非小细胞肺癌的白人男性,该患者为吸烟者。;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:LC1sq细胞、NH-6细胞、OVCAR3细胞
MV-4:11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SUM-159-PT细胞、FHs74 Int细胞、H-1755细胞
NCIH322 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:SW-1116细胞、H-865细胞、Henrietta Lacks cells细胞
CT26.WT Cells;背景说明:CT26细胞是被N-亚硝基-N-甲基脲烷(NNMU)诱导得到的未分化的小鼠结肠癌细胞,该细胞的一个克隆形成的细胞系被命名为CT26.WT。CT26.WT被逆转录病毒载体LXSN稳定转化形成了一个致死性的亚克隆CT26.CL25,这一病毒载体含有lacZ基因、编码肿瘤相关抗原(TAA)和beta半乳糖苷酶。CT26.WT和CT26.CL25细胞在小鼠中生长速度和致死率都很相似,不同的是CT26.CL25细胞可以表达肿瘤相关抗原和beta半乳糖苷酶,因此这两株细胞可以联合用于免疫治疗和宿主免疫反应的研究。 ;传代方法:1:4-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NCIH1341细胞、SCC-25细胞、SN-12C细胞
BaF3 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代;3-4天1次。;生长特性:悬浮生长;形态特性:圆形;相关产品有:ECV304细胞、NCIH526细胞、H-1437细胞
JOSK-M Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SKNO-1细胞、Colo699细胞、HcaF细胞
GM20644 Cells(提供STR鉴定图谱)
HAP1 KAT2B (-) 2 Cells(提供STR鉴定图谱)
RERF-LC-MS Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NH6细胞、H-211细胞、RBL-I细胞
KLM-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RPE1细胞、KALS1细胞、MEL细胞
NCI-H322T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Rh30细胞、H-660细胞、BC-PAP细胞
LK 2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MZ-CRC-1细胞、LADMAC细胞、J774 A1细胞
KYSE520 Cells;背景说明:食管鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TE-85细胞、Lewis lung carcinoma细胞、LC-2 ad细胞
LLCMK2 Cells;背景说明:胚胎;肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L615细胞、Hs888Lu细胞、Hs_578t细胞
Daoy Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:多边形;相关产品有:KMB-17细胞、UMRC-2细胞、Rat-2细胞
SKNDZ Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:751-NA-15细胞、HCC366细胞、PFSK-1细胞
HPS1703 Cells(提供STR鉴定图谱)
JUCGRMi002-B Cells(提供STR鉴定图谱)
MDCK-dEGF_2-EKARrEV-NLS Cells(提供STR鉴定图谱)
ND50093 Cells(提供STR鉴定图谱)
Pso-5 Cells(提供STR鉴定图谱)
UCF0094 Cells(提供STR鉴定图谱)
ZK Cells(提供STR鉴定图谱)
HAP1 TNPO1 (-) 2 Cells(提供STR鉴定图谱)
NCI-H1838 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:A3细胞、Strain V细胞、B16细胞
Hut292 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:H650细胞、GM3569细胞、H-841细胞
NCIH378 Cells;背景说明:小细胞肺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:COLO320-DM细胞、COS-7细胞、EC9706细胞
Karpas422 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CAL 12细胞、Hepa 1-6细胞、W133细胞
Granta-519 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RMS13细胞、RPMI 8226/S细胞、SUM 102PT细胞
Granta-519 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RMS13细胞、RPMI 8226/S细胞、SUM 102PT细胞
NB19-RIKEN Cells;背景说明:神经母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MODE-K细胞、NCI-H2342细胞、IPEC-J2细胞
BPH1 Cells;背景说明:良性前列腺增生;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U118MG细胞、MDA-MB-435-S细胞、Lewis lung carcinoma line 1细胞
NCIH82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:HT55细胞、NTera 2细胞、Stanford University-Diffuse Histiocytic Lymphoma-1细胞
FTC133 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HCC1395细胞、HSAS1细胞、HCA 7细胞
GP2-293 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AM38细胞、COLO320-DM细胞、NCI-H1944细胞
H 9 Cells;背景说明:H9细胞是HUT78(ATCCTIB161)的克隆系(Callo,RC,etal)。细胞表面带有CD3、CD4标记。研究表明,该细胞系对人体免疫缺陷病毒(HIV-1)敏感,可用于检测、分离和增殖HIV-1,也可用于其它人类Tcell病毒的研究。;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Tadarida brasiliensis 1 lung细胞、BE(2)C细胞、P3/NS1/1-Ag4.1细胞
Calf Pulmonary Artery Endothelial Cells;背景说明:肺血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GTL16细胞、T47-D细胞、MDA-468细胞
KM12SM Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OV90细胞、16-HBEo细胞、SW 948细胞
WERI-Rb-1 Cells;背景说明:WERI-Rb-I细胞株是1974年R.M. McFall 和 T.W. Sery建立的两株人眼癌细胞系中的一株。 细胞能在Difco Bacto-Agar中存活但不形成克隆。 扫描电镜显示在表面囊泡,板状伪足和微绒毛在数量上和频率上的改变。 细胞分化研究,肿瘤治疗的动物模型和生化评价都涉及这株细胞。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:圆形细胞聚集成葡萄状;相关产品有:HDLM-2细胞、RT112细胞、UM-UC-1细胞
SNU-2727 Cells(提供STR鉴定图谱)
H-2106 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:Japanese Tissue Culture-39细胞、SBC-5细胞、C4 I细胞
RD Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:MDA MB231细胞、Hs281T细胞、KYSE0410细胞
COR-L51 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:C26细胞、MC/9细胞、LM8细胞
CHP 126 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-23细胞、OVCAR.3细胞、Cates-1B细胞
DoHH2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:VMRCRCZ细胞、KYSE 180细胞、H-2291细胞
SNB-19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:LP1细胞、CCD18细胞、SKOV3细胞
KYSE-510人食管癌细胞代次低|培养基|送STR图谱
NCI-H510 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;每周换液2次。;生长特性:混合生长;形态特性:上皮细胞样;相关产品有:NCI-H1755细胞、BE(2)M17细胞、BI-Mel细胞
LICR-HN-6 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MB468细胞、38C-13细胞、Roswell Park Memorial Institute 6666细胞
Hi5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:JC细胞、SKCO1细胞、CEMx721.174.T2细胞
DHL-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KS-1 [Human glioblastoma]细胞、OV 1063细胞、FL-83B细胞
B-95-8 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:L-cell细胞、PK15细胞、SR细胞
HBL 100 Cells;背景说明:该细胞由E.V.Gaffney及其同事从一位没有乳癌家族史的供者乳汁中建立,培养出来的细胞染色体组型在第7代时就不正常;电镜照片显示有微丝、张力原纤维和桥粒;Southern转移表明有整合型SV40病毒基因,当作正常细胞。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:RKOE6细胞、SUNE-1细胞、H-1417细胞
SUDHL2 Cells;背景说明:弥漫性大细胞淋巴瘤;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:GM01232细胞、HcaF细胞、C643细胞
PFSK Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:L-Wnt3A细胞、Pt K2细胞、Med 283细胞
BayGenomics ES cell line RRO524 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHC065 Cells(提供STR鉴定图谱)
HCA-57HDR Cells(提供STR鉴定图谱)
PCRP-CBFB-1B2 Cells(提供STR鉴定图谱)
BN-175 Cells(提供STR鉴定图谱)
HPS0253 Cells(提供STR鉴定图谱)
" "PubMed=9033652; DOI=10.1002/(SICI)1097-0215(19970207)70:4<437::AID-IJC11>3.0.CO;2-C
Tanaka H., Shimada Y., Imamura M., Shibagaki I., Ishizaki K.
Multiple types of aberrations in the p16 (INK4a) and the p15(INK4b) genes in 30 esophageal squamous-cell-carcinoma cell lines.
Int. J. Cancer 70:437-442(1997)
PubMed=11092977; DOI=10.1111/j.1349-7006.2000.tb00895.x; PMCID=PMC5926289
Pimkhaokham A., Shimada Y., Fukuda Y., Kurihara N., Imoto I., Yang Z.-Q., Imamura M., Nakamura Y., Amagasa T., Inazawa J.
Nonrandom chromosomal imbalances in esophageal squamous cell carcinoma cell lines: possible involvement of the ATF3 and CENPF genes in the 1q32 amplicon.
Jpn. J. Cancer Res. 91:1126-1133(2000)
PubMed=15172977; DOI=10.1158/0008-5472.CAN-04-0172
Sonoda I., Imoto I., Inoue J., Shibata T., Shimada Y., Chin K., Imamura M., Amagasa T., Gray J.W., Hirohashi S., Inazawa J.
Frequent silencing of low density lipoprotein receptor-related protein 1B (LRP1B) expression by genetic and epigenetic mechanisms in esophageal squamous cell carcinoma.
Cancer Res. 64:3741-3747(2004)
PubMed=16045545; DOI=10.1111/j.0959-9673.2005.00431.x; PMCID=PMC2517430
Ban S., Michikawa Y., Ishikawa K.-i., Sagara M., Watanabe K., Shimada Y., Inazawa J., Imai T.
Radiation sensitivities of 31 human oesophageal squamous cell carcinoma cell lines.
Int. J. Exp. Pathol. 86:231-240(2005)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"