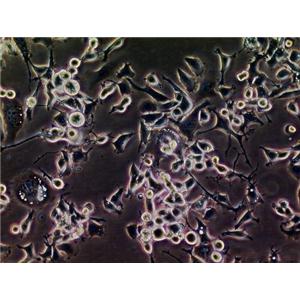
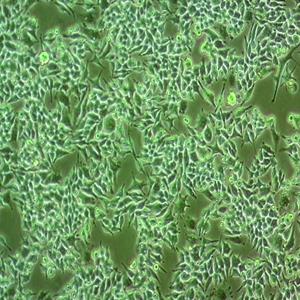
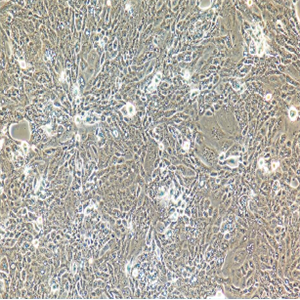
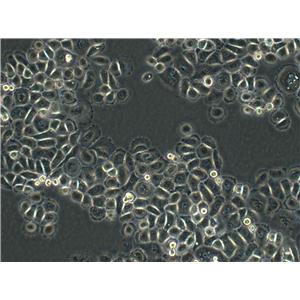
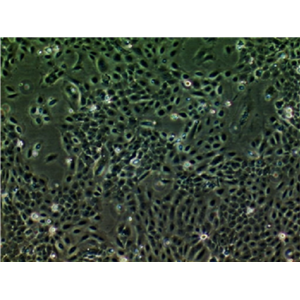
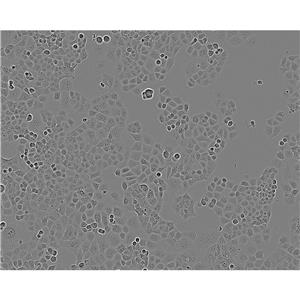
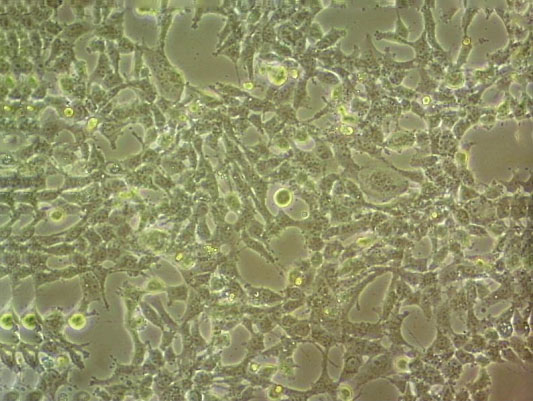
"Capan-1人胰腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
BNCL-2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:CAKI 2细胞、QGY-7701细胞、A72细胞
TSUpr1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SNU761细胞、UCLA NPA871细胞、KG1细胞
HCET Cells;背景说明:角膜上皮细胞;Ad-SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI H295R细胞、FM88细胞、EA.hy 926细胞
Capan-1人胰腺癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
细胞培养应用于分子生物学、细胞生物学、遗传学、免疫学、肿瘤学及病毒学等领域,细胞培养是指将细胞从动物或植物体内取出,然后在适宜的人工环境中生长的过程。细胞可以在培养前直接从组织中取出并通过酶或机械方法进行解离,也可以来源于已建立的细胞系。传代培养是指当细胞生长至高密度时,将其分殖至新的培养瓶中,以维持其生长和增殖。贴壁细胞是指在培养基表面附着生长的细胞,悬浮细胞是指在培养基中悬浮生长的细胞,不依附于培养皿表面。半贴壁半悬浮细胞同时具备贴壁细胞和悬浮细胞的特点,通常在培养基中部分附着生长,部分悬浮于培养基上。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
Capan-1人胰腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
Sci-1 Cells;背景说明:胚胎;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Anip-973细胞、EC-9706细胞、ECC 10细胞
SK-N-FI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Tb 1 Lu细胞、P3/X63/Ag8细胞、MCF 10A细胞
GM0637 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SUM149细胞、TOV112D细胞、Leukemic 1210细胞
SCaBER Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:QGY7703细胞、RGC6细胞、L 428细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
贴壁细胞消化传代时通常采用两种方法:一、加入胰酶等细胞脱落后,再加培养基中止胰酶作用,离心传代;二、加入胰酶后,镜下观察待细胞始脱落时,弃胰酶,加培养分瓶。但前者太麻烦,而后者有可能对细胞施加胰酶选择,因为总是贴壁不牢的细胞先脱落,对肿瘤细胞来说,这部分细胞有可能是恶性程度较GAO的细胞亚群。一种简单的消化传代方法。加入PBS洗去血清或加入胰酶先中和血清的作用(30s),弃之,再加入适量胰酶作用10s-40s(根据细胞消化的难易程度),弃之,这样依赖残余的胰酶就可将细胞消化单细胞。对于较难消化的细胞,可以用2%利多卡因消化5-8分钟,然后再弃去,加培养基吹打也可以,对细胞的影响不大。不用PBS也不用Hanks洗,只要把旧培养吸的干净一点,直接加酶消化应该不会有什么问题。弃培养后,用0.04%的EDA冲洗一次,再用1/4v的0.04%的EDA室温孵育5min,弃取大部分EDA,加入与剩余EDA等量的胰酶(预热)总体积1/10v。消化到有细胞脱落。不过有人说EDA对细胞不HAO,有证据吗?培养的BASMC:倒掉旧培养加入少量胰酶冲一下,倒掉再加入0.125-0.25%胰酶约6-10滴或1ml(25ml bole)消化再加入适量新培养基中和,并分瓶这种方法简单、省事;效果很HAO并且不损失细胞!
ATDC-5 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:KMST-6细胞、OVCAR-3细胞、MBMEC细胞
SK-MEL3 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:D283MED细胞、MDA-MB231细胞、PaTu 8988s细胞
HCS2/8 Cells;背景说明:软骨肉瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SUSM-1细胞、CHP126细胞、H-2227细胞
RD 2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:SACCLM细胞、PANC-10-05细胞、COLO-HSR细胞
H-2291 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SuDHL 4细胞、BCaP-37细胞、HeLa/DDP细胞
L5178-R Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Karpas-299细胞、GM3190细胞、P3X63-Ag8.653细胞
HCoEpiC Cells;背景说明:结肠上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P36细胞、C8D1A细胞、HONE-1细胞
2BS Cells;背景说明:胚肺;成纤维细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:alphaTC1 Clone 6细胞、NCI-H2030细胞、H69细胞
NK 10a Cells;背景说明:1967年,该细胞系KleinE和KleinG建系,源于一名16岁患有Burkitt's淋巴瘤的黑人男性,beta-2-微球蛋白阴性,表达EBNA,VCA,sIg。该细胞携带EB病毒,是一个典型的B淋巴母细胞系,可用于白血病发病机制的研究。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:NCIH1734细胞、alpha TC1.6细胞、MPP-89细胞
MCF-7ADR Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Walker-Ca.256细胞、KASUMI1细胞、EJ 138细胞
H2009 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MCF7细胞、OC-316细胞、HEK293-H细胞
V-79-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HBZY 1细胞、C3H/10T1/2 clone 8细胞、A204细胞
NCIH1650 Cells;背景说明:该细胞是从一名27岁白人男性(10年烟龄)支气管肺泡癌患者的胸腔积液中分离得到的。;传代方法:1:4-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:MUTZ1细胞、Anip973细胞、32Dc3细胞
NK10a Cells;背景说明:1967年,该细胞系KleinE和KleinG建系,源于一名16岁患有Burkitt's淋巴瘤的黑人男性,beta-2-微球蛋白阴性,表达EBNA,VCA,sIg。该细胞携带EB病毒,是一个典型的B淋巴母细胞系,可用于白血病发病机制的研究。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HuTu 80细胞、UMUC14细胞、SN12PM6细胞
Hs 822.T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SK-N-MC细胞、CV 1细胞、GCT0404细胞
EFM-192B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH548细胞、CEK细胞、Hs 578T细胞
MOLT 4 Cells;背景说明:MOLT-4与MOLT-3来源于一名19岁的男性急性淋巴细胞性白血病的复发患者,该患者前期接受过多种药物联合化疗。MOLT-4细胞系为T淋巴细胞起源,p53基因的第248位密码子有一个G→A突变,不表达p53,不表达免疫球蛋白或EB病毒;可产生高水平的末端脱氧核糖转移酶;表达CD1(49%),CD2(35%),CD3A(26%)B(33%)C(34%),CD4(55%),CD5(72%),CD6(22%),CD7(77%)。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:NCI-H676B细胞、NBLAN5T细胞、TMK-1细胞
3Ao/cDDP Cells(提供STR鉴定图谱)
Abcam HeLa ZRANB1 KO Cells(提供STR鉴定图谱)
Akata delta2 EBER1(+) Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRJ374 Cells(提供STR鉴定图谱)
BayGenomics ES cell line Xk343 Cells(提供STR鉴定图谱)
C822B Cells(提供STR鉴定图谱)
DA00344 Cells(提供STR鉴定图谱)
E246 Cells(提供STR鉴定图谱)
GM04603 Cells(提供STR鉴定图谱)
Panc-3_27 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:WiDr细胞、SUIT 2细胞、PC3M-2B4细胞
Capan-1人胰腺癌细胞代次低|培养基|送STR图谱
KYSE70 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TF-1细胞、H-1838细胞、52PE细胞
MH-S Cells;背景说明:巨噬细胞;雄性;SV40转化;BALB/cJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Stanford University-Diffuse Histiocytic Lymphoma-1细胞、PC12(高分化)细胞、KE39细胞
TOV21G Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Ontario Cancer Institute-Acute Myeloid Leukemia-4细胞、PANC-1细胞、SNU398细胞
16HBE140 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NCI-SNU-475细胞、LCLC-103H细胞、BTI-TN5B1-4细胞
AAV-293 Cells;背景说明:我们推荐使用AAV-293细胞株繁殖腺病毒相关重组病毒。 AAV-293源自普遍使用的 HEK293细胞株,但产生的病毒滴度更高。 HEK293细胞是剪切过的腺病毒5型DNA转染的人胚肾细胞。 跟HEK293细胞一样,AAV-293细胞反式表达腺病毒E1基因,当共转染三个AAV助质粒(一个含ITR的质粒,pAAV-RC, 和E1缺失助质粒)时,可以产生有感染力的腺病毒-相关病毒颗粒。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:P19细胞、A1847细胞、HECV细胞
7F9 Cells(提供STR鉴定图谱)
NCI H82 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代,每周换液2-3次;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:SP2/0 Ag-14细胞、SNU668细胞、Clone Y-1细胞
Jurkat Clone E6-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CNE2Z细胞、D-324MED细胞、RIN-m细胞
AML12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MC-3T3-E1细胞、PIG3细胞、Panc-08.13细胞
HN4 Cells;背景说明:喉鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LA4细胞、MDA361细胞、H-1618细胞
HDQP1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Tissue Culture-1细胞、HuP-T4细胞、Ect1/E6E7细胞
Caov-4 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IR983F细胞、Cor L51细胞、SW13细胞
RIN Cl-5F Cells;背景说明:胰岛β细胞瘤;雄性;NEDH;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNB19细胞、HCC9204细胞、GM15452细胞
HOC Cells;背景说明:肝;卵圆 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-2073细胞、293S细胞、H1648细胞
GM20270 Cells(提供STR鉴定图谱)
HAP1 ITGA2B (-) 2 Cells(提供STR鉴定图谱)
COLO680N Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCC-HSF-1细胞、SNU182细胞、LS411N细胞
DMS 273 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:AN-3细胞、OCI-Ly7细胞、Hs940-T细胞
JHH-2 Cells;背景说明:肝癌;腹水转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MRC-9细胞、LP1细胞、IM95细胞
E.L.4 Cells;背景说明:EL4是从用9,10-二甲基-1,2-并蒽在C57BL小鼠中诱导的淋巴瘤中建立的。 能抗0.1 mM 化可的松,对20 mcg/ml PHA敏感。 还有一个亚株(EL4.IL-2, ATCC TIB-181)可以生成高水平的IL-2。 检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:HEL9217细胞、HEL299细胞、SVHUC细胞
STO Cells;背景说明:STO是一株继代生长的胚成纤维细胞系,可用于制备饲养层细胞(feederlayers)和其他研究。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:NCIH1092细胞、WEHI3B细胞、H-146细胞
SK-N-F1 Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs 274.T细胞、IM-95细胞、SW 1271细胞
KPNRTBM1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:32D clone3细胞、H 9细胞、CP-70细胞
SW 900 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H295R-S1细胞、H1563细胞、Tb1.Lu细胞
HPS1204 Cells(提供STR鉴定图谱)
JS2 TLR4-/- D299G/T399I Cells(提供STR鉴定图谱)
MDCC-UD13 Cells(提供STR鉴定图谱)
ND42504 Cells(提供STR鉴定图谱)
Psi-CRIP-MFGmIFN-gamma Cells(提供STR鉴定图谱)
Ubigene HCT 116 HCN4 KO Cells(提供STR鉴定图谱)
W4 [Mouse ESC] Cells(提供STR鉴定图谱)
HCA7 colony 11 Cells(提供STR鉴定图谱)
TE-85 clone 5 Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TE6细胞、MRC-5细胞、FT-293细胞
UMUC-14 Cells;背景说明:肾癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCAEC细胞、NS-20Y细胞、CTLL2细胞
H1734 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H-250细胞、211H细胞、BE2C细胞
WISH Cells;背景说明:最初以为这株细胞的起源是正常羊膜,但随后通过同工酶分析、HeLa标记染色体和DNA指纹法分析,证实该细胞是HeLa细胞污染的;角蛋白阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长 ;形态特性:上皮细胞样;相关产品有:OLN 93细胞、OVCA433细胞、SW-1116细胞
RGCs Cells;背景说明:视网膜;神经节 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-MES-1细胞、C81-61细胞、3T6 Swiss Albino细胞
RGCs Cells;背景说明:视网膜;神经节 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-MES-1细胞、C81-61细胞、3T6 Swiss Albino细胞
PJ34 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:P3HRI细胞、SNU-878细胞、HOP 62细胞
HSC-4 Cells;背景说明:详见相关文献介绍;传代方法:1 x 10^5 cells/10cm dish;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:RPMC细胞、COLO 679细胞、SK-N-BE细胞
Metastatic Variant 522 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ZR-75-30细胞、MV3细胞、MKN-74细胞
SF-126 Cells;背景说明:该细胞来源于星形胶质细胞瘤;胶质纤维酸性蛋白(GFAP)阴性;可以特异地结合β-内啡肽。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:HGSMC细胞、OCIAML2细胞、Lu-99A细胞
RGC-6 Cells;背景说明:胶质细胞株C6是由Benda等用N-nitrosomethylurea诱导的大鼠胶质瘤克隆,并经过一系列的体外培养和动物传代交替后建成的。 当细胞从低密度生长到满瓶时,S-100产量增加10倍。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:RPMI7666细胞、RKO-AS45-1细胞、RCC10 RGB细胞
RGC5 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HeLa.S3细胞、L- cell细胞、SW839细胞
PA12 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:R-1059-D细胞、Tadarida brasiliensis 1 lung细胞、EMT-6细胞
LICR-HN6 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NK10a细胞、MD Anderson-Metastatic Breast-468细胞、T-ALL 1细胞
BALB/c 3T3 clone A31 Cells;背景说明:胚胎;成纤维;自发永生;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SVHUC细胞、SUM-159细胞、H-1573细胞
SNE 3 Cells(提供STR鉴定图谱)
WEHI-231 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H2170细胞、CCC-HPF-1细胞、SW-900细胞
Hs 683.T Cells;背景说明:该细胞源自76岁白人男性的左颞叶侧胶质瘤组织,有微绒毛,无桥粒。 ;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:TPC1细胞、RPTC细胞、HS5细胞
LMH Cells;背景说明:肝癌;雄性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SAOS 2细胞、Madin-Darby Canine Kidney细胞、NCI-H920细胞
Hs 870.T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代;每周换液2-3次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RPVSMC细胞、MRC5细胞、KYSE510细胞
U-118MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PK-15细胞、293EBNA细胞、HPDEC细胞
HLEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:50.B1细胞、HEK293A细胞、KE-39细胞
Capan-1人胰腺癌细胞代次低|培养基|送STR图谱
U118 Cells;背景说明:注意: 据报道来自不同个体的胶质母细胞瘤细胞株U-118 MG (HTB-15) 和 U-138 MG (HTB-16)有着一致的VNTR和相近的STR模式。 U-118 MG 和 U-138 MG细胞遗传学上很相似并有至少六个衍生标记染色体。 这是1966年至1969年间J. Ponten和同事从恶性神经胶质瘤中构建的细胞株中的一株(其它包括ATCC HTB-14和 ATCC HTB-16 and ATCC HTB-17)。 1987年用BM-Cycline培养6周去除了支原体污染。 ;传代方法: 消化3-5分钟。1:2传代。3天内可长满。;生长特性:贴壁生长;形态特性:混合型;相关产品有:HNE-2细胞、GM17346细胞、MRAEC细胞
HuH1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H747细胞、Wien133细胞、AKR细胞
HS-294T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:混合星状和多边形;相关产品有:HF-91细胞、Hep 2细胞、LIXC002细胞
HuPT4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MC-38细胞、CEM-0细胞、Panc 04.03细胞
HOP92 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MCF.7细胞、NUGC3细胞、251 MG细胞
4175 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVCAR433细胞、TE 32.T细胞、H-865细胞
Centre Antoine Lacassagne-27 Cells;背景说明:该细胞1982年由J. Gioanni建系,源自一位56岁白人男性的舌头中倍的病变部位,角蛋白强阳性;传代方法:1:6传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:P3-X63-Ag8细胞、SK-UT-1细胞、Human Kidney-2细胞
RPMI 6666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:UMUC14细胞、PC-10细胞、LU65细胞
BayGenomics ES cell line RRO171 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHB238 Cells(提供STR鉴定图谱)
H572 Cells(提供STR鉴定图谱)
PCC3/A/1 Cells(提供STR鉴定图谱)
AOBA-1 Cells(提供STR鉴定图谱)
HPL1E Cells(提供STR鉴定图谱)
" "PubMed=7017212; DOI=10.1093/jnci/66.6.1003
Pollack M.S., Heagney S.D., Livingston P.O., Fogh J.
HLA-A, B, C and DR alloantigen expression on forty-six cultured human tumor cell lines.
J. Natl. Cancer Inst. 66:1003-1012(1981)
PubMed=7459858
Rousset M., Zweibaum A., Fogh J.
Presence of glycogen and growth-related variations in 58 cultured human tumor cell lines of various tissue origins.
Cancer Res. 41:1165-1170(1981)
PubMed=6278935; PMCID=PMC1916189
Kyriazis A.P., Kyriazis A.A., Scarpelli D.G., Fogh J., Rao M.S., Lepera R.
Human pancreatic adenocarcinoma line Capan-1 in tissue culture and the nude mouse: morphologic, biologic, and biochemical characteristics.
Am. J. Pathol. 106:250-260(1982)
PubMed=6582512; DOI=10.1073/pnas.81.2.568; PMCID=PMC344720
Mattes M.J., Cordon-Cardo C., Lewis J.L. Jr., Old L.J., Lloyd K.O.
Cell surface antigens of human ovarian and endometrial carcinoma defined by mouse monoclonal antibodies.
Proc. Natl. Acad. Sci. U.S.A. 81:568-572(1984)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=1764370; DOI=10.1038/bjc.1991.467; PMCID=PMC1977874
Barton C.M., Staddon S.L., Hughes C.M., Hall P.A., O'Sullivan C., Kloppel G., Theis B., Russell R.C.G., Neoptolemos J., Williamson R.C.N., Lane D.P., Lemoine N.R.
Abnormalities of the p53 tumour suppressor gene in human pancreatic cancer.
Br. J. Cancer 64:1076-1082(1991)
PubMed=8426738
Kalthoff H., Schmiegel W.H., Roeder C., Kasche D., Schmidt A., Lauer G., Thiele H.-G., Honold G., Pantel K., Riethmuller G., Scherer E., Maurer J., Maacke H., Deppert W.
p53 and K-RAS alterations in pancreatic epithelial cell lesions.
Oncogene 8:289-298(1993)
PubMed=8026879; DOI=10.1002/ijc.2910580207
Berrozpe G., Schaeffer J., Peinado M.A., Real F.X., Perucho M.
Comparative analysis of mutations in the p53 and K-ras genes in pancreatic cancer.
Int. J. Cancer 58:185-191(1994)
PubMed=8286197; DOI=10.1038/bjc.1994.24; PMCID=PMC1968784
Lohr J.-M., Trautmann B., Gottler M., Peters S., Zauner I., Maillet B., Kloppel G.
Human ductal adenocarcinomas of the pancreas express extracellular matrix proteins.
Br. J. Cancer 69:144-151(1994)
PubMed=8968085
Goggins M.G., Schutte M., Lu J., Moskaluk C.A., Weinstein C.L., Petersen G.M., Yeo C.J., Jackson C.E., Lynch H.T., Hruban R.H., Kern S.E.
Germline BRCA2 gene mutations in patients with apparently sporadic pancreatic carcinomas.
Cancer Res. 56:5360-5364(1996)
PubMed=9331070
Teng D.H.-F., Perry W.L. 3rd, Hogan J.K., Baumgard M.L., Bell R., Berry S., Davis T., Frank D., Frye C., Hattier T., Hu R., Jammulapati S., Janecki T., Leavitt A., Mitchell J.T., Pero R., Sexton D., Schroeder M., Su P.-H., Swedlund B., Kyriakis J.M., Avruch J., Bartel P., Wong A.K.C., Oliphant A., Thomas A., Skolnick M.H., Tavtigian S.V.
Human mitogen-activated protein kinase kinase 4 as a candidate tumor suppressor.
Cancer Res. 57:4177-4182(1997)
PubMed=10027410; DOI=10.1016/S0002-9440(10)65298-4; PMCID=PMC1850008
Ghadimi B.M., Schrock E., Walker R.L., Wangsa D., Jauho A., Meltzer P.S., Ried T.
Specific chromosomal aberrations and amplification of the AIB1 nuclear receptor coactivator gene in pancreatic carcinomas.
Am. J. Pathol. 154:525-536(1999)
PubMed=10408907; DOI=10.1016/S0304-3835(98)00380-2
Bartsch D.K., Barth P., Bastian D., Ramaswamy A., Gerdes B., Chaloupka B., Deiss Y., Simon B., Schudy A.
Higher frequency of DPC4/Smad4 alterations in pancreatic cancer cell lines than in primary pancreatic adenocarcinomas.
Cancer Lett. 139:43-49(1999)
PubMed=11169957; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1014>3.0.CO;2-U
Wallrapp C., Hahnel S., Boeck W., Soder A., Mincheva A., Lichter P., Leder G., Gansauge F., Sorio C., Scarpa A., Gress T.M.
Loss of the Y chromosome is a frequent chromosomal imbalance in pancreatic cancer and allows differentiation to chronic pancreatitis.
Int. J. Cancer 91:340-344(2001)
PubMed=11668190; DOI=10.1177/002215540104901105
Quentmeier H., Osborn M., Reinhardt J., Zaborski M., Drexler H.G.
Immunocytochemical analysis of cell lines derived from solid tumors.
J. Histochem. Cytochem. 49:1369-1378(2001)
PubMed=12692724; DOI=10.1007/s00428-003-0784-4
Sipos B., Moser S., Kalthoff H., Torok V., Lohr J.-M., Kloppel G.
A comprehensive characterization of pancreatic ductal carcinoma cell lines: towards the establishment of an in vitro research platform.
Virchows Arch. 442:444-452(2003)
PubMed=14695172
Iacobuzio-Donahue C.A., Ashfaq R., Maitra A., Adsay N.V., Shen-Ong G.L.-C., Berg K., Hollingsworth M.A., Cameron J.L., Yeo C.J., Kern S.E., Goggins M.G., Hruban R.H.
Highly expressed genes in pancreatic ductal adenocarcinomas: a comprehensive characterization and comparison of the transcription profiles obtained from three major technologies.
Cancer Res. 63:8614-8622(2003)
PubMed=15162061; DOI=10.1159/000077512
Grigorova M., Staines J.M., Ozdag H., Caldas C., Edwards P.A.W.
Possible causes of chromosome instability: comparison of chromosomal abnormalities in cancer cell lines with mutations in BRCA1, BRCA2, CHK2 and BUB1.
Cytogenet. Genome Res. 104:333-340(2004)
PubMed=15367885; DOI=10.1097/00006676-200410000-00004
Loukopoulos P., Kanetaka K., Takamura M., Shibata T., Sakamoto M., Hirohashi S.
Orthotopic transplantation models of pancreatic adenocarcinoma derived from cell lines and primary tumors and displaying varying metastatic activity.
Pancreas 29:193-203(2004)
PubMed=16912165; DOI=10.1158/0008-5472.CAN-06-0721
Calhoun E.S., Hucl T., Gallmeier E., West K.M., Arking D.E., Maitra A., Iacobuzio-Donahue C.A., Chakravarti A., Hruban R.H., Kern S.E.
Identifying allelic loss and homozygous deletions in pancreatic cancer without matched normals using high-density single-nucleotide polymorphism arrays.
Cancer Res. 66:7920-7928(2006)
PubMed=18264088; DOI=10.1038/nature06548
Edwards S.L., Brough R., Lord C.J., Natrajan R., Vatcheva R., Levine D.A., Boyd J., Reis-Filho J.S., Ashworth A.
Resistance to therapy caused by intragenic deletion in BRCA2.
Nature 451:1111-1115(2008)
PubMed=18298655; DOI=10.1111/j.1582-4934.2008.00289.x; PMCID=PMC3828895
Pilarsky C., Ammerpohl O., Sipos B., Dahl E., Hartmann A., Wellmann A., Braunschweig T., Lohr J.-M., Jesenofsky R., Friess H., Wente M.N., Kristiansen G., Jahnke B., Denz A., Ruckert F., Schackert H.K., Kloppel G., Kalthoff H., Saeger H.-D., Grutzmann R.
Activation of Wnt signalling in stroma from pancreatic cancer identified by gene expression profiling.
J. Cell. Mol. Med. 12:2823-2835(2008)
PubMed=18380791; DOI=10.1111/j.1349-7006.2008.00779.x; PMCID=PMC11158928
Suzuki A., Shibata T., Shimada Y., Murakami Y., Horii A., Shiratori K., Hirohashi S., Inazawa J., Imoto I.
Identification of SMURF1 as a possible target for 7q21.3-22.1 amplification detected in a pancreatic cancer cell line by in-house array-based comparative genomic hybridization.
Cancer Sci. 99:986-994(2008)
CLPUB00416
Oberlin L.
Treatment of pancreatic carcinoma cell lines in vitro and vivo with a monoclonal antibody against the transferrin receptor.
Thesis VMD (2009); Justus-Liebig-Universitat Giessen; Giessen; Germany
DOI=10.4172/jpb.1000057
Yamada M., Fujii K., Koyama K., Hirohashi S., Kondo T.
The proteomic profile of pancreatic cancer cell lines corresponding to carcinogenesis and metastasis.
J. Proteomics Bioinformatics 2:1-18(2009)
PubMed=20037478; DOI=10.4161/cbt.8.21.9685; PMCID=PMC2824894
Kent O.A., Mullendore M.E., Wentzel E.A., Lopez-Romero P., Tan A.-C., Alvarez H., West K.M., Ochs M.F., Hidalgo M., Arking D.E., Maitra A., Mendell J.T.
A resource for analysis of microRNA expression and function in pancreatic ductal adenocarcinoma cells.
Cancer Biol. Ther. 8:2013-2024(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20418756; DOI=10.1097/MPA.0b013e3181c15963; PMCID=PMC2860631
Deer E.L., Gonzalez-Hernandez J., Coursen J.D., Shea J.E., Ngatia J.G., Scaife C.L., Firpo M.A., Mulvihill S.J.
Phenotype and genotype of pancreatic cancer cell lines.
Pancreas 39:425-435(2010)
PubMed=21750719; DOI=10.1371/journal.pone.0021639; PMCID=PMC3130048
Barber L.J., Rosa Rosa J.M., Kozarewa I., Fenwick K., Assiotis I., Mitsopoulos C., Sims D., Hakas J., Zvelebil M., Lord C.J., Ashworth A.
Comprehensive genomic analysis of a BRCA2 deficient human pancreatic cancer.
PLoS ONE 6:E21639-E21639(2011)
PubMed=22109279; DOI=10.1007/s13577-011-0037-9
Ding Q., Yoshimitsu M., Kuwahata T., Maeda K., Hayashi T., Obara T., Miyazaki Y., Matsubara S., Natsugoe S., Takao S.
Establishment of a highly migratory subclone reveals that CD133 contributes to migration and invasion through epithelial-mesenchymal transition in pancreatic cancer.
Hum. Cell 25:1-8(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
DOI=10.4172/2324-9293.1000104
Wagenhauser M.U., Ruckert F., Niedergethmann M., Grutzmann R., Saeger H.-D.
Distribution of characteristic mutations in native ductal adenocarcinoma of the pancreas and pancreatic cancer cell lines.
Cell Biol. Res. Ther. 2:1000104.1-1000104.5(2013)
PubMed=25167228; DOI=10.1038/bjc.2014.475; PMCID=PMC4453732
Hamidi H., Lu M., Chau K., Anderson L., Fejzo M.S., Ginther C., Linnartz R., Zubel A., Slamon D.J., Finn R.S.
KRAS mutational subtype and copy number predict in vitro response of human pancreatic cancer cell lines to MEK inhibition.
Br. J. Cancer 111:1788-1801(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27259358; DOI=10.1074/mcp.M116.058313; PMCID=PMC4974343
Humphrey E.S., Su S.-P., Nagrial A.M., Hochgrafe F., Pajic M., Lehrbach G.M., Parton R.G., Yap A.S., Horvath L.G., Chang D.K., Biankin A.V., Wu J.-M., Daly R.J.
Resolution of novel pancreatic ductal adenocarcinoma subtypes by global phosphotyrosine profiling.
Mol. Cell. Proteomics 15:2671-2685(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"