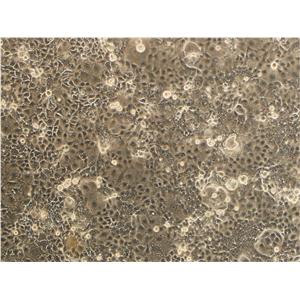
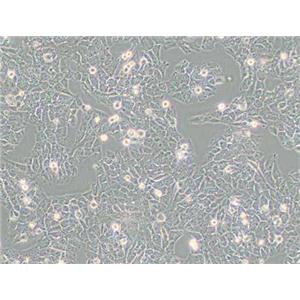
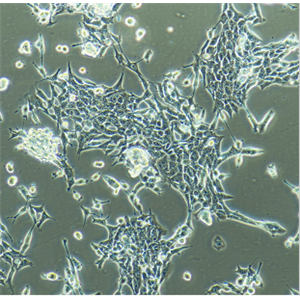
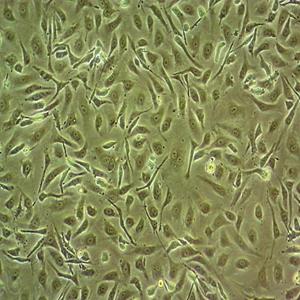
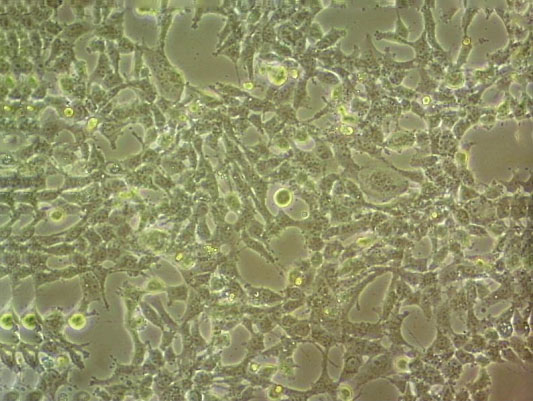
"SK-N-AS人神经母细胞瘤细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
H-2171 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:H-2030细胞、MO59J细胞、HIT T-15细胞
HMC-1 Cells;背景说明:肥大 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:ML-2细胞、NCI-H711细胞、H35 Reuber细胞
H2195 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-SNU-449细胞、COLO679细胞、NCI H716细胞
SK-N-AS人神经母细胞瘤细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
ATCC细胞库(American Type Culture Colection),该中心一直致力于细胞分类、鉴定和保藏工作。ATCC是全球最大的生物资源保藏中心,ATCC通过行业标准产品、服务和创新解决方案支持全球学术、政府、生物技术、制药、食品、农业和工业领域的科学进步。ATCC提供的服务和定制解决方案包括细胞和微生物培养、鉴定、生物衍生物的开发和生产、性能测试和生物资源保藏服务。美国国家标准协会(ANSI)认可了ATCC标准开发组织,并制定了标准协议,以确保生物材料的可靠性和可重复性。ATCC的使命是为了获取、鉴定、保存、开发、标准化和分发生物资源和生物信息,以提高和应用生物科学知识。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
SK-N-AS人神经母细胞瘤细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
HSC2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HUT125细胞、HEC251细胞、NKM-1细胞
CWR22-Rv1 Cells;背景说明:22RV1是来自异种移植(在阉割引起前列腺癌衰退又在其父亲的雄性激素信赖型CWR22嫁接后复发的小鼠中连续传代)的人前列腺癌上皮细胞系。此细胞系表达前列腺特异抗原。二羟基睾丸脂酮轻微刺激细胞生长,经westernblot检测溶解产物与抗雄性激素受体抗体起免疫反应。EGF刺激细胞生长,但TGFβ-1不能抑制细胞生长。该细胞在裸鼠中成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:P3-X63-Ag8-6-5-3细胞、Hs822T细胞、Panc203细胞
H35 Reuber Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CV-1 in Origin Simian-1细胞、RN细胞、MMAc-SF细胞
CCRF/CEM-C7 Cells;背景说明:急性T淋巴细胞白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SLMT-1细胞、COLO206细胞、SCCVII/St细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
正确的细胞复苏需知事项:细胞冻存HAO了,接下来要注意什么问题呢?没错,就是记得到时间了,拿出来复苏。那么,细胞复苏的过程中又有哪些该注意的事项呢?细胞活力和形态检查的作用何在?活力检查——千万不要使用不健康的细胞,可能有污染(真菌、支原体等),如果发现有污染毫不犹豫的丢弃!形态检查——检查细胞的固有形态和生长行为。冻存细胞:补充新的培养——在您开始冻存细胞的前一天补充新的培养。在细胞长至70%单层时收获细胞,计数活细胞数,用冻存调整细胞密度~5 x106 s/ml (根据不同的细胞类型调整);冻存——用冻存洗细胞并用冻存重悬细胞,有不同类型的冻存,根据细胞类型选择Zui合适的冻存(常用的冻存成分有):5-10% DMSO——注意确保DMSO不含有其他的毒性物质;5-15%甘油;如果细胞在无血清培养基内生长,应在50%条件培养基内(细胞在无血清培养基内生长24小时)内冻存和复苏。在冻存管上标记HAO细胞类型,日期,冻存人等信息,并保证每冻存管不超过1.5ml。放入罐之前记录冻存管的数量和位置。以Zui快的速度转移冻存管知罐内,因此,此步骤ZuiHAO使用干冰,或者把冻存管浸入装有的小盒内。此外还要注意,在冻存管上没有足够的空间记录细胞的详细信息,做HAO记录是非常非常重要的!还有一个Zui重要的,一定要在异地的罐内保存同样的一份细胞,以免其中的一个罐出现问题!细胞正确的复苏方式和正确的冻存方式同样重要,熟记以下要点:当从罐内取出细胞时,有可能会出现冻存管破裂的情况,使用保护面罩和防护服十分必要;其实,细胞复苏只是一个简单的实验,不过这其中却不可避免有一些需要注意的细节,不然,也不一定会尽如人意。例如说,人身健康方面:一定要记得做HAO防冻工作,戴上护目镜;尽量降低DMSO对细胞的损伤等等。
NB-19 Cells;背景说明:神经母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HREC细胞、PGLH7细胞、F98细胞
Vero C1008 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO 206F细胞、SU 86.86细胞、TW01细胞
Rat Skin 1 Cells;背景说明:该细胞系来源于一大鼠的皮肤组织。2007年由中国科学院昆明细胞库建立。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:UCLA SO M21细胞、B16BL-6细胞、CAL39细胞
CD18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:LUDLU 1细胞、MARC-145细胞、HCC0827细胞
MIA Paca2 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HUT-28细胞、rRTEC细胞、BL2141细胞
L-M(TK-) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GB-1细胞、BALB/3T3细胞、KMM1细胞
HLFa Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:H1563细胞、SNU-398细胞、H2196细胞
Instituto Biologico-Rim Suino-2 Cells;背景说明:肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SSP-25细胞、H6c7细胞、HANK细胞
MLOY4 Cells;背景说明:骨;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PC615.3细胞、GM346细胞、GM00346细胞
MCF.7 Cells;背景说明:MCF-7细胞保留了多个分化了的乳腺上皮的特性,包括:能通过胞质雌激素受体加工雌二醇并能形成圆形复合物(domes)。该细胞含有Tx-4癌基因。肿瘤坏死因子α(TNFalpha)可以抑制MCF-7细胞的生长。抗雌激素处理细胞能调变IGFBP'S的分泌。;传代方法:1:2传代,3-4天长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SKNEP1细胞、HCEC细胞、AGS细胞
COLO-738 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Roswell Park Memorial Institute 1846细胞、M3 Clone M-3细胞、GM05862细胞
CV-1.K Cells;背景说明:CV-1细胞株是1964年由JensenFC等建系的,源自成年雄性非洲绿猴肾,被用于Rous肉瘤病毒的转染研究。可作为SV40载体的转染宿主。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:OVMANA细胞、Ku812F细胞、RH8994细胞
WISH Cells;背景说明:最初以为这株细胞的起源是正常羊膜,但随后通过同工酶分析、HeLa标记染色体和DNA指纹法分析,证实该细胞是HeLa细胞污染的;角蛋白阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长 ;形态特性:上皮细胞样;相关产品有:OLN 93细胞、OVCA433细胞、SW-1116细胞
BEL 7405 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:NCI-H128细胞、D-341细胞、IOSE80UBC细胞
SNG-M Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:多边形;相关产品有:SKMEL5细胞、OVCA432细胞、CCC-HIE-2细胞
KU-812-F Cells;背景说明:慢性粒细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SEM细胞、Mevo细胞、OCIAML2细胞
Sf-21 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KAT5细胞、MiaPaca.2细胞、PC10细胞
1301-U2 Cells(提供STR鉴定图谱)
Abcam HeLa CEBPB KO Cells(提供STR鉴定图谱)
AG15982 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRE286 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG128 Cells(提供STR鉴定图谱)
C0028 Cells(提供STR鉴定图谱)
CW10014 Cells(提供STR鉴定图谱)
DA06468 Cells(提供STR鉴定图谱)
GM01019 Cells(提供STR鉴定图谱)
CCLP-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NPC-TW-039细胞、CORL51细胞、A20细胞
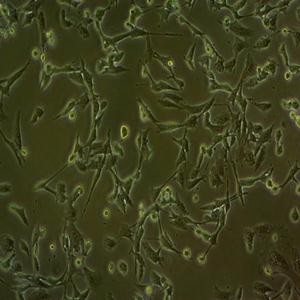
SK-N-AS人神经母细胞瘤细胞代次低|培养基|送STR图谱
HMCB Cells;背景说明:详见相关文献介绍;传代方法:1:6—1:10传代,2天换液1次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Madin-Darby Bovine Kidney细胞、MDCKII-WT细胞、NPC-TW 01细胞
MKN 7 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MCAEC细胞、COR-L279细胞、JEG-3细胞
OVTOKO Cells;背景说明:卵巢透明细胞癌;脾转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RL-65细胞、Tn 5B1-4细胞、ketr 3细胞
Hs840_T Cells;背景说明:详见相关文献介绍;传代方法:1:4—1:8传代,每周换液2—3次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:LIXC002细胞、578T细胞、EoL-1 cell细胞
SW-1417 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液1-2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Jurkat FHCRC细胞、NCI-H889细胞、JURKAT E-6.1细胞
30G8 Cells(提供STR鉴定图谱)
PC 61.5.3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:alpha TC1 clone 6细胞、SK-MEL-MeWo细胞、U-251-MG细胞
H-1435 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长,松散;形态特性:详见产品说明书;相关产品有:YD15细胞、H-2135细胞、OCI/AML2细胞
HIEC-6 Cells;背景说明:肠;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM04679细胞、SW1573细胞、RPMI2650细胞
MKN 7 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:MCAEC细胞、COR-L279细胞、JEG-3细胞
PTK2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:JIII细胞、OVCAR3细胞、MDA-MB-435 S细胞
Jiyoye Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:ABC-1细胞、LNCaP C4-2细胞、3AO细胞
G292 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Nb 2细胞、HS940细胞、Lymph Node Carcinoma of the prostate细胞
RIN-m Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MOLM-13细胞、DMS 53细胞、BMU-S1细胞
GM24104 Cells(提供STR鉴定图谱)
HAP1 NDUFS3 (-) 3 Cells(提供STR鉴定图谱)
HBL-1 [Human diffuse large B-cell lymphoma] Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RPMI8402细胞、NIH3T3细胞、C 643细胞
UACC-812 Cells;背景说明:该细胞是由Liebovitz A等于1986年从一名43岁的白人女性乳腺导管癌患者的乳腺切除肿瘤组织中分离建立的;手术前该病人曾接受过广泛的化疗。该细胞HER-2/neu癌基因序列有15倍的扩增;雌激素受体ER、孕激素受体PR和糖蛋白P阴性。;传代方法:1:3传代;5-7天1次。 ;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HT144-mel细胞、CL 1-5细胞、H69C细胞
GT39 Cells;背景说明:胃癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI.H460细胞、RPMI8226细胞、HCC1588细胞
Loucy Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:Hep G2/C3A细胞、Mc Ardle 7777细胞、Hs294T细胞
EFO-27 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PLA-802细胞、NCIH1092细胞、PNT1A细胞
SKES1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:5传代;每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:Madison 109细胞、Hs819.T细胞、HONE1细胞
HOSEpiC Cells;背景说明:卵巢;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Toledo细胞、Hs-27细胞、P-36细胞
HUVEC-C[HUVEC] Cells;背景说明:脐静脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:N1S1细胞、M-07e细胞、Virginia Mason Research Center-Lung Cancer D细胞
HQ02285 Cells(提供STR鉴定图谱)
KHm-5 Cells(提供STR鉴定图谱)
MIZMe011-A Cells(提供STR鉴定图谱)
NP3 [Human lung] Cells(提供STR鉴定图谱)
RCM-2 Cells(提供STR鉴定图谱)
Ubigene HeLa TPCN1 KO Cells(提供STR鉴定图谱)
XPH13PV Cells(提供STR鉴定图谱)
HEK-Blue IL-18 Cells(提供STR鉴定图谱)
GP-293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Mo7e细胞、NCI-522细胞、LCMS细胞
HSC1 Cells;背景说明:皮肤鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PC 61 5.3细胞、HLMVEC细胞、HT1376细胞
MDA231-LM2-4175 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CSQT-2细胞、Leukemia L1210细胞、HCT-FET细胞
NCI-H-295 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KM932细胞、LN-229细胞、NCCIT细胞
CCD-966SK Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3X63Ag8653细胞、H-1836细胞、PJ34细胞
CCD-966SK Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P3X63Ag8653细胞、H-1836细胞、PJ34细胞
KOSC2 Cells;背景说明:详见相关文献介绍;传代方法:2.2 x 10^4 cells/ml ;生长特性:贴壁生长;形态特性:上皮样;相关产品有:TALL-104细胞、SU-DHL2细胞、HGF-1细胞
U373-MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Human Melanoma Cell Bowes细胞、HCC1954细胞、Me-Wo细胞
Human Epidermoid carcinoma #2 Cells;背景说明:最初认为这个细胞源自喉上皮癌,但随后通过同功酶分析、HeLa标记染色体和DNA指纹分析发现,起源细胞已被HeLa污染。 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCC1569细胞、NBL-4细胞、NS20Y细胞
K299 Cells;背景说明:间变性大细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CL34细胞、H-2347细胞、NK-92细胞
CCRF-SB Cells;背景说明:急性T淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RT-BM 1细胞、NS1-1 Ag4.1细胞、C12细胞
CEF Cells;背景说明:胚胎;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Mesothelial cells transfected with pRSV-T 5A细胞、343 MG细胞、MALME 3M细胞
NB19-RIKEN Cells;背景说明:神经母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MODE-K细胞、NCI-H2342细胞、IPEC-J2细胞
G-Olig2 Cells;背景说明:胚胎干细胞;129X1/SvJ;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C3H10T1-2细胞、SNUC1细胞、Calu 6细胞
JCA-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NTC-200细胞、H-1975细胞、Fox/NY细胞
STBCi020-B Cells(提供STR鉴定图谱)
L6565 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM00637F细胞、MB157细胞、DU145细胞
211H Cells;背景说明:MSTO-211H细胞株是1985年从一位肺二相间皮瘤患者的胸水中建株的。这个病人接受过多种药物联合前期化疗。MSTO-211H细胞具有高亲和力的EGF结合位点,并表达神经元特异性烯醇酶(NSE)及人绒毛膜促性腺激素(HCG)的α与β亚基。未检测到左旋多巴胺脱羧酶(DDC),邦巴辛与神经tensin。细胞过表达c-myc原癌基因,并没有观察到基因重排或扩增。V-src,v-abl,v-erbB,c-raf1,Ha-ras,Ki-ras,和N-ras的表达呈阳性。未检测到N-m;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:Gejiu Lung Carcinoma-82细胞、TO175T细胞、HSC 3细胞
8505C Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:BGC823细胞、NCI-H1930细胞、Danio rerio Gill细胞
SNU-5 Cells;背景说明:该细胞来源于一名低分化胃癌患者的转移性腹水,1987年分离建立。该细胞表达CEA和TAG-72。;传代方法:2-3天补液一次。;生长特性:多细胞聚集、悬浮生长;形态特性:上皮细胞样;相关产品有:SKLU01细胞、HCT_116细胞、KYSE-50细胞
NCI-H1395 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;5-6天传代一次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:NCI-HUT-520细胞、MESSA细胞、H-748细胞
DU 145 Cells;背景说明:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,酸性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:Fetal Human Lens-124细胞、McA-RH 8994细胞、U118-MG细胞
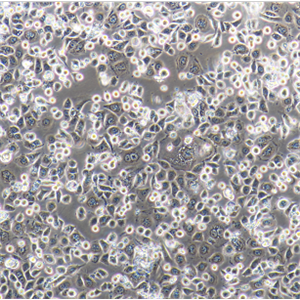
SK-N-AS人神经母细胞瘤细胞代次低|培养基|送STR图谱
Cor L88 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCI-H1573细胞、SW-900细胞、CV1细胞
M059K Cells;背景说明:详见相关文献介绍;传代方法:1:6-1:8传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NMCG1细胞、C-Lu65细胞、M07e细胞
Vero C1008 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COLO 206F细胞、SU 86.86细胞、TW01细胞
OVCAR3 Cells;背景说明:该细胞1982年由T.C. Hamilton等建系,源自一位60卵巢腺癌的腹水,是卵巢癌抗药性研究的模型。;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BLO-11细胞、MAntle cell VERona-1细胞、HBE细胞
HRGEC Cells;背景说明:肾小球;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OCI-LY-18细胞、MDA-231细胞、TCam-2细胞
SUM190PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PLA-801C细胞、HCT.116细胞、RL95细胞
LM TK negative Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC941122细胞、Normal Rat Kidney-49F细胞、GP2d细胞
RA Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LA-N-5细胞、H-2073细胞、HUC细胞
BayGenomics ES cell line RHA082 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XD135 Cells(提供STR鉴定图谱)
CPTC-S100A4-2 Cells(提供STR鉴定图谱)
MECA-89 Cells(提供STR鉴定图谱)
SFA1.2B4 Cells(提供STR鉴定图谱)
LNCaP-luc2 Cells(提供STR鉴定图谱)
" "DOI=10.1016/B978-0-12-333530-2.50006-X
Israel M.A., Thiele C.J.
Tumor cell lines of the peripheral nervous system.
(In book chapter) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.43-78; Academic Press; New York; USA (1994)
PubMed=7838528
Cheng N.C., Van Roy N., Chan A., Beitsma M., Westerveld A., Speleman F., Versteeg R.
Deletion mapping in neuroblastoma cell lines suggests two distinct tumor suppressor genes in the 1p35-36 region, only one of which is associated with N-myc amplification.
Oncogene 10:291-297(1995)
PubMed=9283597; DOI=10.1016/S0165-4608(96)00362-7
Van Roy N., Jauch A., Van Gele M., Laureys G., Versteeg R., De Paepe A., Cremer T., Speleman F.
Comparative genomic hybridization analysis of human neuroblastomas: detection of distal 1p deletions and further molecular genetic characterization of neuroblastoma cell lines.
Cancer Genet. Cytogenet. 97:135-142(1997)
DOI=10.1007/0-306-46872-7_2
Thiele C.J.
Neuroblastoma.
(In book chapter) Human cell culture. Vol. 1. Cancer cell lines part 1; Masters J.R.W., Palsson B.O. (eds.); pp.21-53; Kluwer Academic Publishers; New York; USA (1999)
PubMed=11550280; DOI=10.1002/gcc.1174
Van Roy N., Van Limbergen H., Vandesompele J., Van Gele M., Poppe B., Salwen H.R., Laureys G., Manoel N., De Paepe A., Speleman F.
Combined M-FISH and CGH analysis allows comprehensive description of genetic alterations in neuroblastoma cell lines.
Genes Chromosomes Cancer 32:126-135(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=12702577
Saito-Ohara F., Imoto I., Inoue J., Hosoi H., Nakagawara A., Sugimoto T., Inazawa J.
PPM1D is a potential target for 17q gain in neuroblastoma.
Cancer Res. 63:1876-1883(2003)
PubMed=15892104; DOI=10.1002/gcc.20198
Mosse Y.P., Greshock J., Margolin A.A., Naylor T., Cole K.A., Khazi D., Hii G., Winter C., Shahzad S., Asziz M.U., Biegel J.A., Weber B.L., Maris J.M.
High-resolution detection and mapping of genomic DNA alterations in neuroblastoma.
Genes Chromosomes Cancer 43:390-403(2005)
PubMed=16822308; DOI=10.1186/1471-2407-6-177; PMCID=PMC1533846
Dam V., Morgan B.T., Mazanek P., Hogarty M.D.
Mutations in PIK3CA are infrequent in neuroblastoma.
BMC Cancer 6:177.1-177.10(2006)
PubMed=17506115; DOI=10.1002/nbm.1181
Peet A.C., McConville C.M., Wilson M.P., Levine B.A., Reed M., Dyer S.A., Edwards E.C., Strachan M.C., McMullan D.J., Wilkes T.M., Grundy R.G.
1H MRS identifies specific metabolite profiles associated with MYCN-amplified and non-amplified tumour subtypes of neuroblastoma cell lines.
NMR Biomed. 20:692-700(2007)
PubMed=18082704; DOI=10.1016/j.jpedsurg.2007.08.026
Komuro H., Saihara R., Shinya M., Takita J., Kaneko S., Kaneko M., Hayashi Y.
Identification of side population cells (stem-like cell population) in pediatric solid tumor cell lines.
J. Pediatr. Surg. 42:2040-2045(2007)
PubMed=18534018; DOI=10.1186/1476-4598-7-50; PMCID=PMC2442611
Combaret V., Boyault S., Iacono I., Brejon S., Rousseau R., Puisieux A.
Effect of bortezomib on human neuroblastoma: analysis of molecular mechanisms involved in cytotoxicity.
Mol. Cancer 7:50.1-50.12(2008)
PubMed=18724359; DOI=10.1038/nature07261; PMCID=PMC2672043
Mosse Y.P., Laudenslager M., Longo L., Cole K.A., Wood A., Attiyeh E.F., Laquaglia M.J., Sennett R., Lynch J.E., Perri P., Laureys G., Speleman F., Kim C., Hou C.-P., Hakonarson H., Torkamani A., Schork N.J., Brodeur G.M., Tonini G.P., Rappaport E., Devoto M., Maris J.M.
Identification of ALK as a major familial neuroblastoma predisposition gene.
Nature 455:930-935(2008)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20655465; DOI=10.1016/j.cell.2010.06.004; PMCID=PMC2913027
Holzel M., Huang S.-D., Koster J., Ora I., Lakeman A., Caron H.N., Nijkamp W., Xie J., Callens T., Asgharzadeh S., Seeger R.C., Messiaen L.M., Versteeg R., Bernards R.
NF1 is a tumor suppressor in neuroblastoma that determines retinoic acid response and disease outcome.
Cell 142:218-229(2010)
PubMed=22213050; DOI=10.1002/ijc.27415; PMCID=PMC3757132
Gawecka J.E., Geerts D., Koster J., Caliva M.J., Sulzmaier F.J., Opoku-Ansah J., Wada R.K., Bachmann A.S., Ramos J.W.
PEA15 impairs cell migration and correlates with clinical features predicting good prognosis in neuroblastoma.
Int. J. Cancer 131:1556-1568(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23202128; DOI=10.1038/ng.2493; PMCID=PMC3557959
Sausen M., Leary R.J., Jones S., Wu J., Reynolds C.P., Liu X.-Y., Blackford A.L., Parmigiani G., Diaz L.A. Jr., Papadopoulos N., Vogelstein B., Kinzler K.W., Velculescu V.E., Hogarty M.D.
Integrated genomic analyses identify ARID1A and ARID1B alterations in the childhood cancer neuroblastoma.
Nat. Genet. 45:12-17(2013)
PubMed=24466371; DOI=10.1593/tlo.13544; PMCID=PMC3890703
Loschmann N., Michaelis M., Rothweiler F., Zehner R., Cinatl J., Voges Y., Sharifi M., Riecken K., Meyer J., von Deimling A., Fichtner I., Ghafourian T., Westermann F., Cinatl J. Jr.
Testing of SNS-032 in a panel of human neuroblastoma cell lines with acquired resistance to a broad range of drugs.
Transl. Oncol. 6:685-696(2013)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28192521; DOI=10.1371/journal.pone.0172140; PMCID=PMC5305101
Saintas E., Abrahams L., Ahmad G.T., Ajakaiye A.-O.M., AlHumaidi A.S.H.A.M., Ashmore-Harris C., Clark I., Dura U.K., Fixmer C.N., Ike-Morris C., Mato Prado M., McCullough D., Mishra S., Scholer K.M.U., Timur H., Williamson M.D.C., Alatsatianos M., Bahsoun B., Blackburn E., Hogwood C.E., Lithgow P.E., Rowe M., Yiangou L., Rothweiler F., Cinatl J. Jr., Zehner R., Baines A.J., Garrett M.D., Gourlay C.W., Griffin D.K., Gullick W.J., Hargreaves E., Howard M.J., Lloyd D.R., Rossman J.S., Smales C.M., Tsaousis A.D., von der Haar T., Wass M.N., Michaelis M.
Acquired resistance to oxaliplatin is not directly associated with increased resistance to DNA damage in SK-N-ASrOXALI4000, a newly established oxaliplatin-resistant sub-line of the neuroblastoma cell line SK-N-AS.
PLoS ONE 12:E0172140-E0172140(2017)
PubMed=28350380; DOI=10.1038/sdata.2017.33; PMCID=PMC5369315
Harenza J.L., Diamond M.A., Adams R.N., Song M.M., Davidson H.L., Hart L.S., Dent M.H., Fortina P., Reynolds C.P., Maris J.M.
Transcriptomic profiling of 39 commonly-used neuroblastoma cell lines.
Sci. Data 4:170033-170033(2017)
PubMed=30459281; DOI=10.1126/scisignal.aar5680
Van den Eynden J., Umapathy G., Ashouri A., Cervantes-Madrid D., Szydzik J., Ruuth K., Koster J., Larsson E., Guan J.-K., Palmer R.H., Hallberg B.
Phosphoproteome and gene expression profiling of ALK inhibition in neuroblastoma cell lines reveals conserved oncogenic pathways.
Sci. Signal. 11:eaar5680.1-eaar5680.16(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"