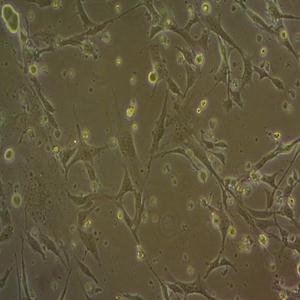
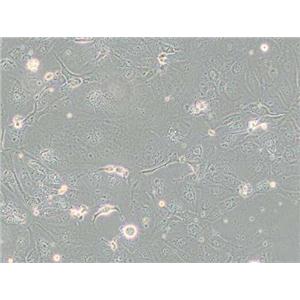
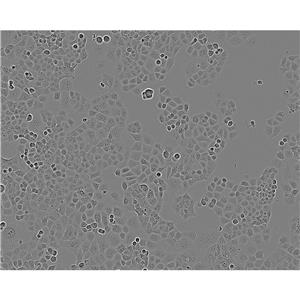
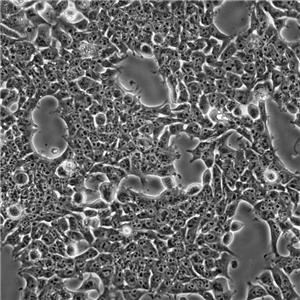
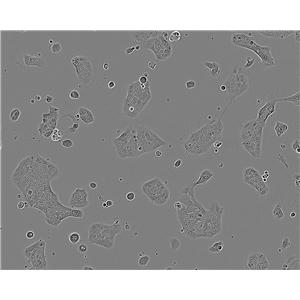
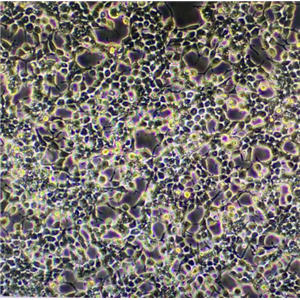
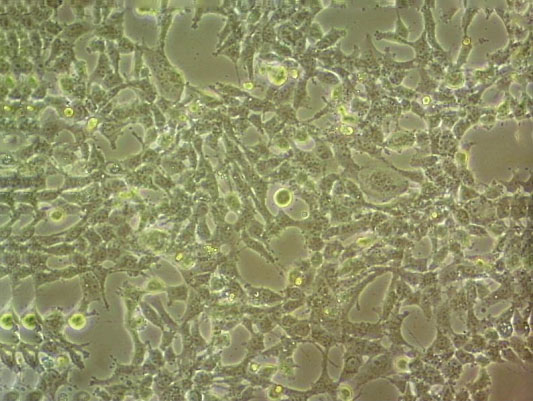
"NCI-H2347人肺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
公司细胞系形态漂亮、增殖倍数高、纯度高、功能性强,细胞培养就跟养孩子一个样。养孩子要喂奶,养细胞要加补液,都需要在前期补充足够的营养,初始状态的细胞或刚刚复苏的细胞还要适量加入血清或细胞因子来帮助它们的存活增殖,如果营养物质缺乏,细胞就会不生长甚至死亡。养孩子要从小培养学习,养细胞也得培养宝宝顺利生下来,你会经常抚摸他,给他看各种颜色,刺激他的五感。细胞也是一样,分离后的细胞需要使用特定的细胞因子进行活化、增殖。另外加入因子的种类、因子的浓度、加入时间、加入顺序都会影响细胞最终的结果。养孩子最怕孩子生病,养细胞最怕被污染,平时你会仔细观察宝宝是否呕吐、是否突然哭闹,猜测宝宝是否生病了。对于细胞,我们也需要时刻进行观察的,假如培养液浑浊(污染了),则需要换液后加抗生素;假如细胞增殖不明显,形态变差,则可能是因为营养不足了,对贴壁细胞可以消化后重新用新的培养基接种并加倍加入细胞因子含量;对悬浮细胞增殖能力不强的,则不着急补液,只是先补加血清、细胞因子看是否可以好转。培养时还得全程在无菌的环境,一个小小的偏差,细胞就会死亡。
换液周期:每周2-3次
WRL 68 Cells;背景说明:胚胎;肝 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RAT2细胞、LIXC002细胞、U 138 MG细胞
YD-15 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Ketr3细胞、NSC34细胞、Panc-10.05细胞
LLC-MK2 Cells;背景说明:胚胎;肾;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Primary Liver Carcinoma/Poliomyelitis Research Foundation/5细胞、PC3细胞、L-M (TK-)细胞
NCI-H2347人肺癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
NCI-H2347人肺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
NCIH2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:74Int细胞、HPAFII细胞、Homo sapiens No. 578, breast cells细胞
CAL 33 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HO1-N1细胞、HOP 92细胞、KM932细胞
ML2 Cells;背景说明:急性髓单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HARA-B细胞、H-526细胞、SGC996细胞
CFPAC1 Cells;背景说明:详见相关文献介绍;传代方法:1:3-10传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:PJ34细胞、143B TK-细胞、G-402细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
贴壁细胞的消化方法介绍:1、胰酶。这是用得Zui多的。一般浓度在0.25-0.5%。作用时间根据细胞种类、作用温度等因素而变化很大,从几分钟到几十分钟不等。0.25%的胰酶作用于单层贴壁的细胞,在37度条件下,一般消化1-5分钟就足够了。终止是用血清。主要作用于细胞间。配制时不能用含、镁的平衡,否则影响活性。保存于-20度。2、胶原酶。这种方法比较少,一般是用原代培养时,从组织消化下细胞。这种方法作用温和,对细胞损伤较小,但是,价格也较贵。中止同样是用血清。3、EDA。用得也是非常多。一般浓度在0.02%左右。作用于细胞与间质,对细胞间也有一定作用。注意,它能显著影响pH值,而且在弱碱性条件下才易溶。因此,配制时应调节HAO碱度。它不能被终和。因此,消化下来的细胞要洗一遍。4、商品化的无酶消化。个人的使用经常觉得对细胞的损伤比较大,但是分离成单细胞悬的能力确实比较强。5、物理法。直接吹打或用细胞刮子将细胞刮下来。6、冷冻法。此方法仅能用于细胞传代时。无法使组织上的细胞脱落下来。本方法的原理,我想是因细胞冷冻后收缩,从而从培养瓶上脱落下来。YOU点是:对细胞损伤小,不需要中止或洗细胞,方便,不需要另外配制消化。别适用那些贴壁不是别紧,又别娇气的细胞。不足是细胞常成小片脱落。此种方法曾用于因用其它方法传代导致大量细胞死亡操作的间充质干细胞、DC细胞的培养,效果非常满意。具体过程是:1、用较多的4度的PBS 洗涤一遍细胞(以6孔板为例,加1.5ml/孔),2、再加0.5毫升4度的PBS,静置操作台上,很快细胞就小片脱落,3、轻轻吹打,细胞即完全脱落,4、按一定比例传代。
U-CH1 Cells;背景说明:骶骨脊索瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CAL33细胞、Rin-M-5F细胞、CT26WT细胞
L cell line Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:IBMF-7细胞、Hs839T细胞、SUM102PT细胞
H-1650 Cells;背景说明:该细胞是从一名27岁白人男性(10年烟龄)支气管肺泡癌患者的胸腔积液中分离得到的。;传代方法:1:4-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:IPLB-SF 21细胞、DAKIKI Clone 1细胞、U 266细胞
L78 Cells;背景说明:肺腺;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L929(NCTC)细胞、HEC-151细胞、MCF-12F细胞
JM-Jurkat Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:PFSK-1细胞、TE85细胞、Hs274T细胞
MPVECs Cells;背景说明:肺;微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WM 239A细胞、PL-9细胞、Hs819.T细胞
R D Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:CAL 120细胞、MDA-MB-175VII细胞、SUM 149细胞
PC 12 Cells;背景说明:该细胞系来自能移植的雄性大鼠肾上腺嗜铬细胞瘤。这些细胞表达神经生长因子(NGF)受体。NGF可诱导产生神经表型。这些细胞不合成。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:多角形;相关产品有:RPMI-2650细胞、RPMI 6666细胞、SKN-SH细胞
3T3-L1 ad Cells;背景说明:3T3-L1是从3T3细胞(Swissalbino)中经克隆分离得到的连续传代的亚系。该细胞从快速分裂到汇合和接触性抑制状态经历了前脂肪细胞到脂肪样细胞的转变。该细胞鼠痘病毒阴性;可产生甘油三酯,高浓度血清可增强细胞内脂肪堆积。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:D-283 Med细胞、Hs 819.T细胞、H2009细胞
RCC10 RGB Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC1187细胞、HEC-1B细胞、Rat Skin 1细胞
CEM-0 Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:33604细胞、NCIH1341细胞、NB1-RGB细胞
bEnd3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OCI-LY-7细胞、T 98 G细胞、HEL9217细胞
B16 BL6 Cells;背景说明:黑色素瘤;雄性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KOSC2细胞、HeLa/S3细胞、Jurkat-77细胞
C643 Cells;背景说明:甲状腺未分化癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:VCaP细胞、CL1细胞、Hepa1-6细胞
HOS TE 85 Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:293AD细胞、LI7细胞、MOVAS-1细胞
PIG3 Cells;背景说明:皮肤;黑色素 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HT 29细胞、786-O RCC细胞、JURKAT E-61细胞
A375-MEL Cells;背景说明:A375源自一位54岁女性,是Giard DJ等人建立的一系列细胞株中的一株。该细胞可在免疫抑制小鼠上成瘤,在琼脂上形成克隆。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:Hs 746.T细胞、SKLU-1细胞、Porcine Kidney-15细胞
29037 Cells(提供STR鉴定图谱)
Abcam HeLa RRAGB KO Cells(提供STR鉴定图谱)
AG24410 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRI156 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XH657 Cells(提供STR鉴定图谱)
C2060 Cells(提供STR鉴定图谱)
DA00055 Cells(提供STR鉴定图谱)
ES[MC1R(20):tetPou5f1(18)] Cells(提供STR鉴定图谱)
GM06651 Cells(提供STR鉴定图谱)
COLO829 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:HS-695T细胞、L-02细胞、H341细胞
NCI-H2347人肺癌细胞代次低|培养基|送STR图谱
CMT 93 Cells;背景说明:结肠癌;C57BL/ICRF;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FaDu细胞、Hs445细胞、P3/NS-1细胞
HeLa S3 Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H82细胞、H676B细胞、Mink细胞
KBM-5 Cells;背景说明:慢性髓白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:BPH-1细胞、University of Michigan-Urothelial Carcinoma-1细胞、Oregon J-111细胞
NCI H226 Cells;背景说明:1980年分离建立。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Human Microvascular Endothelial Cell line-1细胞、HCC78细胞、BAR-T细胞
SNU354 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NCI522细胞、PK (15)细胞、BN-CL2细胞
AKR/JB.Sp Cells(提供STR鉴定图谱)
COLO-741 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HPAF2细胞、Swiss/3T3细胞、MDA-435细胞
CNE Cells;背景说明:此细胞株来源于一位58岁女性鼻咽癌患者。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:JB6Cl30细胞、SP-2细胞、MC26细胞
HCC1954BL Cells;背景说明:外周血B淋巴细胞;EBV转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HA1800细胞、S-16细胞、LOU-NH-91细胞
EC9706 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:EA hy 926细胞、COR-L 105细胞、McA-RH 8994细胞
MES 13 Cells;背景说明:肾小球系膜;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MCA38细胞、HLEB-3细胞、SUM 149PT细胞
NR 8383 Cells;背景说明:NR8383(正常大鼠,1983年8月3日)来源于肺灌洗时的正常大鼠肺泡巨噬细胞。细胞在gerbil肺细胞连续培养液存在下培养了大约8-9个月。随后,不再需要外源生长因子。通过有限稀释法从单个细胞克隆并亚克隆NR8383细胞,并三次用软琼脂亚克隆。细胞表现出巨噬细胞的特性,吞噬酵母多糖和铜绿,非特异性脂酶活性,Fc受体,氧化降解;分泌IL-1,TNFbeta和IL-6,可重复地响应外源生长因子。NR8383细胞响应博莱霉素,分泌TGFbeta前体。在博莱霉素刺激下,TGFbe;传代方法:1:2传代;生长特性:半贴壁生长;形态特性:巨噬细胞;相关产品有:RH-30细胞、SF 539细胞、PLC8024细胞
Acanthosis Nigricans 3rd attempt-CArcinoma Cells;背景说明:AN3CA细胞建系于1964年。它衍生于子宫内膜癌患者淋巴结转移组织,具有癌细胞的基本特性,能在体外长期传代培养,接种实验动物产生明显肿瘤。但细胞的生物学特性及超微结构尚未深入研究,仅发现该细胞系促黑激素合成为阴性。细胞常用于人子宫内膜癌细胞生物学及其相关特性研究。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:U251细胞、SNU1040细胞、MLFC细胞
DanG Cells;背景说明:胰腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:207细胞、TGW细胞、T2(174 x CEM.T2)细胞
GM12621 Cells(提供STR鉴定图谱)
HAP1 ERCC5 (-) 3 Cells(提供STR鉴定图谱)
3D4/21 Cells;背景说明:肺泡巨噬细胞;SV40转化;Landrace;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:32D CL3细胞、MNNG/HOS Cl #5细胞、TFH细胞
HCC-1438 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LN229细胞、PA I细胞、BpRcl细胞
U343 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HEK AD293细胞、FO [Mouse myeloma]细胞、SKNBE(2)细胞
N-9 Cells;背景说明:小胶质细胞;雄性;CD-1;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:PK15细胞、MSF细胞、CG4细胞
LCLC-103H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Mouse Forestomach Carcinoma细胞、RPMI 7666细胞、PA-TU-8988S细胞
Mac-1 Cells;背景说明:皮肤T淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U87细胞、SNU761细胞、3396细胞
G-402 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:6传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:FU-MMT-1细胞、NCI-H2141细胞、MGH-U1 (EJ)细胞
SW527 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:R3/1细胞、P3/NS-1细胞、RCM-1 [Human rectum adenocarcinoma]细胞
HiPS-RIKEN-4D Cells(提供STR鉴定图谱)
IPS(IMR90)-1 Cells(提供STR鉴定图谱)
MBA72R Cells(提供STR鉴定图谱)
ND10694 Cells(提供STR鉴定图谱)
PL28 Cells(提供STR鉴定图谱)
U-1761 Cells(提供STR鉴定图谱)
UM-SCVa-1 Cells(提供STR鉴定图谱)
HCRL-ATO10 Cells(提供STR鉴定图谱)
AMC-HN-8 Cells;背景说明:喉癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BEL7405细胞、HMSC细胞、SKMES-1细胞
SU86-86 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H-4细胞、S3 HeLa细胞、UPCI:SCC090细胞
SKO-007 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HCC1833细胞、NBL-5细胞、HKBML细胞
NCI-H2066 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:Panc_08_13细胞、Hs606T细胞、3T3细胞
HUCEC Cells;背景说明:宫颈;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RKO细胞、SUD-4细胞、HT-29/CX-1细胞
LC-1Sq Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH2172细胞、Hs 281.T细胞、Ramos G6.C10细胞
BT483 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SN-12C细胞、RPMI8226/S细胞、NCI-H1648细胞
BJA-B-1 Cells;背景说明:Burkitt's淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:GES1细胞、OVCAR-10细胞、SLMT-1细胞
H740 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Neuro2a细胞、SUM-190细胞、IOSE80细胞
Anip[973] Cells;背景说明:肺腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:B16BL6细胞、WTRL1细胞、293-EBNA细胞
LC-1 sq Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CEM/C1细胞、NCI-H1618细胞、Bac1 2F5细胞
LCD Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-SNU-520细胞、ISHI细胞、GM12878细胞
HEB Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:KMS26细胞、H-847细胞、EB1细胞
293-EBNA Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:10传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Calf Pulmonary Artery Endothelial细胞、NRK49F细胞、CBRH-7919细胞
RGC5 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:HeLa.S3细胞、L- cell细胞、SW839细胞
SGC-996 Cells(提供STR鉴定图谱)
JB-6 Cl 30 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:U-343 MG细胞、EC-109细胞、V79细胞
NS-1-Ag4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H661细胞、Tb1Lu细胞、OUMS27细胞
H510 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;每周换液2次。;生长特性:混合生长;形态特性:上皮细胞样;相关产品有:OCI-LY-1细胞、BEL7404细胞、IMR-90细胞
KYSE 270 Cells;背景说明:详见相关文献介绍;传代方法:1:5传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HOP92细胞、Hs739T细胞、G-361细胞
CAKI 2 Cells;背景说明:该细胞源自一位69岁白人男性的初期肾腺癌组织;传代方法:1:3—1:6传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:RPMI-6666细胞、BNL 1MEA.7R.1细胞、CHL/IU细胞
HEK/EBNA Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:10传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:K 562细胞、HT3细胞、NCI-H1693细胞
NCI-H2347人肺癌细胞代次低|培养基|送STR图谱
BC-024 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MGH-UI细胞、SUM 159细胞、MV-1-Lu细胞
RT4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:多角 ;相关产品有:U-118-MG细胞、OLN 93细胞、SVOG细胞
H2591 Cells;背景说明:上皮样间皮瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hep G2-Luc细胞、SKOV-3细胞、CEMC7细胞
Institute for Medical Research-32 Cells;背景说明:该细胞是1967年4月由NicholsWW,LeeJ和DwightS建立,来源于一名13月龄白人男婴腹部肿块,临床诊断为神经母细胞瘤,伴有极少部位的类器官样分化。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:存在两种细胞类型,小的神经母细胞样细胞和大的透明成纤维样细胞;相关产品有:MDA-415细胞、CORL26细胞、PC-3M-1E8细胞
IAR 20 Cells;背景说明:肝; BDVI;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NG-108-15细胞、HIBEC细胞、RINm-5F细胞
C2BBe 1 Cells;背景说明:详见相关文献介绍;传代方法:1:6—1:10传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:KYSE-510细胞、MRC5细胞、NCI-H125细胞
HBVSMC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:UT7细胞、Hs 870.T细胞、HC-11细胞
DHL-16 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MPASMC细胞、KYSE50细胞、OV1063细胞
BayGenomics ES cell line RRO218 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHB300 Cells(提供STR鉴定图谱)
H6-2.5E8 Cells(提供STR鉴定图谱)
PCH42-58 Cells(提供STR鉴定图谱)
ARL-16 Cells(提供STR鉴定图谱)
HPS0059 Cells(提供STR鉴定图谱)
" "PubMed=10987304
Girard L., Zochbauer-Muller S., Virmani A.K., Gazdar A.F., Minna J.D.
Genome-wide allelotyping of lung cancer identifies new regions of allelic loss, differences between small cell lung cancer and non-small cell lung cancer, and loci clustering.
Cancer Res. 60:4894-4906(2000)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=16187286; DOI=10.1002/ijc.21491
Garnis C., Lockwood W.W., Vucic E., Ge Y., Girard L., Minna J.D., Gazdar A.F., Lam S., MacAulay C., Lam W.L.
High resolution analysis of non-small cell lung cancer cell lines by whole genome tiling path array CGH.
Int. J. Cancer 118:1556-1564(2006)
PubMed=18083107; DOI=10.1016/j.cell.2007.11.025
Rikova K., Guo A.-L., Zeng Q.-F., Possemato A., Yu J., Haack H., Nardone J., Lee K., Reeves C., Li Y., Hu Y.-R., Tan Z.-P., Stokes M.P., Sullivan L., Mitchell J., Wetzel R., MacNeill J., Ren J.-M., Yuan J., Bakalarski C.E., Villen J., Kornhauser J.M., Smith B., Li D.-Q., Zhou X.-M., Gygi S.P., Gu T.-L., Polakiewicz R.D., Rush J., Comb M.J.
Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer.
Cell 131:1190-1203(2007)
PubMed=19472407; DOI=10.1002/humu.21028; PMCID=PMC2900846
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=24700732; DOI=10.1002/humu.22556; PMCID=PMC4451114
Leroy B., Girard L., Hollestelle A., Minna J.D., Gazdar A.F., Soussi T.
Analysis of TP53 mutation status in human cancer cell lines: a reassessment.
Hum. Mutat. 35:756-765(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"