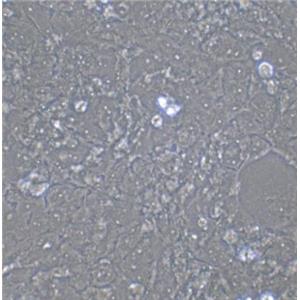
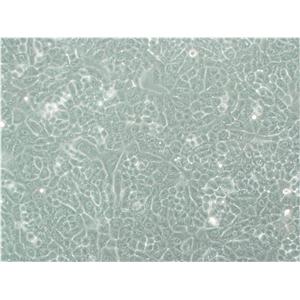
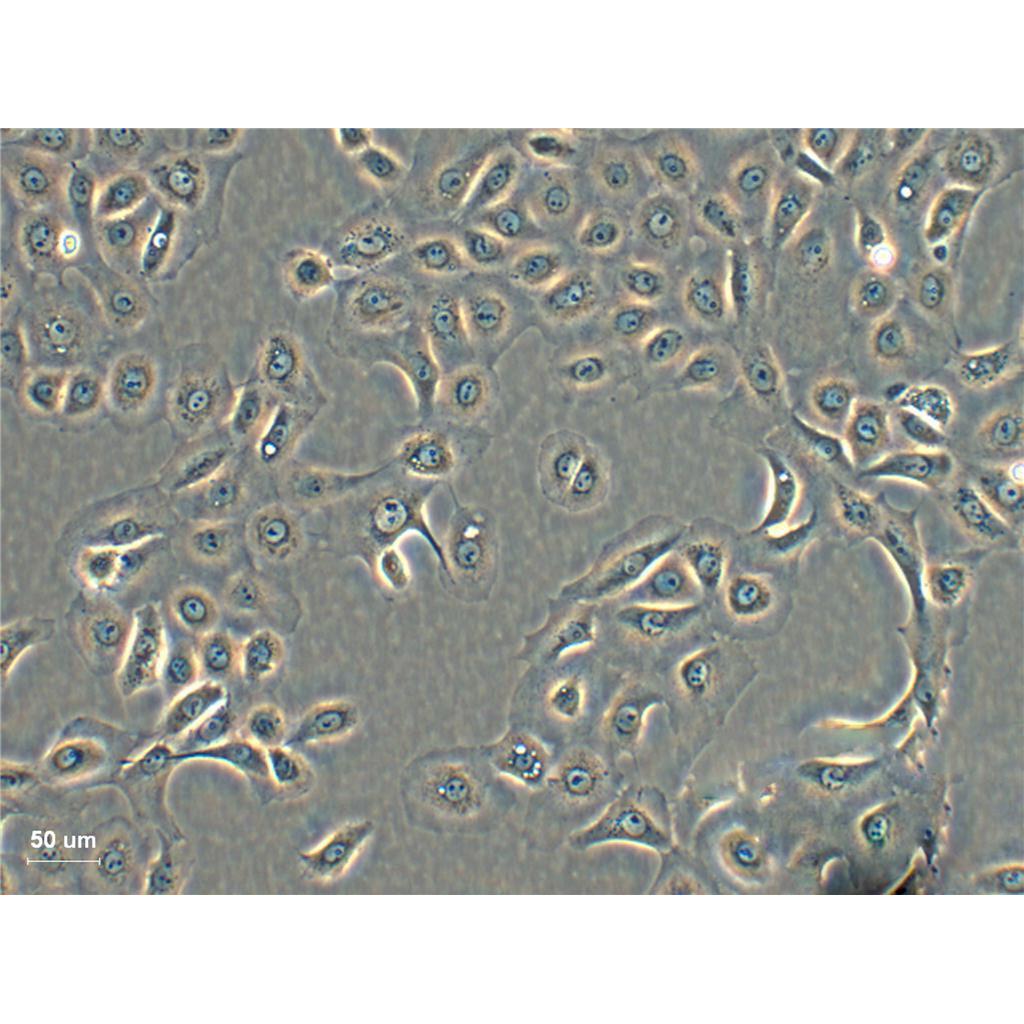
"NCI-H441人肺腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
常见细胞贴壁较弱原因:在遇到运输低温及震荡、室温静置太长时间、添加的培养基或其他试剂过冷、密度较高、聚集未吹散、加液吹打到细胞面等情况时会出现明显的成片脱落现象,此时若脱落现象不严重应尽快放回培养基继续培养,若呈大片脱落的情况时需要收集细胞重新消化吹散并接种;建议使用经过包被或者高贴壁培养瓶培养细胞,尽量避免接触低温或密度过高。
换液周期:每周2-3次
Wistar Institute, Susan Hayflick Cells;背景说明:最初以为这株细胞的起源是正常羊膜,但随后通过同工酶分析、HeLa标记染色体和DNA指纹法分析,证实该细胞是HeLa细胞污染的;角蛋白阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长 ;形态特性:上皮细胞样;相关产品有:HEK293-EBNA1细胞、RPMI-1846细胞、JROECL 21细胞
LC-2-Ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKRC 39细胞、HLFa细胞、WB-F344细胞
BERH-2 Cells;背景说明:肝癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MM1-S细胞、HL-7702细胞、Roswell Park Memorial Institute 2650细胞
NCI-H441人肺腺癌细胞代次低|培养基|送STR图谱
背景信息:NCI-H441建系于1982年(A.F.Gazdar,etal.)。该细胞分离自一名肺腺癌患者的心包。该细胞能在半固体琼脂糖中形成克隆,并能表达肺泡表面活性蛋白A。该细胞在有血清培养基中倍增时间为58小时,在无血清培养基中倍增时间为99-138小时。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
NCI-H441人肺腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
EMT-6 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CTLL2细胞、MCM细胞、GA-10细胞
Wistar Institute, Susan Hayflick Cells;背景说明:最初以为这株细胞的起源是正常羊膜,但随后通过同工酶分析、HeLa标记染色体和DNA指纹法分析,证实该细胞是HeLa细胞污染的;角蛋白阳性。;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长 ;形态特性:上皮细胞样;相关产品有:HEK293-EBNA1细胞、RPMI-1846细胞、JROECL 21细胞
LM1 Cells;背景说明:肾癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDA231-LM2-4175细胞、TCC Sup细胞、149PT细胞
2008 Cells;背景说明:宫颈鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:T241细胞、1301细胞、SK-Col-1细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞传代培养实验:体外培养的原代细胞或细胞株要在体外持续地培养就必须传代,以便获得稳定的细胞株或得到大量的同种细胞,并维持细胞种的延续。培养的细胞形成单层汇合以后,由于密度过大生存空间不足而引起营养枯竭,将培养的细胞分散,从容器中取出,以1:2或1:3以上的比率转移到另外的容器中进行培养,即为传代培养;细胞“一代”指从细胞接种到分离再培养的一段期间,与细胞世代或倍增不同。在一代中,细胞培增3~6次。细胞传代后,一般经过三个阶段:游离期、指数增生期和停止期。常用细胞分裂指数表示细胞增殖的旺盛程度,即细胞群的分裂相数/100个细胞。一般细胞分裂指数介于0.2%~0.5%,肿瘤细胞可达3~5%;细胞接种2~3天分裂增殖旺盛,是活力ZuiHAO时期,称指数增生期(对数生长期),适宜进行各种试验。实验步骤:1.将长成的培养细胞从二氧化碳培养箱中取出,在超净工作台中倒掉瓶内的培养,加入少许消化。(以面盖住细胞为宜),静置5~10分钟。2.在倒置镜下观察被消化的细胞,如果细胞变圆,相互之间不再连接成片,这时应立即在超净台中将消化倒掉,加入3~5ml新鲜培养,吹打,制成细胞悬。3.将细胞悬吸出2ml左右,加到另一个培养瓶中并向每个瓶中分别加3ml左右培养,盖HAO瓶塞,送回二氧化碳培养箱中,继续进行培养。一般情况,传代后的细胞在2小时左右就能附着在培养瓶壁上,2~4天就可在瓶内形成单层,需要再次进行传代。
HIT T15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SuDHL 8细胞、DH82细胞、TCMK-1细胞
Okayama University Medical School-27 Cells;背景说明:软骨肉瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:EHEB细胞、HT 1080.T细胞、2008细胞
Line 522 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H4IIEC3细胞、F442-A细胞、hMSC-BM细胞
SK MEL-28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:Mv 1 Lu细胞、SK-ChA1细胞、RGCs细胞
SKOV-433 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H1703细胞、Hs 729.T细胞、MC-26细胞
Hs-281-T Cells;背景说明:详见相关文献介绍;传代方法:1:2传代,每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NPC-TW01细胞、OVCA432_Bast细胞、HT 1080细胞
McA-RH 8994 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:A-375细胞、Baby Hamster Kidney-21细胞、SCL II细胞
IFRS1 Cells;背景说明:雪旺 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU-423细胞、GM06141细胞、TE13细胞
AZ521 Cells;背景说明:详见相关文献介绍;传代方法:1:4传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:3T6 Swiss Albino细胞、X63Ag8-653细胞、Uhth-74细胞
NCIH1395 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;5-6天传代一次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:C2-C12细胞、MH-S细胞、BJAB-1细胞
NCI-H295R Cells;背景说明:肾上腺皮质癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OCI-LY-8细胞、MIA-PaCa-2细胞、NCI-H1954细胞
CAKI1 Cells;背景说明:该细胞超微结构中包含许多微绒毛、少许微丝、许多小线粒体、发达的高尔基休和内质网、许多脂滴和多层体、次级溶酶体,没有发现病毒颗粒。;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Anip 973细胞、159 PT细胞、HTR8svn细胞
MDA MB 134VI Cells;背景说明:该细胞1973年由R. Cailleau建系,源自74岁乳腺导管癌女性患者的胸腔积液,细胞生长缓慢,松散贴壁,生长过程中会脱落到培养基,不会汇合,过表达FGF受体;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:KYSE410细胞、DHL-4细胞、PK-136细胞
UO-31 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Human Kidney-2细胞、PC2细胞、MC-3T3细胞
BCP-1 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在2×105/ml-1×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:P3X63Ag8-6-5-3细胞、High-5细胞、H-810细胞
H-2452 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:S-16细胞、hOMF细胞、BCaP 37细胞
RS4;11 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:KYSE 150 KYSE150 Kyse150 KY150
细胞、Colo741细胞、D10G41细胞
184A1 Cells(提供STR鉴定图谱)
Abcam HeLa LAMTOR2 KO Cells(提供STR鉴定图谱)
AG23059 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRF532 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG748 Cells(提供STR鉴定图谱)
C0808 Cells(提供STR鉴定图谱)
CW60408 Cells(提供STR鉴定图谱)
DD1872 Cells(提供STR鉴定图谱)
GM02018 Cells(提供STR鉴定图谱)
293-F Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;悬浮生长;形态特性:上皮细胞样;相关产品有:PC3M-IE8细胞、JIII细胞、C8161细胞
NCI-H441人肺腺癌细胞代次低|培养基|送STR图谱
HEK293-H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKNMC细胞、OCI AML4细胞、U87MG细胞
OCI-AML3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HCC0078细胞、UPCI:SCC90细胞、P3X63Ag8.653细胞
MV-1-Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BIU-87/Adr细胞、NOZC-1细胞、T 98 G细胞
H-187 Cells;背景说明:经典小细胞肺癌;胸腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs27F细胞、MDA-MB468细胞、JTC-39细胞
COLO-320-HSR Cells;背景说明:该细胞1984年建系,源自一位33岁患有大肠腺癌男性经5-fu治疗后的腹水。;传代方法:1:2传代。3天内可长满。;生长特性:半贴壁生长;形态特性:详见产品说明书;相关产品有:OVCA 433细胞、TERT-RPE1细胞、RPTEC/TERT1细胞
4.9.2 Cells(提供STR鉴定图谱)
HuT-102 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:圆形;淋巴母细胞样;相关产品有:102PT细胞、SK-UT-1细胞、P3-X63Ag8细胞
HCC-1833 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Menschliche Und Tierische Zellkulture-1细胞、Panc 5.04细胞、HOCF细胞
RM1 Cells;背景说明:前列腺癌;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH208细胞、RAMSCs细胞、CFPAC-1细胞
SK LU 1 Cells;背景说明:该细胞系源于一位60岁的白人女性患者的肺腺癌组织。;传代方法:1:2传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:EMT6细胞、LA-N-6(OAN)细胞、NCIH1836细胞
NCI-H1648 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CT26-clone 25细胞、UPCI:SCC154细胞、MSTO-211H细胞
Hep-G2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Emory University-2细胞、FK81细胞、CCC-HSF-1细胞
HCC-1954BL Cells;背景说明:外周血B淋巴细胞;EBV转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Walker256-TC细胞、WSUDLCL2细胞、MEF细胞
NCI660 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:上皮细胞;相关产品有:87 MG细胞、NCI-H23细胞、MV 4;11细胞
GM22143 Cells(提供STR鉴定图谱)
HAP1 MEF2B (-) 1 Cells(提供STR鉴定图谱)
C3H/10T1/2 CL8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MT4细胞、NUGC3细胞、MCF.10A细胞
H128 Cells;背景说明:小细胞肺癌;胸腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Caco2BBe细胞、MBVP细胞、MGH-U3 (RN)细胞
Hs822T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BL6细胞、HR-1细胞、HLE细胞
BALL-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:HepG2细胞、SACC-LM细胞、LCLC-103H细胞
MFC Cells;背景说明:胃癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MC3T3-L1细胞、C-Li-7细胞、Stanford University-Diffuse Histiocytic Lymphoma-2细胞
Tokyo Medical and Dental university 8 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CEK细胞、HB 611细胞、FLC-7细胞
H-1436 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:PNT1/A细胞、SUM-52-PE细胞、Hca-F细胞
Ramos 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HBE 135-E6/E7细胞、SK-N-BE-2c细胞、Sc-1细胞
HPSI0816i-hizl_3 Cells(提供STR鉴定图谱)
KB8594 Cells(提供STR鉴定图谱)
MeWo-LC1 Cells(提供STR鉴定图谱)
NIH 3T3 Duo Parental Cells(提供STR鉴定图谱)
Ramos/NPC Cells(提供STR鉴定图谱)
TOB0058 Cells(提供STR鉴定图谱)
UFERSA-SBA-1 Cells(提供STR鉴定图谱)
HAP1 SP100 (-) 1 Cells(提供STR鉴定图谱)
769P Cells;背景说明:该细胞系1975年建系,源自一位63岁白人女性的初期透明细胞腺癌组织,细胞呈圆形且边界不清,核浆比大,有微绒毛及桥粒。该细胞可在软琼脂上生长。 ;传代方法:1:4—1:12传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:AHH1细胞、AU-565细胞、H-1770细胞
293-EBNA1 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:10传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NS-1-Ag4-1细胞、RAW264.7细胞、Blotchy fibroblast-11细胞
U251n Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:IM-9细胞、NCIH748细胞、H-1672细胞
SUPB15 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SN12C-PM6细胞、HRPEpiC细胞、A 2058细胞
SKG IIIa Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HS578T细胞、NIH:OVCAR-3细胞、WEHI 3细胞
SKG IIIa Cells;背景说明:详见相关文献介绍;传代方法:2x10^4 cells/ml;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HS578T细胞、NIH:OVCAR-3细胞、WEHI 3细胞
HO8910/PM Cells;背景说明:高转移卵巢癌 Cells;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Bac12F5细胞、U-118-MG细胞、MUS-M1细胞
HLE Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PLC细胞、D10G41细胞、NB4细胞
OV 2008 Cells;背景说明:宫颈鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:FOXNY细胞、HFF-1细胞、NCI-H78细胞
NFS60 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HUT-125细胞、PLC PRF 5细胞、P1-Raji细胞
SKCO-1 Cells;背景说明:该细胞来源于结直肠病人的转移性腹水。;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:FAT细胞、EU-3细胞、293GP细胞
SK_N_BE2C Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MSTO-211 H细胞、NCI-H865细胞、NCIH2126细胞
MM1 Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MV 4;11细胞、Tregs细胞、NIH-3T3细胞
H283 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮细胞的多细胞聚集体,和一些贴壁 Cells;形态特性:上皮细胞;相关产品有:U266 Bl细胞、beta TC6细胞、Gerner 7666细胞
22Rv-1 Cells;背景说明:22RV1是来自异种移植(在阉割引起前列腺癌衰退又在其父亲的雄性激素信赖型CWR22嫁接后复发的小鼠中连续传代)的人前列腺癌上皮细胞系。此细胞系表达前列腺特异抗原。二羟基睾丸脂酮轻微刺激细胞生长,经westernblot检测溶解产物与抗雄性激素受体抗体起免疫反应。EGF刺激细胞生长,但TGFβ-1不能抑制细胞生长。该细胞在裸鼠中成瘤。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SGC7901细胞、HCC-70细胞、Monomac-1细胞
STAN041i-122-1 Cells(提供STR鉴定图谱)
Kit225 K6 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:2780细胞、PA12细胞、NCI-H508细胞
Line 697 Cells;背景说明:B淋巴细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HUT 226细胞、L-cells细胞、HeLa-S3细胞
OC-316 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ZR75_1细胞、HT-115细胞、alpha-TC1.6细胞
NCIH889 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮细胞;相关产品有:LS180细胞、SK-HEP-1细胞、FHL 124细胞
MDA175 Cells;背景说明:该细胞源自一位54岁患有乳腺导管癌白人女性的胸腔积液。;传代方法:1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:SK-MEL28细胞、HN6细胞、MDA-MB-435细胞
HFF-1 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:7传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:MHCC 97-L细胞、McARH7777细胞、HCC827细胞
NCI-H441人肺腺癌细胞代次低|培养基|送STR图谱
Swiss-3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:H-1688细胞、Capan2细胞、HCC1395细胞
Hep 3B Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Factor Dependent Continuous-Paterson 1细胞、SNGM细胞、HEK 293 c18细胞
L-WRN Cells;背景说明:皮下结缔组织;自发永生;雄性;C3H/An;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LC-1细胞、NCIH1836细胞、CW-2细胞
RWPE2 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Stanford University-Diffuse Histiocytic Lymphoma-8细胞、U87细胞、KE 37细胞
KYSE0030 Cells;背景说明:来源于一位64岁,患有高分化的中段食管鳞癌的男性患者。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:hOMF细胞、TE-8细胞、TE-9细胞
Hs683 Cells;背景说明:该细胞源自76岁白人男性的左颞叶侧胶质瘤组织,有微绒毛,无桥粒。 ;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:293-F细胞、A375SM细胞、LuCL4细胞
UM-UC-14 Cells;背景说明:肾癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:EAC-E2G8细胞、SiHa细胞、LLC-PK(1)细胞
HLE-B3 Cells;背景说明:晶状体;Ad12-SV40转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MS-751细胞、CHO-Lec1细胞、HMEL细胞
BayGenomics ES cell line CSH681 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRZ642 Cells(提供STR鉴定图谱)
BR1S384.5 Cells(提供STR鉴定图谱)
L-DR4-Dw15 Cells(提供STR鉴定图谱)
PM-81 Cells(提供STR鉴定图谱)
R118 Cells(提供STR鉴定图谱)
" "PubMed=8806092; DOI=10.1002/jcb.240630505
Phelps R.M., Johnson B.E., Ihde D.C., Gazdar A.F., Carbone D.P., McClintock P.R., Linnoila R.I., Matthews M.J., Bunn P.A. Jr., Carney D.N., Minna J.D., Mulshine J.L.
NCI-Navy Medical Oncology Branch cell line data base.
J. Cell. Biochem. Suppl. 24:32-91(1996)
PubMed=10536175; DOI=10.3892/ijo.15.5.927
Fujita T., Kiyama M., Tomizawa Y., Kohno T., Yokota J.
Comprehensive analysis of p53 gene mutation characteristics in lung carcinoma with special reference to histological subtypes.
Int. J. Oncol. 15:927-934(1999)
PubMed=11005564; DOI=10.1038/sj.neo.7900094; PMCID=PMC1550293
Kohno T., Sato T., Takakura S., Takei K., Inoue K., Nishioka M., Yokota J.
Mutation and expression of the DCC gene in human lung cancer.
Neoplasia 2:300-305(2000)
PubMed=11030152; DOI=10.1038/sj.onc.1203815
Modi S., Kubo A., Oie H.K., Coxon A.B., Rehmatulla A., Kaye F.J.
Protein expression of the RB-related gene family and SV40 large T antigen in mesothelioma and lung cancer.
Oncogene 19:4632-4639(2000)
PubMed=18083107; DOI=10.1016/j.cell.2007.11.025
Rikova K., Guo A.-L., Zeng Q.-F., Possemato A., Yu J., Haack H., Nardone J., Lee K., Reeves C., Li Y., Hu Y.-R., Tan Z.-P., Stokes M.P., Sullivan L., Mitchell J., Wetzel R., MacNeill J., Ren J.-M., Yuan J., Bakalarski C.E., Villen J., Kornhauser J.M., Smith B., Li D.-Q., Zhou X.-M., Gygi S.P., Gu T.-L., Polakiewicz R.D., Rush J., Comb M.J.
Global survey of phosphotyrosine signaling identifies oncogenic kinases in lung cancer.
Cell 131:1190-1203(2007)
PubMed=19153074; DOI=10.1093/hmg/ddp034
Medina P.P., Castillo S.D., Blanco S., Sanz-Garcia M., Largo C., Alvarez S., Yokota J., Gonzalez-Neira A., Benitez J., Clevers H.C., Cigudosa J.C., Lazo P.A., Sanchez-Cespedes M.
The SRY-HMG box gene, SOX4, is a target of gene amplification at chromosome 6p in lung cancer.
Hum. Mol. Genet. 18:1343-1352(2009)
PubMed=19472407; DOI=10.1002/humu.21028; PMCID=PMC2900846
Blanco R., Iwakawa R., Tang M.-Y., Kohno T., Angulo B., Pio R., Montuenga L.M., Minna J.D., Yokota J., Sanchez-Cespedes M.
A gene-alteration profile of human lung cancer cell lines.
Hum. Mutat. 30:1199-1206(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20557307; DOI=10.1111/j.1349-7006.2010.01622.x; PMCID=PMC11158680
Iwakawa R., Kohno T., Enari M., Kiyono T., Yokota J.
Prevalence of human papillomavirus 16/18/33 infection and p53 mutation in lung adenocarcinoma.
Cancer Sci. 101:1891-1896(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22961666; DOI=10.1158/2159-8290.CD-12-0112; PMCID=PMC3567922
Byers L.A., Wang J., Nilsson M.B., Fujimoto J., Saintigny P., Yordy J.S., Giri U., Peyton M., Fan Y.-H., Diao L.-X., Masrorpour F., Shen L., Liu W.-B., Duchemann B., Tumula P., Bhardwaj V., Welsh J., Weber S., Glisson B.S., Kalhor N., Wistuba I.I., Girard L., Lippman S.M., Mills G.B., Coombes K.R., Weinstein J.N., Minna J.D., Heymach J.V.
Proteomic profiling identifies dysregulated pathways in small cell lung cancer and novel therapeutic targets including PARP1.
Cancer Discov. 2:798-811(2012)
PubMed=24135919; DOI=10.1038/ncomms3617; PMCID=PMC4107456
Balbin O.A., Prensner J.R., Sahu A., Yocum A., Shankar S., Malik R., Fermin D., Dhanasekaran S.M., Chandler B., Thomas D., Beer D.G., Cao X.-H., Nesvizhskii A.I., Chinnaiyan A.M.
Reconstructing targetable pathways in lung cancer by integrating diverse omics data.
Nat. Commun. 4:2617.1-2617.13(2013)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26361996; DOI=10.1016/j.jprot.2015.09.003
Grundner-Culemann K., Dybowski J.N., Klammer M., Tebbe A., Schaab C., Daub H.
Comparative proteome analysis across non-small cell lung cancer cell lines.
J. Proteomics 130:1-10(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29444439; DOI=10.1016/j.celrep.2018.01.051; PMCID=PMC6343826
Yuan T.L., Amzallag A., Bagni R., Yi M., Afghani S., Burgan W., Fer N., Strathern L.A., Powell K., Smith B., Waters A.M., Drubin D.A., Thomson T., Liao R., Greninger P., Stein G.T., Murchie E., Cortez E., Egan R.K., Procter L., Bess M., Cheng K.T., Lee C.-S., Lee L.C., Fellmann C., Stephens R., Luo J., Lowe S.W., Benes C.H., McCormick F.
Differential effector engagement by oncogenic KRAS.
Cell Rep. 22:1889-1902(2018)
PubMed=29681454; DOI=10.1016/j.cell.2018.03.028; PMCID=PMC5935540
McMillan E.A., Ryu M.-J., Diep C.H., Mendiratta S., Clemenceau J.R., Vaden R.M., Kim J.-H., Motoyaji T., Covington K.R., Peyton M., Huffman K., Wu X.-F., Girard L., Sung Y., Chen P.-H., Mallipeddi P.L., Lee J.Y., Hanson J., Voruganti S., Yu Y., Park S., Sudderth J., DeSevo C., Muzny D.M., Doddapaneni H., Gazdar A.F., Gibbs R.A., Hwang T.H., Heymach J.V., Wistuba I.I., Coombes K.R., Williams N.S., Wheeler D.A., MacMillan J.B., DeBerardinis R.J., Roth M.G., Posner B.A., Minna J.D., Kim H.S., White M.A.
Chemistry-first approach for nomination of personalized treatment in lung cancer.
Cell 173:864-878.e29(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"