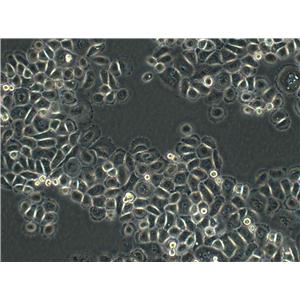
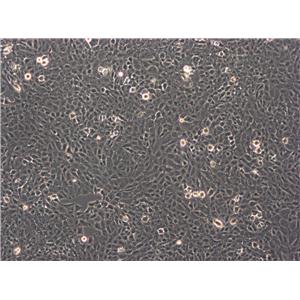
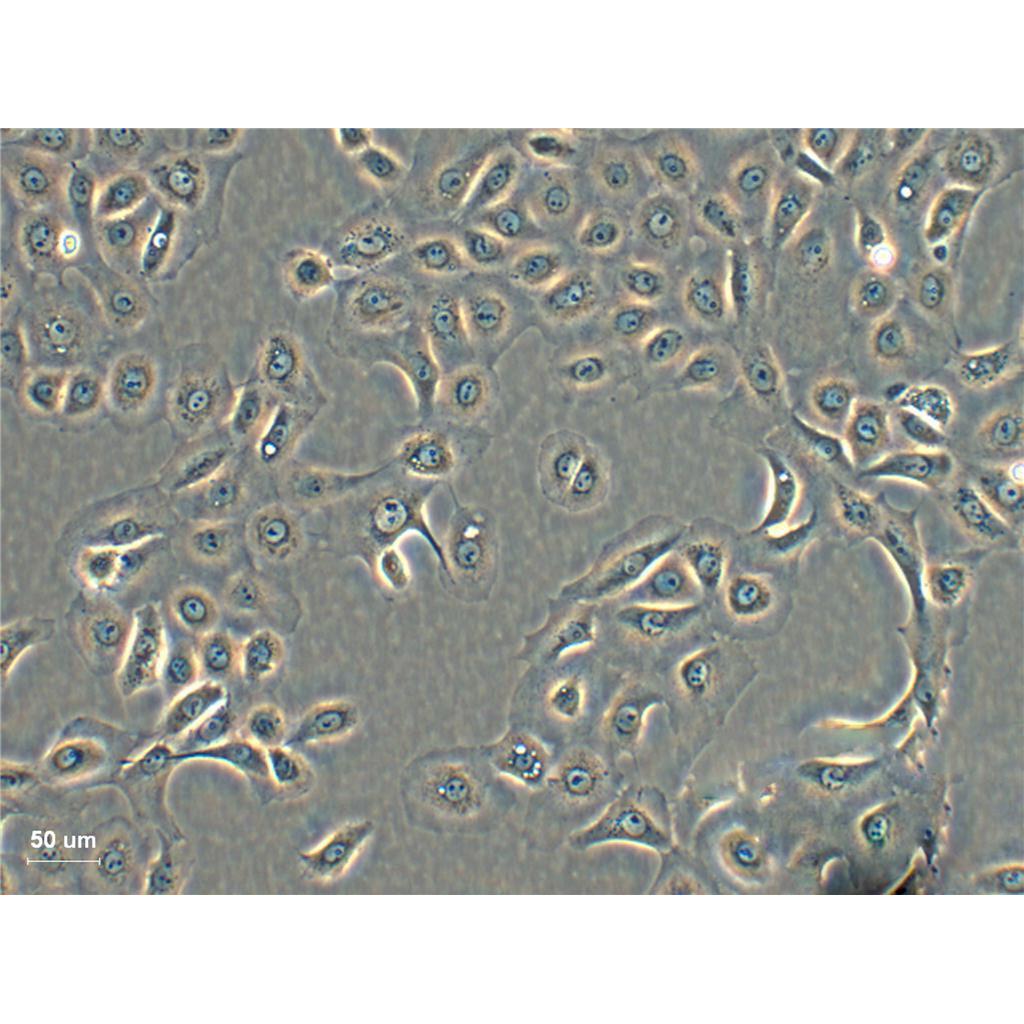
"SW48人结肠腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
换液周期:每周2-3次
H510A Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;每周换液2次。;生长特性:混合生长;形态特性:上皮细胞样;相关产品有:DHL-5细胞、PLB 985细胞、COLO-741细胞
OVCA 432 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:YES2细胞、NT-2细胞、HOC细胞
NCI-H2009 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CHP 212细胞、LS 174T细胞、WEHI3细胞
SW48人结肠腺癌细胞代次低|培养基|送STR图谱
背景信息:详见相关文献介绍
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
绝大部分细胞消化只要用胰酶润洗一遍即可:吸去胰酶后,残留的那些无法计算体积的附着在细胞表面的微量胰酶在37℃一般不到2min足够消化细胞(绝大部分1min不到)。对于这些细胞原则上不要用胰酶孵育细胞,连续这样传代,对细胞伤害很大。简单的程序是PBS润洗吸去,胰酶润洗吸去,然后37℃消化。什么算是消化好了呢?不需要把细胞全部消化成间隔分布很离散的单个圆形才算消化好了,一般你肉眼观察贴壁细胞层,只要能移动了,多半呈沙状移动,其实已经是可以了。一般能移动了,说明细胞与培养基质材料的附着已经消失了,细胞之间的附着也已经消失了,细胞已经独立分布了(虽然没有呈现很广的离散分布)。这个时候应该停止消化,不要等到看到镜下所有细胞都分离得非常好,间隙很大,才停止。细胞系在贴壁的过程中仍然会聚集,这个是贴壁培养的细胞,尤其是肿瘤细胞的一个特性,你可以尝试,准备100%的单个细胞悬液,贴壁后观察细胞,仍然是几个几个细胞聚集在一起。一些悬浮培养细胞也是如此,容易聚集,不要过几个小时就拿出来吹打成单细胞悬液。细胞只要能从基质上脱离下来,即使是成片的(比如Calu-3细胞),吹打不超过20次(一般10次即可),成小规模聚集(10个细胞左右)是正常的,不要再去延长消化时间,等待单细胞悬液出现。比较难消化的细胞:润洗方法5min还不能消化,以结肠癌细胞为例,比如:HCT15、LS411和KM12细胞,胰酶消化,一般10 cm培养皿,一次加入300ul-500ul就足够了。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。一些正常细胞也会有难消化的时候,比如tsDC细胞,用胰酶孵育,3min左右即可看到成片沙状移动。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
SW48人结肠腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
NRCC Cells;背景说明:肾透明细胞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Fu97细胞、8505C细胞、COLO320HSR细胞
Eca-109 Cells;背景说明:1973年建系,来自人食管中段鳞癌组织,小块法原代培养建系。BALB/c裸鼠移植成瘤。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:SKMEL1细胞、F-36P细胞、HCMEC(BL12-H)细胞
H4-II-E-C3 Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Mel-624细胞、CESS细胞、THEECs细胞
DU 145 Cells;背景说明:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,酸性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:Fetal Human Lens-124细胞、McA-RH 8994细胞、U118-MG细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
贴壁消化难题:1,先用PBS 把细胞洗两遍,使瓶内没有血清了,减少对胰酶的中和,然后用新配的0.25%的胰酶加入3ml左右,放在37度,然后可以在细胞有些消化下来时,拿着瓶口,运用手腕的力量轻轻震荡瓶内体,这样细胞很快就下来了,还不需要吹打,分散也均匀;2,成团、絮状:消化里加入eda可以减少细胞成团的现象,血清可以终止胰酶的作用,如果是进口血清的话也能终止eda的作用。用胰酶消化后胰酶可以倒掉,也可以不倒,直接加血清终止,如果消化中加入了eda的话,就要将消化倒干净,如果细胞贴壁要求不是很严格的话,一般不需要进行离心。鼻咽癌细胞的贴壁能力很强,用0.5%胰酶(含0.1%EDA)一般要消化12~15min。用PBS洗涤时要洗净残余的培养基,加入胰酶后在培养箱中消化(避免细胞室温下受损以及在此温度时胰酶活性Zui强)至细胞收缩变圆(可显微镜下观察)且有少许细胞脱落(有流下来的趋势),随后立即弃去胰酶(如果脱落的细胞很多且需要大量细胞实验,则不能弃去胰酶),加入培养基仔细吹打(不能用无血清培养基或者PBS替代,否则细胞聚集成团块或絮状)。一般我都离心一次弃去上清(去除残留的胰酶及漂浮的死细胞或细胞碎片);消化过度:马上用培养基中和,用吸管吹打细胞,收集全部的细胞到以无菌的离心管中800RPM 3分钟。弃上清,用全培重悬,换新的培养瓶继续培养,状态不HAO的细胞在培养的过程中会死亡脱落,在换的时候可以清除掉!
H196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:WM-2664细胞、HcaF细胞、GM01232细胞
U343 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HEK AD293细胞、FO [Mouse myeloma]细胞、SKNBE(2)细胞
MDAPCa-2b Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:TOV-112D细胞、U-373MG细胞、HS-683细胞
P3/NS1/1-Ag4.1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H2444细胞、IR 983F细胞、LC1细胞
PC3-M Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Hs 611.T细胞、H-647细胞、HO-8910细胞
LS 174T Cells;背景说明:LS 174T是LS 180 (ATCC CL 187)结肠腺癌细胞株的胰蛋白酶化变种。 它比亲本更易传代,象LS 180一样生成大量的癌胚抗原(CEA)。 电镜研究表明有丰富的微丝和细胞质粘液素液泡。 直肠抗原3阳性。 p53抗原表达阴性,但mRNA表达阳性。 与ATCC CL-187来源于同一个肿瘤。LS 174T细胞角蛋白染色阳性。 癌基因c-myc, N-myc, H-ras, N-ras, Myb, 和 fos的表达呈阳性。 癌基因k-ras和sis的表达未做检测。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HS 445T细胞、P3-NS1/1-Ag4-1细胞、L 540细胞
PCI-4B Cells;背景说明:喉鳞癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GDM-1细胞、NUGC4细胞、HUT125细胞
FaDu Cells;背景说明:1968年从一位印度下咽骨肿瘤患者的钻孔活组织切片中建立了FaDu细胞株。在建立的细胞株中发现细胞质中含有成束的细丝,并且细胞分界上的桥粒特别突出。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Farage细胞、D-407细胞、SK-MES-1细胞
GDM-1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:MCF7-GFP细胞、B16 melanoma F10细胞、UWB1289细胞
624 Cells;背景说明:黑色素瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MCMEC细胞、D10.G4.1细胞、CMT 64细胞
H1836 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:H-1581细胞、MUGCHOR1细胞、GM00215A细胞
Hk-2 Cells;背景说明:该细胞属源于正常肾的近曲小管细胞,通过导入HPV-16 E6/E7基因而获得永生化。将含有HPV-16 E6/E7基因的重组的逆转录病毒载体pLXSN 16 E6/E7转染外生包装细胞Psi-2,Psi-2细胞产生的病毒再去感染兼嗜性包装细胞系PA317,最后将PA317产生的病毒颗粒导入正常的肾皮质近曲小管细胞。尽管pLXSN 16 E6/E7中含有新霉素抗性,但未用G418筛选转导克隆。Southern和FISH分析显示HK-2细胞来源于单克隆。PCR检测证实HK-2细胞基因组中含有E6/E7基因。;传代方法:1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:B5537SKIN细胞、BT20细胞、PC-10细胞
Granta 519 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HET-1A细胞、J 774A.1细胞、Central Adrenergic TH-expressing a细胞
F56 [Human neoplasm] Cells;背景说明:F56是一株人的腺癌细胞系,可用于腺癌发生机制的研究,也可用于抗癌药物的筛选。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:CL-11细胞、PANC-02-03细胞、TE-85 clone 5细胞
McA-RH8994 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LAN1细胞、PL-45细胞、Mel624细胞
HSC5 Cells;背景说明:皮肤鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW 900细胞、AR42J细胞、H-510A细胞
RPTC Cells;背景说明:肾小管;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MES13细胞、293A细胞、M.D. Anderson-Prostate Cancer-2b细胞
Abcam HEK293T ARHGDIA KO 1 Cells(提供STR鉴定图谱)
AG09095 Cells(提供STR鉴定图谱)
BayGenomics ES cell line LRY015 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XC201 Cells(提供STR鉴定图谱)
Bre-40 Cells(提供STR鉴定图谱)
COLO 357-TRS4 Cells(提供STR鉴定图谱)
DA03895 Cells(提供STR鉴定图谱)
DA06471 Cells(提供STR鉴定图谱)
GM01023 Cells(提供STR鉴定图谱)
SW620 Cells;背景说明:SW620是从一个51岁男性白人组织中分离得到。由A.Leibovitz等从一个淋巴结建株。细胞系主要由无绒毛的小园球细胞和双极细胞组成。它仅合成少量癌胚抗原(CEA)且在裸鼠中有高度的致瘤性;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Granta 519细胞、MD Anderson-Metastatic Breast-436细胞、Vero-76细胞
SW48人结肠腺癌细胞代次低|培养基|送STR图谱
LC-2/ad Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE-85 clone 5细胞、SDBMSC细胞、AG 9细胞
HTR-8 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H2330细胞、MDA-157细胞、V 79细胞
NCI H548 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC 38细胞、MDAMB175VII细胞、AQ-Mel细胞
RCC-4 Cells;背景说明:肾透明细胞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A549细胞、KPL1细胞、COLO 320 HSR细胞
HCC 70 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Nthy-ori 3-1细胞、SK-MEL28细胞、AC16 [Human hybrid]细胞
30Q8A Cells(提供STR鉴定图谱)
SK-N-BE-2c Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-1915细胞、Human Foreskin Fibroblast细胞、ABC-1细胞
PANC-04-03 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MPC-83细胞、ADR-RES细胞、H-II-E-C3细胞
Factor Dependent Cell-Paterson 1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:HeLa S-3细胞、RPMI-8402细胞、G-Olig2细胞
HepG2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Centre Antoine Lacassagne-148细胞、Primary Liver Carcinoma/Poliomyelitis Research Foundation/5细胞、JKT1细胞
EST50 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:CCD-1095Sk细胞、GLRK 13细胞、SKES1细胞
Rat podocyte Cells;背景说明:肾;足 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WM 239-A细胞、HCC2185细胞、CHL-1细胞
EST50 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:CCD-1095Sk细胞、GLRK 13细胞、SKES1细胞
HAEC Cells;背景说明:主动脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Baby Hamster Kidney 21细胞、A-72细胞、DU4475细胞
GM12566 Cells(提供STR鉴定图谱)
HAP1 EPHB3 (-) Cells(提供STR鉴定图谱)
U-266 AR1 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:DI TNC1细胞、SU86.86细胞、H1819细胞
KM.12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HFLS细胞、GM02131细胞、SK HEP 01细胞
Farage OL Cells;背景说明:Farage细胞源于一名患有弥漫性大B细胞淋巴瘤(DLBCL)白人女性的活检淋巴组织,由HBen-Bassat建系。经IL-4处理,该细胞CD21,CD22,CD54和CD58表达上调,而CD21,CD22,andCD38表达下调。;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:F-36P细胞、SK-RC 52细胞、SK-MEL5细胞
MDA-MB-134-VI Cells;背景说明:该细胞1973年由R. Cailleau建系,源自74岁乳腺导管癌女性患者的胸腔积液,细胞生长缓慢,松散贴壁,生长过程中会脱落到培养基,不会汇合,过表达FGF受体;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:MDA-MB436细胞、TGW-nu细胞、RAOEC细胞
HeLa S3 Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCI-H82细胞、H676B细胞、Mink细胞
LA-N-5 Cells;背景说明:神经母细胞瘤;骨髓转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Merwin Plasma Cell tumor-11细胞、NIH:OVCAR-8细胞、LAD-2细胞
GM-346 Cells;背景说明:皮下结缔组织;自发永生;雄性;C3H/An;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OVHM细胞、KM H-2细胞、NBLS细胞
HCC-1569 Cells;背景说明:详见相关文献介绍;传代方法:1:4—1:6传代,每周换液2—3次;生长特性:混合生长;形态特性:上皮样;相关产品有:WSUDLCL2细胞、AN3 CA细胞、Hs606T细胞
HiPS-RIKEN-13A Cells(提供STR鉴定图谱)
iPS CWRU1 Cells(提供STR鉴定图谱)
MB-KG Cells(提供STR鉴定图谱)
ND10499 Cells(提供STR鉴定图谱)
PL [Sardinops] Cells(提供STR鉴定图谱)
tsJT60 Cells(提供STR鉴定图谱)
ULK1+ULK2 DKO MEF (SV40) Cells(提供STR鉴定图谱)
HAX1corrected-iPSC#1 Cells(提供STR鉴定图谱)
MCF/Adr Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BE(2)M-17细胞、BEL-7404细胞、CCC-ESF-1细胞
H-446 Cells;背景说明:该细胞是1982年由CarneyD和GazdarAF等从一位小细胞肺癌患者的胸腔积液中建立的。细胞的原始形态并不具有小细胞肺癌特征。这个细胞株是小细胞肺癌的生化和形态学上的变种,表达神经元特有的烯醇酶和脑型肌酸激酶同工酶;左旋多巴脱羧酶、蚕素、抗利尿激素、催产素或胃泌激素释放肽未达到可检测水平。与正常细胞相比,该细胞c-mycDNA序列扩增约20倍,RNA增加15倍。最初传代培养基用含有5%FBS的RPMI1640,另外添加10nM化可的松、0.005mg/ml胰岛素、0.01mg/ml转铁;传代方法:1:2传代;生长特性:贴壁/悬浮生长,混合;形态特性:上皮样;相关产品有:U251-MG细胞、Hs 888.T细胞、A9(Hamprecht)细胞
H-2106 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:Japanese Tissue Culture-39细胞、SBC-5细胞、C4 I细胞
HL-60 Clone 15 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在1×105-1×106/ml,每2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HHUA细胞、Roswell Park Memorial Institute 7666细胞、TE-6细胞
NCIH2141 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:Large Cell Lung Cancer-103H细胞、BJAB-1细胞、MESSA-DX5细胞
P3X63 Ag8.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Factor Dependent Cell-Paterson 1细胞、HHL-5细胞、MDA231-LM2-4175细胞
ID8/MOSEC Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:QGP1细胞、NCIN87细胞、ADR-RES细胞
H1666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长,松散;形态特性:上皮细胞;相关产品有:Hs 888.Lu细胞、H-2122细胞、LA795细胞
NRK49F Cells;背景说明:肾;成纤维细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SN4741细胞、GM04678细胞、3T3-L1 ad细胞
THP-1 Cells;背景说明:该细胞从一名1岁的患有急性单核细胞性白血病的男孩的外周血中分离建立。该细胞可以吞噬乳胶颗粒和激活的红细胞,细胞膜和胞浆内均没有免疫球蛋白,表达C3R和FcR;可受佛波酯TPA诱导向单核系方向分化;可作为转染宿主。;传代方法:维持细胞浓度在2-4×105-8×105/ml,勿超过1×106/ml;2-3天换液1次。;生长特性:悬浮生长;形态特性:单核细胞;相关产品有:OVCAR420细胞、P388-D1细胞、High Five细胞
P3-x63-Ag8 653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H23细胞、NCIH2126细胞、MV3细胞
P3-X63-Ag8-653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H250细胞、JKT1细胞、SJSA细胞
SK-ChA-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CHL细胞、PC-3细胞、TOV-112D细胞
H250 Cells;背景说明:小细胞肺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:VM-CUB1细胞、RERF-LC-MS细胞、NU-GC-2细胞
RCK-8 Cells;背景说明:弥漫大B淋巴瘤;腹腔转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:University of Arizona Cell Culture-893细胞、WC00079细胞、RPMI 7666细胞
SG237R Cells(提供STR鉴定图谱)
BL1339 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:L363细胞、OVISE细胞、H-358细胞
HCC-78 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:H358细胞、RPMC细胞、L-Wnt-3A细胞
NCIH526 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长 ;形态特性:上皮细胞;相关产品有:Panc8.13细胞、WERI Rb-1细胞、ATC241细胞
OCM-1A Cells;背景说明:脉络膜黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A.704细胞、Bronchial Epithelium transformed with Ad12-SV40 2B细胞、OCILY3细胞
Fukuoka University-Malignant mixed Mullerian Tumor-1 Cells;背景说明:子宫肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs600T细胞、NCI-SNU-182细胞、NRK clone 52E细胞
SW-1116 Cells;背景说明:CSAp阴性(CSAp-)。 结肠抗原3,阴性。 角蛋白免疫过氧化物酶染色阳性。 癌基因c-myc, K-ras, H-ras, myb, sis 和fos的表达呈阳性。 未检测到癌基因N-myc和N-ras的表达。 表达肿瘤特异的核基质蛋白CC-4,CC-5和CC-6。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CL1细胞、Transformed Human Liver Epithelial-3细胞、MODE-K细胞
SW48人结肠腺癌细胞代次低|培养基|送STR图谱
WM239A Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PC-3M-IE8细胞、OVCAR420细胞、TCMK1细胞
CORL51 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1299细胞、CF Pac1细胞、Hs746-T细胞
Intestinal Epithelioid Cell line No. 18 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hx-147细胞、H510A细胞、NIH:OVCAR3细胞
EA.Hy926 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NCI-H1770细胞、H-2029细胞、HEK293F细胞
D10 Cells;背景说明:T淋巴细胞;AKR/J;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HDLM2细胞、COR-L26细胞、Vx-2细胞
IOSE80 Cells;背景说明:卵巢;上皮细胞;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H69C细胞、LLC1细胞、Panc3_27细胞
Rin-M-5F Cells;背景说明:胰岛β细胞瘤;雄性;NEDH;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H748细胞、L M (TK-)细胞、MBT2细胞
SUM-149PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OsACL细胞、PANC.1细胞、NE1-E6E7细胞
BayGenomics ES cell line RRS010 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA263 Cells(提供STR鉴定图谱)
iBMK BIM+/- B Cells(提供STR鉴定图谱)
PCRP-NFIL3-2B5 Cells(提供STR鉴定图谱)
IAR 27F Cells(提供STR鉴定图谱)
HPSI0514i-suul_2 Cells(提供STR鉴定图谱)
" "PubMed=1000501
Leibovitz A., Stinson J.C., McCombs W.B. 3rd, McCoy C.E., Mazur K.C., Mabry N.D.
Classification of human colorectal adenocarcinoma cell lines.
Cancer Res. 36:4562-4569(1976)
PubMed=327080; DOI=10.1093/jnci/59.1.221
Fogh J., Fogh J.M., Orfeo T.
One hundred and twenty-seven cultured human tumor cell lines producing tumors in nude mice.
J. Natl. Cancer Inst. 59:221-226(1977)
PubMed=833871; DOI=10.1093/jnci/58.2.209
Fogh J., Wright W.C., Loveless J.D.
Absence of HeLa cell contamination in 169 cell lines derived from human tumors.
J. Natl. Cancer Inst. 58:209-214(1977)
PubMed=924690; DOI=10.1002/ijc.2910200505
Kerbel R.S., Pross H.F., Leibovitz A.
Analysis of established human carcinoma cell lines for lymphoreticular-associated membrane receptors.
Int. J. Cancer 20:673-679(1977)
PubMed=7459858
Rousset M., Zweibaum A., Fogh J.
Presence of glycogen and growth-related variations in 58 cultured human tumor cell lines of various tissue origins.
Cancer Res. 41:1165-1170(1981)
PubMed=7104989; DOI=10.1016/0165-4608(82)90076-0
Chen T.-R., Hay R.J., Macy M.L.
Karyotype consistency in human colorectal carcinoma cell lines established in vitro.
Cancer Genet. Cytogenet. 6:93-117(1982)
PubMed=6220172
Dracopoli N.C., Fogh J.
Polymorphic enzyme analysis of cultured human tumor cell lines.
J. Natl. Cancer Inst. 70:469-476(1983)
PubMed=6652615; DOI=10.1016/0165-4608(83)90092-4
Chen T.-R., Hay R.J., Macy M.L.
Intercellular karyotypic similarity in near-diploid cell lines of human tumor origins.
Cancer Genet. Cytogenet. 10:351-362(1983)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=8422623; DOI=10.1002/1097-0142(19930115)71:2<315::AID-CNCR2820710208>3.0.CO;2-B
Tibbetts L.M., Doremus C.M., Tzanakakis G.N., Vezeridis M.P.
Liver metastases with 10 human colon carcinoma cell lines in nude mice and association with carcinoembryonic antigen production.
Cancer 71:315-321(1993)
PubMed=8464898; DOI=10.1073/pnas.90.7.2842; PMCID=PMC46192
Browning M.J., Krausa P., Rowan A.J., Bicknell D.C., Bodmer J.G., Bodmer W.F.
Tissue typing the HLA-A locus from genomic DNA by sequence-specific PCR: comparison of HLA genotype and surface expression on colorectal tumor cell lines.
Proc. Natl. Acad. Sci. U.S.A. 90:2842-2845(1993)
PubMed=8197130; DOI=10.1073/pnas.91.11.4751; PMCID=PMC43866
Bicknell D.C., Rowan A.J., Bodmer W.F.
Beta 2-microglobulin gene mutations: a study of established colorectal cell lines and fresh tumors.
Proc. Natl. Acad. Sci. U.S.A. 91:4751-4755(1994)
PubMed=7651727
Kastrinakis W.V., Ramchurren N., Rieger K.M., Hess D.T., Loda M., Steele G., Summerhayes I.C.
Increased incidence of p53 mutations is associated with hepatic metastasis in colorectal neoplastic progression.
Oncogene 11:647-652(1995)
PubMed=9000572
Hoang J.-M., Cottu P.-H., Thuille B., Salmon R.J., Thomas G., Hamelin R.
BAT-26, an indicator of the replication error phenotype in colorectal cancers and cell lines.
Cancer Res. 57:300-303(1997)
PubMed=9294210; DOI=10.1073/pnas.94.19.10330; PMCID=PMC23362
Ilyas M., Tomlinson I.P.M., Rowan A.J., Pignatelli M., Bodmer W.F.
Beta-catenin mutations in cell lines established from human colorectal cancers.
Proc. Natl. Acad. Sci. U.S.A. 94:10330-10334(1997)
PubMed=9515795
Sparks A.B., Morin P.J., Vogelstein B., Kinzler K.W.
Mutational analysis of the APC/beta-catenin/Tcf pathway in colorectal cancer.
Cancer Res. 58:1130-1134(1998)
PubMed=10612807; DOI=10.1002/(SICI)1098-2264(200002)27:2<183::AID-GCC10>3.0.CO;2-P; PMCID=PMC4721570
Ghadimi B.M., Sackett D.L., Difilippantonio M.J., Schrock E., Neumann T., Jauho A., Auer G., Ried T.
Centrosome amplification and instability occurs exclusively in aneuploid, but not in diploid colorectal cancer cell lines, and correlates with numerical chromosomal aberrations.
Genes Chromosomes Cancer 27:183-190(2000)
PubMed=10737795; DOI=10.1073/pnas.97.7.3352; PMCID=PMC16243
Rowan A.J., Lamlum H., Ilyas M., Wheeler J.M.D., Straub J., Papadopoulou A., Bicknell D.C., Bodmer W.F., Tomlinson I.P.M.
APC mutations in sporadic colorectal tumors: a mutational 'hotspot' and interdependence of the 'two hits'.
Proc. Natl. Acad. Sci. U.S.A. 97:3352-3357(2000)
PubMed=11314036; DOI=10.1038/sj.onc.1204211
Forgacs E., Wren J.D., Kamibayashi C., Kondo M., Xu X.L., Markowitz S.D., Tomlinson G.E., Muller C.Y., Gazdar A.F., Garner H.R., Minna J.D.
Searching for microsatellite mutations in coding regions in lung, breast, ovarian and colorectal cancers.
Oncogene 20:1005-1009(2001)
PubMed=11526487; DOI=10.1038/sj.onc.1204611
Gayet J., Zhou X.-P., Duval A., Rolland S., Hoang J.-M., Cottu P.-H., Hamelin R.
Extensive characterization of genetic alterations in a series of human colorectal cancer cell lines.
Oncogene 20:5025-5032(2001)
PubMed=12615714
Hempen P.M., Zhang L., Bansal R.K., Iacobuzio-Donahue C.A., Murphy K.M., Maitra A., Vogelstein B., Whitehead R.H., Markowitz S.D., Willson J.K.V., Yeo C.J., Hruban R.H., Kern S.E.
Evidence of selection for clones having genetic inactivation of the activin A type II receptor (ACVR2) gene in gastrointestinal cancers.
Cancer Res. 63:994-999(2003)
PubMed=16418264; DOI=10.1073/pnas.0510146103; PMCID=PMC1327731
Liu Y., Bodmer W.F.
Analysis of p53 mutations and their expression in 56 colorectal cancer cell lines.
Proc. Natl. Acad. Sci. U.S.A. 103:976-981(2006)
PubMed=18258742; DOI=10.1073/pnas.0712176105; PMCID=PMC2268141
Emaduddin M., Bicknell D.C., Bodmer W.F., Feller S.M.
Cell growth, global phosphotyrosine elevation, and c-Met phosphorylation through Src family kinases in colorectal cancer cells.
Proc. Natl. Acad. Sci. U.S.A. 105:2358-2362(2008)
PubMed=19927377; DOI=10.1002/gcc.20730; PMCID=PMC2818350
Knutsen T., Padilla-Nash H.M., Wangsa D., Barenboim-Stapleton L., Camps J., McNeil N.E., Difilippantonio M.J., Ried T.
Definitive molecular cytogenetic characterization of 15 colorectal cancer cell lines.
Genes Chromosomes Cancer 49:204-223(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=20570890; DOI=10.1158/0008-5472.CAN-10-0192; PMCID=PMC2943514
Janakiraman M., Vakiani E., Zeng Z.-S., Pratilas C.A., Taylor B.S., Chitale D., Halilovic E., Wilson M., Huberman K., Ricarte Filho J.C.M., Persaud Y., Levine D.A., Fagin J.A., Jhanwar S.C., Mariadason J.M., Lash A., Ladanyi M., Saltz L.B., Heguy A., Paty P.B., Solit D.B.
Genomic and biological characterization of exon 4 KRAS mutations in human cancer.
Cancer Res. 70:5901-5911(2010)
PubMed=20606684; DOI=10.1038/sj.bjc.6605780; PMCID=PMC2920028
Bracht K., Nicholls A.M., Liu Y., Bodmer W.F.
5-fluorouracil response in a large panel of colorectal cancer cell lines is associated with mismatch repair deficiency.
Br. J. Cancer 103:340-346(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=24042735; DOI=10.1038/oncsis.2013.35; PMCID=PMC3816225
Ahmed D., Eide P.W., Eilertsen I.A., Danielsen S.A., Eknaes M., Hektoen M., Lind G.E., Lothe R.A.
Epigenetic and genetic features of 24 colon cancer cell lines.
Oncogenesis 2:e71.1-e71.8(2013)
PubMed=24755471; DOI=10.1158/0008-5472.CAN-14-0013
Mouradov D., Sloggett C., Jorissen R.N., Love C.G., Li S., Burgess A.W., Arango D., Strausberg R.L., Buchanan D., Wormald S., O'Connor L., Wilding J.L., Bicknell D.C., Tomlinson I.P.M., Bodmer W.F., Mariadason J.M., Sieber O.M.
Colorectal cancer cell lines are representative models of the main molecular subtypes of primary cancer.
Cancer Res. 74:3238-3247(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25926053; DOI=10.1038/ncomms8002
Medico E., Russo M., Picco G., Cancelliere C., Valtorta E., Corti G., Buscarino M., Isella C., Lamba S., Martinoglio B., Veronese S., Siena S., Sartore-Bianchi A., Beccuti M., Mottolese M., Linnebacher M., Cordero F., Di Nicolantonio F., Bardelli A.
The molecular landscape of colorectal cancer cell lines unveils clinically actionable kinase targets.
Nat. Commun. 6:7002.1-7002.10(2015)
PubMed=25944804; DOI=10.1158/1078-0432.CCR-14-2457
Bazzocco S., Dopeso H., Carton-Garcia F., Macaya I., Andretta E., Chionh F., Rodrigues P., Garrido M., Alazzouzi H., Nieto R., Sanchez A., Schwartz S. Jr., Bilic J., Mariadason J.M., Arango D.
Highly expressed genes in rapidly proliferating tumor cells as new targets for colorectal cancer treatment.
Clin. Cancer Res. 21:3695-3704(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26537799; DOI=10.1074/mcp.M115.051235; PMCID=PMC4762531
Holst S., Deuss A.J.M., van Pelt G.W., van Vliet S.J., Garcia-Vallejo J.J., Koeleman C.A.M., Deelder A.M., Mesker W.E., Tollenaar R.A.E.M., Rombouts Y., Wuhrer M.
N-glycosylation profiling of colorectal cancer cell lines reveals association of fucosylation with differentiation and caudal type homebox 1 (CDX1)/villin mRNA expression.
Mol. Cell. Proteomics 15:124-140(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28683746; DOI=10.1186/s12943-017-0691-y; PMCID=PMC5498998
Berg K.C.G., Eide P.W., Eilertsen I.A., Johannessen B., Bruun J., Danielsen S.A., Bjornslett M., Meza-Zepeda L.A., Eknaes M., Lind G.E., Myklebost O., Skotheim R.I., Sveen A., Lothe R.A.
Multi-omics of 34 colorectal cancer cell lines -- a resource for biomedical studies.
Mol. Cancer 16:116.1-116.16(2017)
PubMed=28854368; DOI=10.1016/j.celrep.2017.08.010; PMCID=PMC5583477
Roumeliotis T.I., Williams S.P., Goncalves E., Alsinet C., Del Castillo Velasco-Herrera M., Aben N., Ghavidel F.Z., Michaut M., Schubert M., Price S., Wright J.C., Yu L., Yang M., Dienstmann R., Guinney J.H., Beltrao P., Brazma A., Pardo M., Stegle O., Adams D.J., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Choudhary J.S.
Genomic determinants of protein abundance variation in colorectal cancer cells.
Cell Rep. 20:2201-2214(2017)
PubMed=29101300; DOI=10.15252/msb.20177701; PMCID=PMC5731344
Frejno M., Zenezini Chiozzi R., Wilhelm M., Koch H., Zheng R.-S., Klaeger S., Ruprecht B., Meng C., Kramer K., Jarzab A., Heinzlmeir S., Johnstone E., Domingo E., Kerr D.J., Jesinghaus M., Slotta-Huspenina J., Weichert W., Knapp S., Feller S.M., Kuster B.
Pharmacoproteomic characterisation of human colon and rectal cancer.
Mol. Syst. Biol. 13:951-951(2017)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"