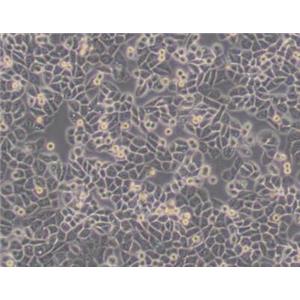
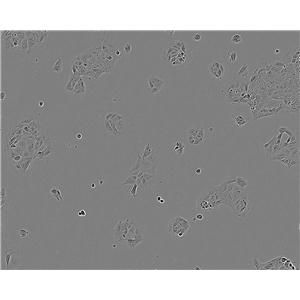
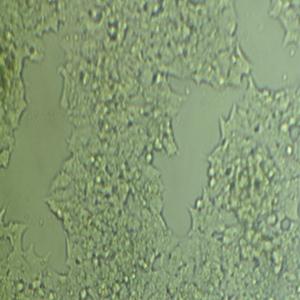
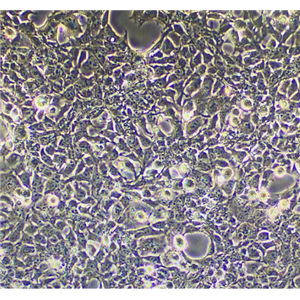
"MDA-MB-468人乳腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
细胞系的选择需要考虑到细胞系的功能特点、生长速率、铺板效率、生长条件和生长特征、克隆效率、培养方式等因素,如果您想高产量表达重组蛋白,您可以选择可以悬浮生长的快速生长细胞系。细胞培养的操作步骤主要包括传代、换液、冻存和复苏。这些步骤确保了细胞能够在实验室环境中长期存活并继续增殖。传代是将细胞从一个容器转移到另一个容器的过程,以扩大细胞数量;换液是为了清除代谢废物并补充新鲜培养基;冻存则是为了长期保存细胞,而复苏则是重新激活冷冻保存的细胞使其恢复正常生长。
换液周期:每周2-3次
MOLP2 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MDA231细胞、Panc2.03细胞、PTK1细胞
293 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NPA87细胞、HT-144细胞、A875细胞
SW 13 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCD18Co细胞、UMUC-14细胞、CAOV4细胞
MDA-MB-468人乳腺癌细胞代次低|培养基|送STR图谱
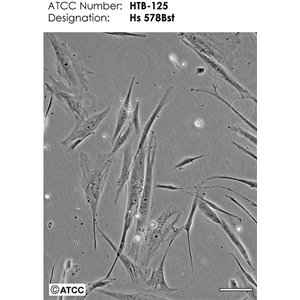
背景信息:该细胞是1977年由CailleauR等从一位患有转移性乳腺癌的51岁黑人女性的胸腔积中分离得到的。虽然供体组织的G6PD等位基因杂合,但此细胞株始终表现为G6表型。P53基因273位密码子存在G→A突变,从而导致Arg→His替代。每个细胞上存在1×106个EGF受体。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
MDA-MB-468人乳腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
ROS17/28 Cells;背景说明:骨肉瘤;ACI 9935;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Immortalized Human Hepatocytes细胞、MC57G细胞、IGROV-1细胞
OE-21 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Colon26细胞、Jurkat E6.1细胞、H250细胞
WM115F Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WIL2S细胞、KPL-1细胞、LLC PK1细胞
SNU-C2A Cells;背景说明:详见相关文献介绍;传代方法:每周两次换液;生长特性:松散附着、多单元的聚合;形态特性:上皮细胞样;相关产品有:AAV293细胞、SNU-354细胞、SNU-5细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞株(系)的使用,为医学研究和测试工作带来了大的方便。但细胞的传代是有限制的,长期连续传代的细胞,不仅消耗大量的人力和物力,而且细胞的生长与形态等会有一定退变或转化,因而细胞失去原有的遗传性,有时还会由于细胞污染而造成传代中断,种子丢失。因此,在实际工作中常需冻存一定数量的细胞,以备替换使用。细胞冷冻与复苏是细胞培养 室的常规工作和通用技术。目前,细胞冻存Zui常用的技术是冷冻保存法,主要采用加适量保护剂的缓慢冷冻法冻存细胞。细胞在不加任何保护剂的情况下直接冷冻,细胞内外的水分会很快形成冰晶,从而引起一系列不良反应。如细胞脱水使局部电解质浓度增GAO,pH值改变,部分蛋白质由于上述原因而变性,引起细胞内部空间结构紊乱,溶酶体膜由此遭到损伤而释放出溶酶体酶,使细胞内结构成分造成破坏,线粒体肿胀,功能丢失,并造成能量代谢障碍。胞膜上的类脂蛋白复合体也易破坏引起细胞膜通透性的改变,使细胞内容物丢失。如果细胞内冰晶形成较多,随冷冻温度的降低,冰晶体积膨胀造成细胞核DNA空间构型发生不可逆的损伤,而致细胞死亡。因此,细胞冷冻技术的关键是尽可能地减少细胞内水分,减少细胞内冰晶的形成。采用甘油或二甲基亚砜作保护剂,这两种物质分子量小,溶解度大,易穿透细胞,可以使冰点下降,提GAO细胞膜对水的通透性,且对细胞无明显毒性。慢速冷冻方法又可使细胞内的水分渗出细胞外,减少胞内形成冰结晶的机会,从而减少冰晶对细胞的损伤。
HPaSteC(HPSC) Cells;背景说明:胰腺星状 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C3H-10T1/2细胞、MyLa 2059细胞、HEL-92细胞
Sp 2817 Cells;背景说明:前列腺癌;左锁骨上淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPMI1846细胞、OUMS27细胞、NCI-BL6细胞
TE-7 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Human Intestinal Epithelial Cell-6细胞、MGH-UI细胞、H2291细胞
293H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SMC-1细胞、HEK (AD293)细胞、MSB-1细胞
U 937 Cells;背景说明:该细胞是由NilssonK实验室于1974年从一名37岁的患有恶性组织细胞性淋巴瘤的白人男性的胸水中分离建立的。1979年来的研究显示该细胞在人混合淋巴细胞培养物上清、佛波酯、VitD3、γ-IFN、TNF和维A酸的诱导下可以向终末单核细胞分化。该细胞不合成免疫球蛋白,EBV阴性;可产生溶菌酶、β-2-微球蛋白,受PMA刺激后可产生TNF-α;表达C3R;可作转染宿主;表达Fas,对TNF和抗Fas的抗体敏感。;传代方法:维持细胞浓度在1×105-2×106/ml;根据细胞浓度3-4换液1次。;生长特性:悬浮生长;形态特性:单核细胞;相关产品有:EAHY-926细胞、N1S1细胞、HBdSMC细胞
LUDLU1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OCILY3细胞、CT-26 WT细胞、K562细胞
SNU-638 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hu-P-T4细胞、VeroC1008细胞、Anip[973]细胞
OCI-Ly 10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:IEC 18细胞、HLE B-3细胞、WILL-2细胞
HTori:3 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KBM-7/Hap8细胞、H4-II-E细胞、SCC7细胞
MONO-MAC 6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H-2135细胞、P30/OHK细胞、EOC 20细胞
751-NA-15 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SMA-560细胞、SW 480细胞、KYSE 70细胞
MRASMC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Tohoku Hospital Pediatrics-1细胞、EC9706细胞、CCD-18Co细胞
SC Cells;背景说明:急性单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:DU145细胞、HT 115细胞、H87细胞
HOP-62 Cells;背景说明:肺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LK2细胞、HUTU80细胞、T24细胞
HCT 8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KNS42细胞、P-388D1细胞、WEHI 231细胞
MDA MB231 Cells;背景说明:MDA-MB-231来自患有转移乳腺腺癌的51岁女病人的胸水。在裸鼠和ALS处理的BALB/c小鼠中,它能形成低分化腺癌(III级)。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:GS-9L细胞、T-ALL1细胞、KMST-6细胞
PFSK-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GT38细胞、Tu-212细胞、Roswell Park Memorial Institute 8402细胞
Abcam HEK293T PTTG1 KO Cells(提供STR鉴定图谱)
AG12070 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRB053 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XE347 Cells(提供STR鉴定图谱)
BY00290 Cells(提供STR鉴定图谱)
CS28iALS-C9n2 Cells(提供STR鉴定图谱)
DA04419 Cells(提供STR鉴定图谱)
FD-1-iPSC Cells(提供STR鉴定图谱)
GM08278 Cells(提供STR鉴定图谱)
TE-14 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:A-20细胞、C33A细胞、HEC1A细胞
MDA-MB-468人乳腺癌细胞代次低|培养基|送STR图谱
D283MED Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮细胞的多细胞聚集体,和一些贴壁 Cells;形态特性:上皮细胞;相关产品有:Lu99A细胞、HEK293S细胞、DoTc2细胞
SK-MEL-28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:HSF细胞、AQ-Mel细胞、4-1st细胞
HPMEC Cells;背景说明:肺微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hela-mock细胞、GEO细胞、FAK+/+细胞
NCI-H1522 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H441细胞、GM03671细胞、SK-HEP-1细胞
JIMT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HECV细胞、OCILY19细胞、SUPB15细胞
B6ft-1 Cells(提供STR鉴定图谱)
MJ Cells;背景说明:详见相关文献介绍;传代方法:每周换液两次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:RN细胞、NCI-SNU-119细胞、Jurkat-FHCRC细胞
CCD-841CoN Cells;背景说明:结肠上皮细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HPF细胞、Tsup-1细胞、hTERT-RPE细胞
HLE B-3 Cells;背景说明:晶状体;Ad12-SV40转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:M.D. Anderson-Prostate Cancer-2b细胞、Ly1细胞、CZ-1细胞
WERI-Rb 1 Cells;背景说明:WERI-Rb-I细胞株是1974年R.M. McFall 和 T.W. Sery建立的两株人眼癌细胞系中的一株。 细胞能在Difco Bacto-Agar中存活但不形成克隆。 扫描电镜显示在表面囊泡,板状伪足和微绒毛在数量上和频率上的改变。 细胞分化研究,肿瘤治疗的动物模型和生化评价都涉及这株细胞。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:圆形细胞聚集成葡萄状;相关产品有:C4-2细胞、RK13细胞、Y3-Ag1,2,3细胞
C-4-I Cells;背景说明:宫颈鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Capan2细胞、beta-TC6细胞、Jijoye细胞
Pt K2 (NBL-5) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NP69SV40T细胞、Cor L88细胞、NCIH1650细胞
Pt K2 (NBL-5) Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NP69SV40T细胞、Cor L88细胞、NCIH1650细胞
NIH:OVCAR-5 Cells;背景说明:卵巢癌;腹水转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Ca9-22细胞、HCT-GEO细胞、Hs870T细胞
GM10225 Cells(提供STR鉴定图谱)
HAP1 C9orf72 (-) SMCR8 (-) 4 Cells(提供STR鉴定图谱)
H35 Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OV 2008细胞、6T-CEM细胞、OVCAR8细胞
MES-SA/Dx5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:8传代;每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞样 ;相关产品有:Glioma-261细胞、B16-F1细胞、HANK1细胞
L-6TG Cells;背景说明:肌母 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:P-2003细胞、A875细胞、GS-9L细胞
MX1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BXPC3细胞、ASH-3细胞、Lu-65细胞
KYSE510 Cells;背景说明:食管鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Eph4 1424细胞、AE1201细胞、Ramos 2G6.4C10细胞
LS-123 Cells;背景说明:详见相关文献介绍;传代方法:1:4—1:8传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs 739.T细胞、HEK293-F细胞、H2330细胞
RMa-bm Cells;背景说明:骨髓巨噬 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半悬浮;形态特性:详见产品说明书;相关产品有:CT26WT细胞、HEL-92.1.7细胞、HCC2218细胞
HFL-1 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:CCRF-SB细胞、RCC10RGB细胞、H196细胞
HG03649 Cells(提供STR鉴定图谱)
IEC-CF7 Cells(提供STR鉴定图谱)
LRo0022 Cells(提供STR鉴定图谱)
NCKDi004-A Cells(提供STR鉴定图谱)
PB32-4 Cells(提供STR鉴定图谱)
Ubigene HEK293 GSTP1 KO Cells(提供STR鉴定图谱)
WG1531 Cells(提供STR鉴定图谱)
HAP1 STAT3 (-) 1 Cells(提供STR鉴定图谱)
293 H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RGC5细胞、NCI-H2291细胞、H-125细胞
H-23 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:L 363细胞、SK-N-BE(2C)细胞、NCI-H187细胞
PZ-HPV-7 Cells;背景说明:前列腺上皮细胞;HPV18转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-SNU-216细胞、174xCEM.T2细胞、HEK-293FT细胞
H-1734 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:JM-1细胞、3T3NIH细胞、HBL-100细胞
2E8 Cells;背景说明:详见相关文献介绍;传代方法:2-4天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:HCT 8细胞、U-87细胞、SKUT1细胞
2E8 Cells;背景说明:详见相关文献介绍;传代方法:2-4天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:HCT 8细胞、U-87细胞、SKUT1细胞
HCT GEO Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ISHI细胞、Rainbow Trout Embryo细胞、T_T_细胞
MCF-7B Cells;背景说明:浸润性导管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jurkat E6.1细胞、TFK-1细胞、NCIH1703细胞
HOS-MNNG Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GB1细胞、HUC细胞、H187细胞
CAL-148 Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:253J-Bladder-V细胞、T-47-D细胞、COLO680N细胞
293S Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CL-34细胞、LC1sq细胞、RBL-2H3细胞
293S Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CL-34细胞、LC1sq细胞、RBL-2H3细胞
UMC11 Cells;背景说明:肺肿瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U-373-MG细胞、KRC/Y细胞、NTERA-2 cl.D1细胞
EFM-192A Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H524细胞、Anip 973细胞、Mv1Lu细胞
NCI-H-295 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KM932细胞、LN-229细胞、NCCIT细胞
SCA3.A8 Cells(提供STR鉴定图谱)
OCI-Ly19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:T84细胞、K-1735细胞、TNC-1B12B4细胞
COLO-824 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Med 341细胞、Rat Skin 1细胞、M1细胞
GM2219C Cells;背景说明:MOLT-4与MOLT-3来源于一名19岁的男性急性淋巴细胞性白血病的复发患者,该患者前期接受过多种药物联合化疗。MOLT-4细胞系为T淋巴细胞起源,p53基因的第248位密码子有一个G→A突变,不表达p53,不表达免疫球蛋白或EB病毒;可产生高水平的末端脱氧核糖转移酶;表达CD1(49%),CD2(35%),CD3A(26%)B(33%)C(34%),CD4(55%),CD5(72%),CD6(22%),CD7(77%)。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:AK细胞、HEK 293-EBNA细胞、Loucy细胞
H-1563 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCI-H2023细胞、MIN6细胞、NCIH1568细胞
Tn-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MHCC97H细胞、AG06814-M细胞、16-HBE细胞
HLEB3 Cells;背景说明:晶状体;Ad12-SV40转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:McA-RH 7777细胞、2780CP细胞、NRK-52E细胞
MDA-MB-468人乳腺癌细胞代次低|培养基|送STR图谱
Jiyoye Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:ABC-1细胞、LNCaP C4-2细胞、3AO细胞
NCI-H196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NIE-115细胞、Centre Antoine Lacassagne-12T细胞、L615细胞
Z138 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:15传代;每周2-3次。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:NCI-H520细胞、HeLa 229细胞、KATOIII细胞
T-ALL1 Cells;背景说明:该细胞源于一名复发T-ALL(急性T淋巴细胞性白血病)的儿童的外周血;具有很强的细胞毒性,体内体外实验中都能破坏肿瘤细胞;IL-2可使细胞更好地生长;α/β TCR阳性,γ/δ TCR阴性;可产生IFNγ、TNF-α和GM-CSF。;传代方法:维持细胞密度在4×105-1×106 cells/ml之间,2-3天换液1次 ;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:RS(4;11)细胞、CATH.a细胞、1.1B4细胞
Ly8 Cells;背景说明:弥漫大B淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Vx-2细胞、HR1K细胞、3T3-Swiss albino细胞
C8D1A Cells;背景说明:该永生化细胞系源自出生8天小鼠小脑组织,由B Pessac, D Trisler建立。该细胞具有小神经胶质细胞特征。该细胞为GFAP阳性细胞,除此之外,没有检测到其它神经胶质神经元或小神经胶质细胞的分子标记。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NCIH1930细胞、EFM-192A细胞、MCF7/WT细胞
WEHI-164 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:K7M2细胞、BCP1细胞、HuH7细胞
NCIH196 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:DU 145细胞、HEK-293-EBNA细胞、SK-NMC细胞
BayGenomics ES cell line CSD368 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRT115 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC281 Cells(提供STR鉴定图谱)
ISOS-1 Cells(提供STR鉴定图谱)
PCRP-STAT5B-1B10 Cells(提供STR鉴定图谱)
LO-HRP-15 Cells(提供STR鉴定图谱)
" "DOI=10.1016/B978-0-12-333530-2.50009-5
Leibovitz A.
Cell lines from human breast.
(In book chapter) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.161-184; Academic Press; New York; USA (1994)
PubMed=9671407; DOI=10.1038/sj.onc.1201814
Sweeney K.J., Swarbrick A., Sutherland R.L., Musgrove E.A.
Lack of relationship between CDK activity and G1 cyclin expression in breast cancer cells.
Oncogene 16:2865-2878(1998)
PubMed=10969801
Forozan F., Mahlamaki E.H., Monni O., Chen Y.-D., Veldman R., Jiang Y., Gooden G.C., Ethier S.P., Kallioniemi A.H., Kallioniemi O.-P.
Comparative genomic hybridization analysis of 38 breast cancer cell lines: a basis for interpreting complementary DNA microarray data.
Cancer Res. 60:4519-4525(2000)
PubMed=11414198; DOI=10.1007/s004320000207
Lahm H., Andre S., Hoeflich A., Fischer J.R., Sordat B., Kaltner H., Wolf E., Gabius H.-J.
Comprehensive galectin fingerprinting in a panel of 61 human tumor cell lines by RT-PCR and its implications for diagnostic and therapeutic procedures.
J. Cancer Res. Clin. Oncol. 127:375-386(2001)
PubMed=12353263; DOI=10.1002/gcc.10107
Popovici C., Basset C., Bertucci F., Orsetti B., Adelaide J., Mozziconacci M.-J., Conte N., Murati A., Ginestier C., Charafe-Jauffret E., Ethier S.P., Lafage-Pochitaloff M., Theillet C., Birnbaum D., Chaffanet M.
Reciprocal translocations in breast tumor cell lines: cloning of a t(3;20) that targets the FHIT gene.
Genes Chromosomes Cancer 35:204-218(2002)
PubMed=12800145; DOI=10.1002/gcc.10218
Adelaide J., Huang H.-E., Murati A., Alsop A.E., Orsetti B., Mozziconacci M.-J., Popovici C., Ginestier C., Letessier A., Basset C., Courtay-Cahen C., Jacquemier J., Theillet C., Birnbaum D., Edwards P.A.W., Chaffanet M.
A recurrent chromosome translocation breakpoint in breast and pancreatic cancer cell lines targets the neuregulin/NRG1 gene.
Genes Chromosomes Cancer 37:333-345(2003)
PubMed=15153330; DOI=10.1593/neo.3292; PMCID=PMC1502105
Watts G.S., Oshiro M.M., Junk D.J., Wozniak R.J., Watterson S.J., Domann F.E., Futscher B.W.
The acetyltransferase p300/CBP-associated factor is a p53 target gene in breast tumor cells.
Neoplasia 6:187-194(2004)
PubMed=15677628; DOI=10.1093/carcin/bgi032
Gorringe K.L., Chin S.-F., Pharoah P.D.P., Staines J.M., Oliveira C., Edwards P.A.W., Caldas C.
Evidence that both genetic instability and selection contribute to the accumulation of chromosome alterations in cancer.
Carcinogenesis 26:923-930(2005)
PubMed=16397213; DOI=10.1158/0008-5472.CAN-05-2853
Elstrodt F., Hollestelle A., Nagel J.H.A., Gorin M., Wasielewski M., van den Ouweland A.M.W., Merajver S.D., Ethier S.P., Schutte M.
BRCA1 mutation analysis of 41 human breast cancer cell lines reveals three new deleterious mutants.
Cancer Res. 66:41-45(2006)
PubMed=16541312; DOI=10.1007/s10549-006-9186-z
Wasielewski M., Elstrodt F., Klijn J.G.M., Berns E.M.J.J., Schutte M.
Thirteen new p53 gene mutants identified among 41 human breast cancer cell lines.
Breast Cancer Res. Treat. 99:97-101(2006)
PubMed=17157791; DOI=10.1016/j.ccr.2006.10.008; PMCID=PMC2730521
Neve R.M., Chin K., Fridlyand J., Yeh J., Baehner F.L., Fevr T., Clark L., Bayani N., Coppe J.-P., Tong F., Speed T., Spellman P.T., DeVries S., Lapuk A., Wang N.J., Kuo W.-L., Stilwell J.L., Pinkel D., Albertson D.G., Waldman F.M., McCormick F., Dickson R.B., Johnson M.D., Lippman M.E., Ethier S.P., Gazdar A.F., Gray J.W.
A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes.
Cancer Cell 10:515-527(2006)
PubMed=18516279; DOI=10.1016/j.molonc.2007.02.004; PMCID=PMC2391005
Kenny P.A., Lee G.Y., Myers C.A., Neve R.M., Semeiks J.R., Spellman P.T., Lorenz K., Lee E.H., Barcellos-Hoff M.H., Petersen O.W., Gray J.W., Bissell M.J.
The morphologies of breast cancer cell lines in three-dimensional assays correlate with their profiles of gene expression.
Mol. Oncol. 1:84-96(2007)
PubMed=18262045; DOI=10.1016/j.cancergencyto.2007.05.030
Xu J., Chambers A.F., Tuck A.B., Rodenhiser D.I.
Molecular cytogenetic characterization of human breast cancer cell line MDA-MB-468 and its variant 468LN, which displays aggressive lymphatic metastasis.
Cancer Genet. Cytogenet. 181:1-7(2008)
PubMed=19582160; DOI=10.1371/journal.pone.0006146; PMCID=PMC2702084
Kao J., Salari K., Bocanegra M., Choi Y.-L., Girard L., Gandhi J., Kwei K.A., Hernandez-Boussard T., Wang P., Gazdar A.F., Minna J.D., Pollack J.R.
Molecular profiling of breast cancer cell lines defines relevant tumor models and provides a resource for cancer gene discovery.
PLoS ONE 4:E6146-E6146(2009)
DOI=10.25904/1912/1434
Morrison B.J.
Breast cancer stem cells: tumourspheres and implications for therapy.
Thesis PhD (2010); Griffith University; Brisbane; Australia
PubMed=19593635; DOI=10.1007/s10549-009-0460-8
Hollestelle A., Nagel J.H.A., Smid M., Lam S., Elstrodt F., Wasielewski M., Ng S.S., French P.J., Peeters J.K., Rozendaal M.J., Riaz M., Koopman D.G., ten Hagen T.L.M., de Leeuw B.H.C.G.M., Zwarthoff E.C., Teunisse A.F.A.S., van der Spek P.J., Klijn J.G.M., Dinjens W.N.M., Ethier S.P., Clevers H.C., Jochemsen A.G., den Bakker M.A., Foekens J.A., Martens J.W.M., Schutte M.
Distinct gene mutation profiles among luminal-type and basal-type breast cancer cell lines.
Breast Cancer Res. Treat. 121:53-64(2010)
PubMed=20070913; DOI=10.1186/1471-2407-10-15; PMCID=PMC2836299
Tsuji K., Kawauchi S., Saito S., Furuya T., Ikemoto K., Nakao M., Yamamoto S., Oka M., Hirano T., Sasaki K.
Breast cancer cell lines carry cell line-specific genomic alterations that are distinct from aberrations in breast cancer tissues: comparison of the CGH profiles between cancer cell lines and primary cancer tissues.
BMC Cancer 10:15.1-15.10(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=21778573; DOI=10.3233/BD-2010-0307; PMCID=PMC3532890
Chavez K.J., Garimella S.V., Lipkowitz S.
Triple negative breast cancer cell lines: one tool in the search for better treatment of triple negative breast cancer.
Breast Dis. 32:35-48(2010)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=23151021; DOI=10.1186/1471-2164-13-619; PMCID=PMC3546428
Grigoriadis A., Mackay A., Noel E., Wu P.-J., Natrajan R., Frankum J., Reis-Filho J.S., Tutt A.
Molecular characterisation of cell line models for triple-negative breast cancers.
BMC Genomics 13:619.1-619.14(2012)
PubMed=23601657; DOI=10.1186/bcr3415; PMCID=PMC3672661
Riaz M., van Jaarsveld M.T.M., Hollestelle A., Prager-van der Smissen W.J.C., Heine A.A.J., Boersma A.W.M., Liu J.-J., Helmijr J.C.A., Ozturk B., Smid M., Wiemer E.A.C., Foekens J.A., Martens J.W.M.
miRNA expression profiling of 51 human breast cancer cell lines reveals subtype and driver mutation-specific miRNAs.
Breast Cancer Res. 15:R33.1-R33.17(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=24094812; DOI=10.1016/j.ccr.2013.08.020; PMCID=PMC3931310
Timmerman L.A., Holton T., Yuneva M., Louie R.J., Padro M., Daemen A., Hu M., Chan D.A., Ethier S.P., van 't Veer L.J., Polyak K., McCormick F., Gray J.W.
Glutamine sensitivity analysis identifies the xCT antiporter as a common triple-negative breast tumor therapeutic target.
Cancer Cell 24:450-465(2013)
PubMed=24162158; DOI=10.1007/s10549-013-2743-3; PMCID=PMC3832776
Prat A., Karginova O., Parker J.S., Fan C., He X.-P., Bixby L.M., Harrell J.C., Roman E., Adamo B., Troester M.A., Perou C.M.
Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes.
Breast Cancer Res. Treat. 142:237-255(2013)
PubMed=24176112; DOI=10.1186/gb-2013-14-10-r110; PMCID=PMC3937590
Daemen A., Griffith O.L., Heiser L.M., Wang N.J., Enache O.M., Sanborn Z., Pepin F., Durinck S., Korkola J.E., Griffith M., Hur J.S., Huh N., Chung J., Cope L., Fackler M.J., Umbricht C.B., Sukumar S., Seth P., Sume V.P., Jakkula L.R., Lu Y.-L., Mills G.B., Cho R.J., Collisson E.A., van 't Veer L.J., Spellman P.T., Gray J.W.
Modeling precision treatment of breast cancer.
Genome Biol. 14:R110.1-R110.14(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25699542; DOI=10.15252/msb.20145664; PMCID=PMC4358660
Muellner M.K., Mair B., Ibrahim Y., Kerzendorfer C., Lechtermann H., Trefzer C., Klepsch F., Muller A.C., Leitner E., Macho-Maschler S., Superti-Furga G., Bennett K.L., Baselga J., Rix U., Kubicek S., Colinge J., Serra V., Nijman S.M.B.
Targeting a cell state common to triple-negative breast cancers.
Mol. Syst. Biol. 11:789-789(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25892236; DOI=10.1016/j.celrep.2015.03.050; PMCID=PMC4425736
Lawrence R.T., Perez E.M., Hernandez D., Miller C.P., Haas K.M., Irie H.Y., Lee S.-I., Blau C.A., Villen J.
The proteomic landscape of triple-negative breast cancer.
Cell Rep. 11:630-644(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28287265; DOI=10.1021/acs.jproteome.6b00470; PMCID=PMC5557415
Yen T.-Y., Bowen S., Yen R., Piryatinska A., Macher B.A., Timpe L.C.
Glycoproteins in claudin-low breast cancer cell lines have a unique expression profile.
J. Proteome Res. 16:1391-1400(2017)
PubMed=28889351; DOI=10.1007/s10549-017-4496-x
Saunus J.M., Smart C.E., Kutasovic J.R., Johnston R.L., Kalita-de Croft P., Miranda M., Rozali E.N., Vargas A.C., Reid L.E., Lorsy E., Cocciardi S., Seidens T., McCart Reed A.E., Dalley A.J., Wockner L.F., Johnson J., Sarkar D., Askarian-Amiri M.E., Simpson P.T., Khanna K.K., Chenevix-Trench G., Al-Ejeh F., Lakhani S.R.
Multidimensional phenotyping of breast cancer cell lines to guide preclinical research.
Breast Cancer Res. Treat. 167:289-301(2018)
PubMed=29273624; DOI=10.1101/gr.226019.117; PMCID=PMC5793780
Franco H.L., Nagari A., Malladi V.S., Li W.-Q., Xi Y.-X., Richardson D., Allton K.L., Tanaka K., Li J., Murakami S., Keyomarsi K., Bedford M.T., Shi X.-B., Li W., Barton M.C., Dent S.Y.R., Kraus W.L.
Enhancer transcription reveals subtype-specific gene expression programs controlling breast cancer pathogenesis.
Genome Res. 28:159-170(2018)
PubMed=30613774; DOI=10.1126/sciadv.aau7314; PMCID=PMC6314821
Vande Voorde J., Ackermann T., Pfetzer N., Sumpton D., Mackay G., Kalna G., Nixon C., Blyth K., Gottlieb E., Tardito S.
Improving the metabolic fidelity of cancer models with a physiological cell culture medium.
Sci. Adv. 5:eaau7314.1-eaau7314.14(2019)
PubMed=30787054; DOI=10.1158/1055-9965.EPI-18-1132; PMCID=PMC6548687
Hooker S.E. Jr., Woods-Burnham L., Bathina M., Lloyd S., Gorjala P., Mitra R., Nonn L., Kimbro K.S., Kittles R.A.
Genetic ancestry analysis reveals misclassification of commonly used cancer cell lines.
Cancer Epidemiol. Biomarkers Prev. 28:1003-1009(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31978347; DOI=10.1016/j.cell.2019.12.023; PMCID=PMC7339254
Nusinow D.P., Szpyt J., Ghandi M., Rose C.M., McDonald E.R. 3rd, Kalocsay M., Jane-Valbuena J., Gelfand E.T., Schweppe D.K., Jedrychowski M.P., Golji J., Porter D.A., Rejtar T., Wang Y.K., Kryukov G.V., Stegmeier F., Erickson B.K., Garraway L.A., Sellers W.R., Gygi S.P.
Quantitative proteomics of the Cancer Cell Line Encyclopedia.
Cell 180:387-402.e16(2020)
PubMed=32576280; DOI=10.1186/s13058-020-01300-y; PMCID=PMC7310532
Finlay-Schultz J., Jacobsen B.M., Riley D., Paul K.V., Turner S., Ferreira-Gonzalez A., Harrell J.C., Kabos P., Sartorius C.A.
New generation breast cancer cell lines developed from patient-derived xenografts.
Breast Cancer Res. 22:68.1-68.12(2020)"