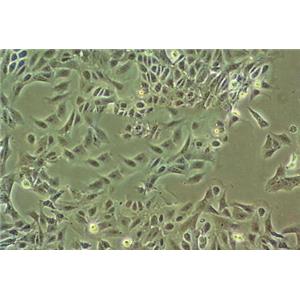
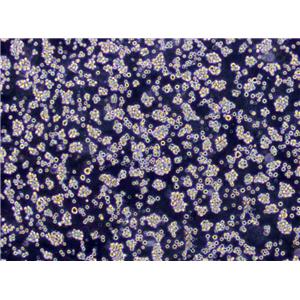
"HEL人红白细胞白血病细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:悬浮生长
公司细胞系形态漂亮、增殖倍数高、纯度高、功能性强,细胞培养就跟养孩子一个样。养孩子要喂奶,养细胞要加补液,都需要在前期补充足够的营养,初始状态的细胞或刚刚复苏的细胞还要适量加入血清或细胞因子来帮助它们的存活增殖,如果营养物质缺乏,细胞就会不生长甚至死亡。养孩子要从小培养学习,养细胞也得培养宝宝顺利生下来,你会经常抚摸他,给他看各种颜色,刺激他的五感。细胞也是一样,分离后的细胞需要使用特定的细胞因子进行活化、增殖。另外加入因子的种类、因子的浓度、加入时间、加入顺序都会影响细胞最终的结果。养孩子最怕孩子生病,养细胞最怕被污染,平时你会仔细观察宝宝是否呕吐、是否突然哭闹,猜测宝宝是否生病了。对于细胞,我们也需要时刻进行观察的,假如培养液浑浊(污染了),则需要换液后加抗生素;假如细胞增殖不明显,形态变差,则可能是因为营养不足了,对贴壁细胞可以消化后重新用新的培养基接种并加倍加入细胞因子含量;对悬浮细胞增殖能力不强的,则不着急补液,只是先补加血清、细胞因子看是否可以好转。培养时还得全程在无菌的环境,一个小小的偏差,细胞就会死亡。
换液周期:每周2-3次
RPTEC/TERT 1 Cells;背景说明:肾;近端小管上皮;HGNC-TERT转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RFL6细胞、NCIH1568细胞、Psi2 DAP细胞
786.O Cells;背景说明:该细胞源自一位原发性肾透明细胞癌患者。该细胞有微绒毛和桥粒,能在软琼脂上生长。此细胞生成一种PTH(甲状旁腺素)样的多肽,与乳癌和肺癌中生成的肽相似,其N端序列与PTH相似,具有PTH样活性,分子量为6000D。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NCIH2347细胞、Caov-3细胞、NCIH1838细胞
Balb/c 3T3 Cells;背景说明:胚胎;成纤维;自发永生;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ACCM细胞、NRK细胞、TOV-21G细胞
HEL人红白细胞白血病细胞代次低|培养基|送STR图谱
背景信息:这株淋巴母细胞样细胞株,源自一位30岁白人男性一,患有恶性红细胞白血病,能够自然产生并能诱导球蛋白合成。细胞的EB病毒核抗原阴性,没有表面免疫球蛋白与细胞质免疫球蛋白。HEL细胞表达HLA抗原(HLA-A3,AW32,BW35),β-2小球蛋白,一定比例的细胞还表达Ia抗原。这个细胞株提供了一种用于研究红细胞分化和球蛋白基因表达的模型。它类似于小鼠中的血友病。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
HEL人红白细胞白血病细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
WSU-DLCL(2) Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MDCK细胞、V 79-4细胞、HCV 29细胞
TK-1 Cells;背景说明:淋巴瘤;AKR/Cum;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:TM3细胞、PC-3M细胞、GLC-15细胞
WM-266-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:CCD-966SK细胞、T-HSC细胞、MCF-7/ADR-RES细胞
P3HR1 Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:SNU119细胞、KYSE 450细胞、Clone 166细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:淋巴母细胞样
贴壁细胞消化传代时通常采用两种方法:一、加入胰酶等细胞脱落后,再加培养基中止胰酶作用,离心传代;二、加入胰酶后,镜下观察待细胞始脱落时,弃胰酶,加培养分瓶。但前者太麻烦,而后者有可能对细胞施加胰酶选择,因为总是贴壁不牢的细胞先脱落,对肿瘤细胞来说,这部分细胞有可能是恶性程度较GAO的细胞亚群。一种简单的消化传代方法。加入PBS洗去血清或加入胰酶先中和血清的作用(30s),弃之,再加入适量胰酶作用10s-40s(根据细胞消化的难易程度),弃之,这样依赖残余的胰酶就可将细胞消化单细胞。对于较难消化的细胞,可以用2%利多卡因消化5-8分钟,然后再弃去,加培养基吹打也可以,对细胞的影响不大。不用PBS也不用Hanks洗,只要把旧培养吸的干净一点,直接加酶消化应该不会有什么问题。弃培养后,用0.04%的EDA冲洗一次,再用1/4v的0.04%的EDA室温孵育5min,弃取大部分EDA,加入与剩余EDA等量的胰酶(预热)总体积1/10v。消化到有细胞脱落。不过有人说EDA对细胞不HAO,有证据吗?培养的BASMC:倒掉旧培养加入少量胰酶冲一下,倒掉再加入0.125-0.25%胰酶约6-10滴或1ml(25ml bole)消化再加入适量新培养基中和,并分瓶这种方法简单、省事;效果很HAO并且不损失细胞!
BSC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COV 504细胞、Tn5B1-4细胞、RBMVEC细胞
U266 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MV-4-11细胞、H1993细胞、AZ-521细胞
SU.86.86 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:D6P2T细胞、U031细胞、IMR90细胞
AK Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Virginia Mason Research Center-Renal Cancer Z细胞、HGMC细胞、TE-13细胞
MV4-11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EBTr (NBL-4)细胞、AC16 [Human hybrid]细胞、SKLU01细胞
VMRCRCZ Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:BE(2)-C细胞、SL1细胞、NU-GC-3细胞
UCH-1 Cells;背景说明:骶骨脊索瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RCC10RGB细胞、LA-N-5细胞、H1944细胞
CEM.C1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:10RGB细胞、NCIH23细胞、MN-9D细胞
LC1sq Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Fukuoka University-Malignant mixed Mullerian Tumor-1细胞、RPMI-8226S细胞、RenCa细胞
C-28I2 Cells;背景说明:软骨;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPPVEC细胞、H-1650细胞、NCI-H1993细胞
EFM19 Cells;背景说明:乳腺管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCC78细胞、MC3T3细胞、SKRC-52细胞
SEG-1 Cells;背景说明:食管腺癌;胸腔积液转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H-1522细胞、CNLMG-B5537SKIN细胞、AR4-2J细胞
KMB-17 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:TE354T细胞、IOSE-80细胞、Med 283细胞
U118-MG Cells;背景说明:注意: 据报道来自不同个体的胶质母细胞瘤细胞株U-118 MG (HTB-15) 和 U-138 MG (HTB-16)有着一致的VNTR和相近的STR模式。 U-118 MG 和 U-138 MG细胞遗传学上很相似并有至少六个衍生标记染色体。 这是1966年至1969年间J. Ponten和同事从恶性神经胶质瘤中构建的细胞株中的一株(其它包括ATCC HTB-14和 ATCC HTB-16 and ATCC HTB-17)。 1987年用BM-Cycline培养6周去除了支原体污染。 ;传代方法: 消化3-5分钟。1:2传代。3天内可长满。;生长特性:贴壁生长;形态特性:混合型;相关产品有:SV-HUC细胞、brain-derived Endothelial cells.3细胞、GH 3细胞
SNU-398 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NOZ细胞、LICR-HN-6细胞、NF639细胞
L-6 myoblast Cells;背景说明:该细胞是Yaffe在甲基胆蒽存在的情况下从大鼠大腿肌原代培养的最初两代细胞中分离得到的;在培养基中融合形成多核的肌管和横纹肌纤维,细胞融合的程度随着代数的增加而下降,因此细胞应低代次冷冻并周期性地重新克隆以选择融合能力强的细胞。鼠痘病毒阴性。该细胞应在达汇合状态前传代,以延缓细胞分化能力的丧失。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成肌细胞样;相关产品有:P30-OHKUBO细胞、SKCO-1细胞、TALL-104细胞
UC-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HCFB(HCF)细胞、NCIH650细胞、DH-82细胞
Abcam A-549 CD274 KO 1 Cells(提供STR鉴定图谱)
Abcam Raji VPS37A KO Cells(提供STR鉴定图谱)
BayGenomics ES cell line BGC474 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRS244 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA445 Cells(提供STR鉴定图谱)
chHES-108 Cells(提供STR鉴定图谱)
DA01912 Cells(提供STR鉴定图谱)
ERC-37 Cells(提供STR鉴定图谱)
GM06332 Cells(提供STR鉴定图谱)
HuPT3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:LU165细胞、624-mel细胞、COLO-738细胞
HEL人红白细胞白血病细胞代次低|培养基|送STR图谱
Ketr3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HRA19细胞、VE细胞、RPMI7666细胞
NCI-H510 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;每周换液2次。;生长特性:混合生长;形态特性:上皮细胞样;相关产品有:NCI-H1755细胞、BE(2)M17细胞、BI-Mel细胞
U118 Cells;背景说明:注意: 据报道来自不同个体的胶质母细胞瘤细胞株U-118 MG (HTB-15) 和 U-138 MG (HTB-16)有着一致的VNTR和相近的STR模式。 U-118 MG 和 U-138 MG细胞遗传学上很相似并有至少六个衍生标记染色体。 这是1966年至1969年间J. Ponten和同事从恶性神经胶质瘤中构建的细胞株中的一株(其它包括ATCC HTB-14和 ATCC HTB-16 and ATCC HTB-17)。 1987年用BM-Cycline培养6周去除了支原体污染。 ;传代方法: 消化3-5分钟。1:2传代。3天内可长满。;生长特性:贴壁生长;形态特性:混合型;相关产品有:HNE-2细胞、GM17346细胞、MRAEC细胞
HEK-EBNA Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:10传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SK-ES细胞、Statens Seruminstitut Rabbit Cornea细胞、MD Anderson-Metastatic Breast-436细胞
EOC-20 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SU-DHL-10细胞、KYSE-30细胞、MTC-TT细胞
AG22820 Cells(提供STR鉴定图谱)
NCIH2066 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:CAL-62细胞、hTERT-HME-1细胞、SUDHL10细胞
8305C_1 Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H2087细胞、P-815细胞、H345细胞
GM03570E Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SU-DHL-2细胞、WM-239A细胞、Lu-165细胞
Hce8693 Cells;背景说明:盲肠腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:UPCI-SCC-154细胞、ME-1 [Human leukemia]细胞、CMT-167细胞
BALB/3T3 clone A31 Cells;背景说明:胚胎;成纤维;自发永生;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CHO-ori细胞、RC-2细胞、LS-174细胞
NCIH2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:74Int细胞、HPAFII细胞、Homo sapiens No. 578, breast cells细胞
NCIH2081 Cells;背景说明:详见相关文献介绍;传代方法:随细胞的密度而增加;生长特性:悬浮生长;形态特性:聚团悬浮;相关产品有:74Int细胞、HPAFII细胞、Homo sapiens No. 578, breast cells细胞
HTori-3.1 Cells;背景说明:甲状腺;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BEL-7405细胞、TEC细胞、P3HR-1细胞
GM11749 Cells(提供STR鉴定图谱)
HAP1 DDR2 (-) 2 Cells(提供STR鉴定图谱)
SK-BR-3 Cells;背景说明:这株细胞源自胸水。没有病毒颗粒。亚显微结构特征包括微丝和桥粒,肝糖原颗粒,大溶酶体,成束的细胞质纤丝。SK-BR-3细胞株过表达HER2/c-erb-2基因产物。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KMS11细胞、CAMA-1细胞、OVCAR 432细胞
MDCK.2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Michigan Cancer Foundation-10A细胞、Shadyside Hospital Pittsburgh-77细胞、Hep G2-Luc细胞
HuH28 Cells;背景说明:肝内胆管癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IHH4细胞、LS 513细胞、PTK-2细胞
RPMI-6666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SKG3A细胞、MC-116细胞、Madin-Darby Bovine Kidney细胞
H378 Cells;背景说明:小细胞肺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:McA-RH 8994细胞、H-929细胞、293H细胞
HCT FET Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NBLAN5T细胞、MDCK supertube细胞、HO1-N-1细胞
LAN6 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TE-8细胞、AD-293细胞、U373细胞
GA-10 (Clone 4) Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:JM细胞、CEM细胞、KMY1022细胞
HH0279 Cells(提供STR鉴定图谱)
iNV12 Cells(提供STR鉴定图谱)
M950504 Cells(提供STR鉴定图谱)
ND07088 Cells(提供STR鉴定图谱)
PER-VAS Cells(提供STR鉴定图谱)
Ubigene HCT 116 CACNA1H KO Cells(提供STR鉴定图谱)
VM-M3/Fluc Cells(提供STR鉴定图谱)
HCT116-SLC10A4-KO-c8 Cells(提供STR鉴定图谱)
Hs729 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:Panc 08.13细胞、HONE1细胞、MC3T3-E细胞
NS-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:MADB 106细胞、MLOY4细胞、KYSE-140细胞
BT 325 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BCaP-37细胞、KNS-62细胞、DU-4475细胞
NCI.H23 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KYSE450细胞、751细胞、AG06814-J细胞
AM38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:RPMI8226/S细胞、VMRCLCD细胞、Co 205细胞
AM38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:RPMI8226/S细胞、VMRCLCD细胞、Co 205细胞
CV 1 Cells;背景说明:CV-1细胞株是1964年由JensenFC等建系的,源自成年雄性非洲绿猴肾,被用于Rous肉瘤病毒的转染研究。可作为SV40载体的转染宿主。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:hADSCs细胞、BALL-1细胞、Z310细胞
MC/CAR Cells;背景说明:B淋巴;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:A-2058细胞、A 72细胞、WEHI 3B细胞
OCI-Ly1 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:THPI细胞、Mahlavu细胞、HCC1500细胞
PC2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Nb2细胞、HOP92细胞、HLEB3细胞
AD-293 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:C4-2 Bone metastatic细胞、KYSE 150 KYSE150 Kyse150 KY150
细胞、Hs 888Lu细胞
AgC11x3A Cells;背景说明:NR8383(正常大鼠,1983年8月3日)来源于肺灌洗时的正常大鼠肺泡巨噬细胞。细胞在gerbil肺细胞连续培养液存在下培养了大约8-9个月。随后,不再需要外源生长因子。通过有限稀释法从单个细胞克隆并亚克隆NR8383细胞,并三次用软琼脂亚克隆。细胞表现出巨噬细胞的特性,吞噬酵母多糖和铜绿,非特异性脂酶活性,Fc受体,氧化降解;分泌IL-1,TNFbeta和IL-6,可重复地响应外源生长因子。NR8383细胞响应博莱霉素,分泌TGFbeta前体。在博莱霉素刺激下,TGFbe;传代方法:1:2传代;生长特性:半贴壁生长;形态特性:巨噬细胞;相关产品有:Cloudman M3细胞、SUM 102细胞、NTera 2/cl.D1细胞
L5178-R Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Karpas-299细胞、GM3190细胞、P3X63-Ag8.653细胞
HOP 62 Cells;背景说明:肺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU886细胞、NCI-SNU-16细胞、H-1105细胞
Human Corneal Epithelial cells-Transformed Cells;背景说明:角膜上皮细胞;Ad-SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH1755细胞、NCI H69细胞、WEHI-3B细胞
Seki Cells(提供STR鉴定图谱)
AAV-293 Cells;背景说明:我们推荐使用AAV-293细胞株繁殖腺病毒相关重组病毒。 AAV-293源自普遍使用的 HEK293细胞株,但产生的病毒滴度更高。 HEK293细胞是剪切过的腺病毒5型DNA转染的人胚肾细胞。 跟HEK293细胞一样,AAV-293细胞反式表达腺病毒E1基因,当共转染三个AAV助质粒(一个含ITR的质粒,pAAV-RC, 和E1缺失助质粒)时,可以产生有感染力的腺病毒-相关病毒颗粒。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:P19细胞、A1847细胞、HECV细胞
HCC827 Cells;背景说明:这株细胞建于1994年三月。这株肺腺癌在EGFR酪酸激酶区域有一个获得性突变(E746-A750缺失)。患者在25岁到26岁时每个月抽1包烟。在诊断前12年不再抽烟。;传代方法:消化5分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:CAL 148细胞、CHO-S细胞、NTERA-2/D1细胞
GM07404 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:639-V细胞、Pa017C细胞、OCI/AML-5细胞
T-47-D Cells;背景说明:浸润性导管癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BTI-TN5B1-4细胞、Panc_08_13细胞、KMST6细胞
MDA-468 Cells;背景说明:该细胞是1977年由CailleauR等从一位患有转移性乳腺癌的51岁黑人女性的胸腔积液中分离得到的。虽然供体组织的G6PD等位基因杂合,但此细胞株始终表现为G6PDA表型。P53基因273位密码子存在G→A突变,从而导致Arg→His替代。每个细胞上存在1×106个EGF受体。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HOC-1细胞、SO-Rb 50细胞、TCCPAN2细胞
Renal Carcinoma Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:LNCaP subline C4-2细胞、H9c2细胞、Hs 888.T细胞
HEL人红白细胞白血病细胞代次低|培养基|送STR图谱
NPC-TW-039 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MDAMB231细胞、OV433细胞、LWnt3A细胞
LCLC-103H Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Mouse Forestomach Carcinoma细胞、RPMI 7666细胞、PA-TU-8988S细胞
HuH1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H747细胞、Wien133细胞、AKR细胞
HEY A8 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ME-180细胞、Mel-RM细胞、PFSK细胞
H676B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CEM T4细胞、SUM 149细胞、KMB 17细胞
H1651 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:OVSAHO细胞、R3/1细胞、NCI-H2141细胞
B10BR Cells;背景说明:永生化黑素 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PLA801-95C细胞、1E8-H细胞、U266-B1细胞
SLK Cells;背景说明:肉瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HPAF II细胞、P3HRI细胞、HRMC细胞
BayGenomics ES cell line RRJ689 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XK742 Cells(提供STR鉴定图谱)
G45-60 Cells(提供STR鉴定图谱)
NS0bcl-2 Cells(提供STR鉴定图谱)
Y1-8BrcAMPr-1 Cells(提供STR鉴定图谱)
MciA Cells(提供STR鉴定图谱)
" "PubMed=3159941; DOI=10.1016/0145-2126(85)90134-1
Drexler H.G., Gaedicke G., Minowada J.
Isoenzyme studies in human leukemia-lymphoma cell lines -- III Beta-hexosaminidase (E.C. 3.2.1.30).
Leuk. Res. 9:549-559(1985)
PubMed=3874327; DOI=10.1016/0145-2126(85)90133-x
Drexler H.G., Gaedicke G., Minowada J.
Isoenzyme studies in human leukemia-lymphoma cells lines -- II. Acid phosphatase.
Leuk. Res. 9:537-548(1985)
CLPUB00447
Mulivor R.A., Suchy S.F.
1992/1993 catalog of cell lines. NIGMS human genetic mutant cell repository. 16th edition. October 1992.
(In misc. document) Institute for Medical Research (Camden, N.J.) NIH 92-2011; pp.1-918; National Institutes of Health; Bethesda; USA (1992)
PubMed=7538619; DOI=10.1016/0145-2126(94)00160-c
Kubota A., Okamura S., Shimoda K., Ikematsu W., Otsuka T., Niho Y.
Analysis of c-kit expression of human erythroleukemia cell line, HEL: clonal variation and relationship with erythroid and megakaryocytic phenotype.
Leuk. Res. 19:283-290(1995)
PubMed=8558913
Morita S., Tsuchiya S., Fujie H., Itano M., Ohashi Y., Minegishi M., Imaizumi M., Endo M., Takano N., Konno T.
Cell surface c-kit receptors in human leukemia cell lines and pediatric leukemia: selective preservation of c-kit expression on megakaryoblastic cell lines during adaptation to in vitro culture.
Leukemia 10:102-105(1996)
PubMed=9290701; DOI=10.1002/(SICI)1098-2744(199708)19:4<243::AID-MC5>3.0.CO;2-D
Jia L.-Q., Osada M., Ishioka C., Gamo M., Ikawa S., Suzuki T., Shimodaira H., Niitani T., Kudo T., Akiyama M., Kimura N., Matsuo M., Mizusawa H., Tanaka N., Koyama H., Namba M., Kanamaru R., Kuroki T.
Screening the p53 status of human cell lines using a yeast functional assay.
Mol. Carcinog. 19:243-253(1997)
PubMed=9510473; DOI=10.1111/j.1349-7006.1998.tb00476.x; PMCID=PMC5921588
Hosoya N., Hangaishi A., Ogawa S., Miyagawa K., Mitani K., Yazaki Y., Hirai H.
Frameshift mutations of the hMSH6 gene in human leukemia cell lines.
Jpn. J. Cancer Res. 89:33-39(1998)
PubMed=9738977; DOI=10.1111/j.1349-7006.1998.tb03275.x; PMCID=PMC5921886
Takizawa J., Suzuki R., Kuroda H., Utsunomiya A., Kagami Y., Joh T., Aizawa Y., Ueda R., Seto M.
Expression of the TCL1 gene at 14q32 in B-cell malignancies but not in adult T-cell leukemia.
Jpn. J. Cancer Res. 89:712-718(1998)
PubMed=10739008; DOI=10.1016/S0145-2126(99)00182-4
Inoue K., Kohno T., Takakura S., Hayashi Y., Mizoguchi H., Yokota J.
Frequent microsatellite instability and BAX mutations in T cell acute lymphoblastic leukemia cell lines.
Leuk. Res. 24:255-262(2000)
PubMed=11021758; DOI=10.1038/sj.leu.2401891
Majka M., Rozmyslowicz T., Honczarenko M.J., Ratajczak J., Wasik M.A., Gaulton G.N., Ratajczak M.Z.
Biological significance of the expression of HIV-related chemokine coreceptors (CCR5 and CXCR4) and their ligands by human hematopoietic cell lines.
Leukemia 14:1821-1832(2000)
PubMed=11226526; DOI=10.1016/S0145-2126(00)00121-1
Inoue K., Kohno T., Takakura S., Hayashi Y., Mizoguchi H., Yokota J.
Corrigendum to: Frequent microsatellite instability and BAX mutations in T cell acute lymphoblastic leukemia cell lines Leukemia Research 24 (2000), 255-262.
Leuk. Res. 25:275-278(2001)
PubMed=14504097; DOI=10.1182/blood-2003-02-0418
Taketani T., Taki T., Sugita K., Furuichi Y., Ishii E., Hanada R., Tsuchida M., Sugita K., Ida K., Hayashi Y.
FLT3 mutations in the activation loop of tyrosine kinase domain are frequently found in infant ALL with MLL rearrangements and pediatric ALL with hyperdiploidy.
Blood 103:1085-1088(2004)
PubMed=16408098; DOI=10.1038/sj.leu.2404081
Quentmeier H., MacLeod R.A.F., Zaborski M., Drexler H.G.
JAK2 V617F tyrosine kinase mutation in cell lines derived from myeloproliferative disorders.
Leukemia 20:471-476(2006)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23955599; DOI=10.1038/ng.2731
Kon A., Shih L.-Y., Minamino M., Sanada M., Shiraishi Y., Nagata Y., Yoshida K.-i., Okuno Y., Bando M., Nakato R., Ishikawa S., Sato-Otsubo A., Nagae G., Nishimoto A., Haferlach C., Nowak D., Sato Y., Alpermann T., Nagasaki M., Shimamura T., Tanaka H., Chiba K., Yamamoto R., Yamaguchi T., Otsu M., Obara N., Sakata-Yanagimoto M., Nakamaki T., Ishiyama K., Nolte F., Hofmann W.-K., Miyawaki S., Chiba S., Mori H., Nakauchi H., Koeffler H.P., Aburatani H., Haferlach T., Shirahige K., Miyano S., Ogawa S.
Recurrent mutations in multiple components of the cohesin complex in myeloid neoplasms.
Nat. Genet. 45:1232-1237(2013)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27277069; DOI=10.1160/TH15-11-0891; PMCID=PMC5080539
Wright J.R., Amisten S., Goodall A.H., Mahaut-Smith M.P.
Transcriptomic analysis of the ion channelome of human platelets and megakaryocytic cell lines.
Thromb. Haemost. 116:272-284(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=29533902; DOI=10.1515/hsz-2017-0321
Drexler H.G., Pommerenke C., Eberth S., Nagel S.
Hodgkin lymphoma cell lines: to separate the wheat from the chaff.
Biol. Chem. 399:511-523(2018)
PubMed=30285677; DOI=10.1186/s12885-018-4840-5; PMCID=PMC6167786
Tan K.-T., Ding L.-W., Sun Q.-Y., Lao Z.-T., Chien W., Ren X., Xiao J.-F., Loh X.-Y., Xu L., Lill M., Mayakonda A., Lin D.-C., Yang H.H., Koeffler H.P.
Profiling the B/T cell receptor repertoire of lymphocyte derived cell lines.
BMC Cancer 18:940.1-940.13(2018)
PubMed=30629668; DOI=10.1371/journal.pone.0210404; PMCID=PMC6328144
Uphoff C.C., Pommerenke C., Denkmann S.A., Drexler H.G.
Screening human cell lines for viral infections applying RNA-Seq data analysis.
PLoS ONE 14:E0210404-E0210404(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"