"U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
HMy2.CIR Cells;背景说明:B细胞;EBV转染;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:P3X63 Ag8.653细胞、OCI/AML3细胞、MC-38细胞
SCC154 Cells;背景说明:舌鳞癌;男性;HPV6转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Plaepi 34细胞、VM-CUB1细胞、BHT101细胞
H-1793 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:ECC-10细胞、Swiss 3T6细胞、SCC-25细胞
U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱
背景信息:骨肉瘤;女性
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
ATCC细胞库(American Type Culture Colection),该中心一直致力于细胞分类、鉴定和保藏工作。ATCC是全球最大的生物资源保藏中心,ATCC通过行业标准产品、服务和创新解决方案支持全球学术、政府、生物技术、制药、食品、农业和工业领域的科学进步。ATCC提供的服务和定制解决方案包括细胞和微生物培养、鉴定、生物衍生物的开发和生产、性能测试和生物资源保藏服务。美国国家标准协会(ANSI)认可了ATCC标准开发组织,并制定了标准协议,以确保生物材料的可靠性和可重复性。ATCC的使命是为了获取、鉴定、保存、开发、标准化和分发生物资源和生物信息,以提高和应用生物科学知识。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
HOS(TE85) Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HAVSMC细胞、NCI-841细胞、NCIH2347细胞
TMD-8 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:293-FT细胞、SKNBE-2细胞、CEM-0细胞
SF126 Cells;背景说明:该细胞来源于星形胶质细胞瘤;胶质纤维酸性蛋白(GFAP)阴性;可以特异地结合β-内啡肽。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:KMH-2细胞、MCA 205细胞、MKN 7细胞
U-373-MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MC 3T3-E1细胞、WSU-DLCL-2细胞、PANC0327细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
贴壁细胞的消化方法介绍:1、胰酶。这是用得Zui多的。一般浓度在0.25-0.5%。作用时间根据细胞种类、作用温度等因素而变化很大,从几分钟到几十分钟不等。0.25%的胰酶作用于单层贴壁的细胞,在37度条件下,一般消化1-5分钟就足够了。终止是用血清。主要作用于细胞间。配制时不能用含、镁的平衡,否则影响活性。保存于-20度。2、胶原酶。这种方法比较少,一般是用原代培养时,从组织消化下细胞。这种方法作用温和,对细胞损伤较小,但是,价格也较贵。中止同样是用血清。3、EDA。用得也是非常多。一般浓度在0.02%左右。作用于细胞与间质,对细胞间也有一定作用。注意,它能显著影响pH值,而且在弱碱性条件下才易溶。因此,配制时应调节HAO碱度。它不能被终和。因此,消化下来的细胞要洗一遍。4、商品化的无酶消化。个人的使用经常觉得对细胞的损伤比较大,但是分离成单细胞悬的能力确实比较强。5、物理法。直接吹打或用细胞刮子将细胞刮下来。6、冷冻法。此方法仅能用于细胞传代时。无法使组织上的细胞脱落下来。本方法的原理,我想是因细胞冷冻后收缩,从而从培养瓶上脱落下来。YOU点是:对细胞损伤小,不需要中止或洗细胞,方便,不需要另外配制消化。别适用那些贴壁不是别紧,又别娇气的细胞。不足是细胞常成小片脱落。此种方法曾用于因用其它方法传代导致大量细胞死亡操作的间充质干细胞、DC细胞的培养,效果非常满意。具体过程是:1、用较多的4度的PBS 洗涤一遍细胞(以6孔板为例,加1.5ml/孔),2、再加0.5毫升4度的PBS,静置操作台上,很快细胞就小片脱落,3、轻轻吹打,细胞即完全脱落,4、按一定比例传代。
MM6 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:Kit225细胞、Rabbit Kidney 13细胞、C3A细胞
JeKo-1 Cells;背景说明:一位套细胞淋巴瘤患者的巨细胞变种显示白血病转变,从其外周血单核细胞出发建立了MCL细胞株JeKo-1。 JeKo-1细胞EB病毒阴性,并表达一种B细胞表型的IgM。 细胞过表达cyclin D1, Bcl-2, c-Myc 及 Rb 蛋白。 Bcl-1/J(H)基因重排得到了PCR证实。 JeKo-1细胞在SCID小鼠中高成瘤。 [PubMed: 9753063];传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:UM2细胞、BCP-1细胞、H1734细胞
TE6 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MEF细胞、Hs 274.T细胞、DLM8细胞
SK-Col-1 Cells;背景说明:该细胞来源于结直肠病人的转移性腹水。;传代方法:1:2-1:3传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H-1838细胞、NIE-115细胞、TYK-nu细胞
MDA MB 436 Cells;背景说明:该细胞源于一名43岁患有乳腺腺癌女性的胸腔积液。;传代方法:1:2传代,每周换液2—3次;生长特性:贴壁生长;形态特性:多角形;相关产品有:U266-B1细胞、EAC-E2G8细胞、KM12-SM细胞
MUTZ-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H1915细胞、NCIH1734细胞、DV-90细胞
SK-MEL-MeWo Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:SP2-0-Ag14细胞、Ly8细胞、NEC细胞
mRTEC Cells;背景说明:肾小管;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MOLM-14细胞、MES13细胞、LL2细胞
SNU423 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCILY-10细胞、GOTO细胞、EFM192A细胞
GM-3573 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:CCD18细胞、COLO-394细胞、CCRF-CEM C7细胞
BIC1 Cells;背景说明:食管腺癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H1048细胞、TR 146细胞、AHH-1细胞
V79-GalK1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM05372E细胞、NCIH322细胞、NCIH2196细胞
H64 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EC109细胞、NCI H82细胞、PLC PRF 5细胞
UT-7 Cells;背景说明:该细胞于1988年建系;源于一名64岁患有急性粒细胞白血病(AMLM7)的男性的骨髓;对多种细胞因子有反应。;传代方法:维持细胞浓度在1.0-1.5×106cells/ml,1:2传代,2-3天1次。;生长特性:悬浮生长,有1%~2%的细胞可轻微贴壁。;形态特性:圆形;相关产品有:HCCC-9810细胞、Panc_04_03细胞、FL62891细胞
Hs 683 Cells;背景说明:该细胞源自76岁白人男性的左颞叶侧胶质瘤组织,有微绒毛,无桥粒。 ;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RDES细胞、Hs606细胞、HuH-6细胞
PIGI Cells;背景说明:皮肤;黑色素细胞;HPV16转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GTL 16细胞、HS-683细胞、Y3细胞
PECAPJ34 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BT细胞、C518细胞、Hs611T细胞
Abcam A-549 NF1 KO Cells(提供STR鉴定图谱)
Abgenix 4.292.3 Cells(提供STR鉴定图谱)
BayGenomics ES cell line CSG446 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRU358 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTC802 Cells(提供STR鉴定图谱)
CHO-CYP2B6 C1 Cells(提供STR鉴定图谱)
DA02492 Cells(提供STR鉴定图谱)
DA04778 Cells(提供STR鉴定图谱)
FS-3 [Human foreskin] Cells(提供STR鉴定图谱)
NCTC clone 1469 Cells;背景说明:1952年建系,源于正常C3H/An小鼠的肝脏组织,表达H-2K抗原,鼠痘病毒阴性。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:淋巴母细胞;相关产品有:Hs 729T细胞、Centre Antoine Lacassagne-27细胞、WM 239细胞
U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱
Reuber-H-35 hepatoma Cells;背景说明:在糖皮质激素、胰岛素或cAMP衍生物的诱导下可以产生酪酸基转移酶;可被逆转录病毒感染;可产生白蛋白、转铁蛋白、凝血酶原;在AxC大鼠中可以成瘤。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCC-1588细胞、MPC5细胞、SW-527细胞
IGR-OV1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1734细胞、EMT6细胞、MFD-1细胞
CHO K1 Cells;背景说明:1957年,PuckTT从成年中国仓鼠卵巢的活检组织建立了CHO细胞,CHO-K1是CHO的一个亚克隆。CHO-K1的生长需要脯酸。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CAL 78细胞、NCIH1623细胞、NCI-H82细胞
T-84 Cells;背景说明:T84细胞株是从一位72岁男性结肠癌患者的肺转移灶建立的可移植人类癌细胞株。 肿瘤组织皮下接种于BALB/c裸鼠,并连续进行移植。 [26072] 在裸鼠身上的移植过程中,细胞株始终保持结肠癌的原始组织性状。 [26072] 在无胸腺小鼠中传代23代后建立了T84细胞株。 这些细胞单层生长到饱和并在接触细胞间展现出紧密连接和桥粒。 [1155] 有很多关于多肽类激素和神经递质并维持定向电解质传输的受体。 [1155] 这株细胞展现了接触细胞中的紧密连接和桥粒。 [1155] 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2-1:4传代;每周2次。;生长特性:贴壁生长;形态特性:上皮细胞,多角;相关产品有:SK-RC 39细胞、HCC1500细胞、BHP10-3细胞
159PT Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H2122细胞、SuDHL 8细胞、CCD-1112sk细胞
11G1 Cells(提供STR鉴定图谱)
H-1048 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代;;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:3T3-442A细胞、C 6细胞、OC316细胞
H.Ep. #2 Cells;背景说明:最初认为这个细胞源自喉上皮癌,但随后通过同功酶分析、HeLa标记染色体和DNA指纹分析发现,起源细胞已被HeLa污染。 角蛋白免疫过氧化物酶染色阳性。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H1792细胞、Hs 739.T细胞、COV-362细胞
CSQT-2 Cells;背景说明:肝癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Blotchy fibroblast-11细胞、COR-L88细胞、BT-474细胞
MGHU3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MOLT.4细胞、WM-115细胞、NCIH1963细胞
Hs-852-T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:U-343MG细胞、H-295R细胞、ACC2细胞
KM12-SM Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:143B TK-细胞、Potorous tridactylus Kidney 2细胞、HLFa细胞
C-28I2 Cells;背景说明:软骨;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPPVEC细胞、H-1650细胞、NCI-H1993细胞
NCI-H378 Cells;背景说明:小细胞肺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WRL-68细胞、RC-4B细胞、KHYG细胞
GM13126 Cells(提供STR鉴定图谱)
HAP1 FBXW11 (-) 1 Cells(提供STR鉴定图谱)
DK-MG Cells;背景说明:胶质母细胞瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CAL-12T细胞、NRCC细胞、OCI-LY-18细胞
KYSE-180 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SUM190细胞、PC-2 [Human pancreatic carcinoma]细胞、SKNBE(1)细胞
SCC4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:COLO 201细胞、SMA 560细胞、Pt K2细胞
WM 451-Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC2185细胞、H-716细胞、MKN 45细胞
SK RC 52 Cells;背景说明:肾癌;纵隔膜转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RCC-10细胞、NCI-BL2141细胞、H-322细胞
IGR-OV1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H1734细胞、EMT6细胞、MFD-1细胞
Bac 1.2F5 Cells;背景说明:巨噬细胞;SV40转化;BALB/c x A.CA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:130-T细胞、K422细胞、NTERA-2cl.D1细胞
CT26WT Cells;背景说明:CT26细胞是被N-亚硝基-N-甲基脲烷(NNMU)诱导得到的未分化的小鼠结肠癌细胞,该细胞的一个克隆形成的细胞系被命名为CT26.WT。CT26.WT被逆转录病毒载体LXSN稳定转化形成了一个致死性的亚克隆CT26.CL25,这一病毒载体含有lacZ基因、编码肿瘤相关抗原(TAA)和beta半乳糖苷酶。CT26.WT和CT26.CL25细胞在小鼠中生长速度和致死率都很相似,不同的是CT26.CL25细胞可以表达肿瘤相关抗原和beta半乳糖苷酶,因此这两株细胞可以联合用于免疫治疗和宿主免疫反应的研究。 ;传代方法:1:4-1:10传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:RAW-264.7细胞、hRMECs细胞、P3JHR-1细胞
HL-60/RS Cells(提供STR鉴定图谱)
iPSC-MHF2 c1 Cells(提供STR鉴定图谱)
MCF 10A-H2B-mCherry Cells(提供STR鉴定图谱)
ND12127 Cells(提供STR鉴定图谱)
PoBf Cells(提供STR鉴定图谱)
UCSD167i-99-1 Cells(提供STR鉴定图谱)
ZtA6-9 Cells(提供STR鉴定图谱)
HAP1 USP1 (-) 4 Cells(提供STR鉴定图谱)
COLO 741 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCI-H820细胞、hs 68细胞、LA-795细胞
NCI-H865 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SU-DHL-6细胞、AZ-521细胞、SU-DHL-2细胞
COLO680N Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CCC-HSF-1细胞、SNU182细胞、LS411N细胞
Hs695 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NB4细胞、C-33-A细胞、M109细胞
SW 1573 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Sp 2817细胞、CBRH7919细胞、MDA231细胞
SKMEL24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:32D clone 3细胞、UMNSAH/DF-1细胞、RGC6细胞
OLN93 Cells;背景说明:胶质细胞;自发永生;Wistar;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Medical Research Council cell strain-5细胞、Panc 02.03细胞、SCC 15细胞
MDCK II Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,3-4天传1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SC1细胞、G-292细胞、130 T细胞
MV3 Cells;背景说明:黑色素瘤;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TE7细胞、IHH-4细胞、RD-2细胞
DU 4475 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2—3次;生长特性:悬浮,多细胞聚集;形态特性:上皮细胞样;相关产品有:WRL68细胞、32D.cl3细胞、K562/ADR细胞
SK-MEL-1 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在1×105-2×105,每周补液2-3次。;生长特性:悬浮生长;形态特性:球形的;相关产品有:BV173细胞、ACC-M细胞、MDA-436细胞
SKGT4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BEND细胞、HCC15细胞、CCRF SB细胞
M-07e Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:KP-2细胞、TGW-nu-1细胞、NCI-H2108细胞
130 T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:H2106细胞、3396细胞、MDAMB231细胞
NWA Cells;背景说明:这株细胞有EB病毒基因组。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NCI H2106细胞、OCI AML3细胞、Natural Killer-92细胞
SHL Cells(提供STR鉴定图谱)
B16 melanoma Cells;背景说明:该细胞源于C57BL/6J小鼠黑色素瘤,可以产生黑色素,同基因小鼠体内移植可成瘤;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:梭形;相关产品有:As-PC1细胞、End1/E6E7细胞、H889细胞
CHO cell clone K1 Cells;背景说明:1957年,PuckTT从成年中国仓鼠卵巢的活检组织建立了CHO细胞,CHO-K1是CHO的一个亚克隆。CHO-K1的生长需要脯酸。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCC-95细胞、SKNSH细胞、HCC9724细胞
PE/CA-PJ34 (clone C12) Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MOLT 4细胞、Swiss-3T3细胞、H-2087细胞
HT 1197 Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代,2天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:RFL 6细胞、HEK293-FT细胞、IALM细胞
MDAMB175 Cells;背景说明:该细胞源自一位54岁患有乳腺导管癌白人女性的胸腔积液。;传代方法:1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:FCCH1018细胞、Reuber H-35细胞、H292细胞
B16F1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:870细胞、IPLB-Sf21细胞、BALB/3T3 cl. A31细胞
U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱
NCTC 3960 Cells;背景说明:黑色素瘤;雄性;DBA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MD Anderson-Metastatic Breast-157细胞、HOS/MNNG细胞、JAR细胞
Stanford University-Diffuse Histiocytic Lymphoma-10 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:NHEK细胞、T-HEECs细胞、SV40-MES13细胞
H-2052 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:SUM149-PT细胞、SCL II细胞、Hs 863.T细胞
RPMI-8226 Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Human Epithelioma-2细胞、P31/Fujioka细胞、X63.Ag8.653细胞
OCILY3 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Madison细胞、T-CAM2细胞、SK-MEL-1细胞
B-95-8 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:L-cell细胞、PK15细胞、SR细胞
FU-MMT-1 Cells;背景说明:子宫肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L-M (TK-)细胞、OACP4C细胞、T98细胞
MGc80-3 Cells;背景说明:1980年建系,人胃癌低分化黏液腺癌组织小块用RPMI-1640培养液培养4天细胞开始生长,首次传代8日。免疫抑制的Wistar雄幼大鼠皮下移植成功。;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:ABC1细胞、WML2细胞、V 79细胞
BayGenomics ES cell line GST126 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XB224 Cells(提供STR鉴定图谱)
CGMCC-13827 Cells(提供STR鉴定图谱)
MANDM4-9H9 Cells(提供STR鉴定图谱)
Royan N45 Cells(提供STR鉴定图谱)
TRL 13-1 Cells(提供STR鉴定图谱)
" "PubMed=7017212; DOI=10.1093/jnci/66.6.1003
Pollack M.S., Heagney S.D., Livingston P.O., Fogh J.
HLA-A, B, C and DR alloantigen expression on forty-six cultured human tumor cell lines.
J. Natl. Cancer Inst. 66:1003-1012(1981)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=2823272; DOI=10.1073/pnas.84.21.7716; PMCID=PMC299371
Masuda H., Miller C., Koeffler H.P., Battifora H.A., Cline M.J.
Rearrangement of the p53 gene in human osteogenic sarcomas.
Proc. Natl. Acad. Sci. U.S.A. 84:7716-7719(1987)
PubMed=2233717; DOI=10.1128/mcb.10.11.5772-5781.1990; PMCID=PMC361354
Diller L., Kassel J., Nelson C.E., Gryka M.A., Litwak G., Gebhardt M., Bressac B., Ozturk M., Baker S.J., Vogelstein B., Friend S.H.
p53 functions as a cell cycle control protein in osteosarcomas.
Mol. Cell. Biol. 10:5772-5781(1990)
PubMed=8275086; DOI=10.1038/ng1193-230
Galili N., Davis R.J., Fredericks W.J., Mukhopadhyay S., Rauscher F.J. 3rd, Emanuel B.S., Rovera G., Barr F.G.
Fusion of a fork head domain gene to PAX3 in the solid tumour alveolar rhabdomyosarcoma.
Nat. Genet. 5:230-235(1993)
PubMed=8040301; DOI=10.1172/JCI117360; PMCID=PMC295111
Giovannini M., Biegel J.A., Serra M., Wang J.-Y., Wei Y.-L.H., Nycum L.M., Emanuel B.S., Evans G.A.
EWS-erg and EWS-Fli1 fusion transcripts in Ewing's sarcoma and primitive neuroectodermal tumors with variant translocations.
J. Clin. Invest. 94:489-496(1994)
PubMed=8617485; DOI=10.1016/S0046-8177(96)90115-X
Scotlandi K., Serra M., Manara M.C., Benini S., Sarti M., Maurici D., Lollini P.-L., Picci P., Bertoni F., Baldini N.
Immunostaining of the p30/32MIC2 antigen and molecular detection of EWS rearrangements for the diagnosis of Ewing's sarcoma and peripheral neuroectodermal tumor.
Hum. Pathol. 27:408-416(1996)
PubMed=12645653; DOI=10.1016/S0165-4608(02)00685-4
Ozaki T., Neumann T., Wai D.H., Schaefer K.-L., van Valen F., Lindner N., Scheel C., Boecker W., Winkelmann W., Dockhorn-Dworniczak B., Horst J., Poremba C.
Chromosomal alterations in osteosarcoma cell lines revealed by comparative genomic hybridization and multicolor karyotyping.
Cancer Genet. Cytogenet. 140:145-152(2003)
PubMed=15150091; DOI=10.1158/0008-5472.CAN-03-0809
Schaefer K.-L., Brachwitz K., Wai D.H., Braun Y., Diallo R., Korsching E., Eisenacher M., Voss R., van Valen F., Baer C., Selle B., Spahn L., Liao S.-K., Lee K.A.W., Hogendoorn P.C.W., Reifenberger G., Gabbert H.E., Poremba C.
Expression profiling of t(12;22) positive clear cell sarcoma of soft tissue cell lines reveals characteristic up-regulation of potential new marker genes including ERBB3.
Cancer Res. 64:3395-3405(2004)
PubMed=15736406
Pautke C., Schieker M., Tischer T., Kolk A., Neth P., Mutschler W., Milz S.
Characterization of osteosarcoma cell lines MG-63, Saos-2 and U-2 OS in comparison to human osteoblasts.
Anticancer Res. 24:3743-3748(2004)
PubMed=17354236; DOI=10.1002/ijc.22643
Muller C.R., Paulsen E.B., Noordhuis P., Pedeutour F., Saeter G., Myklebost O.
Potential for treatment of liposarcomas with the MDM2 antagonist Nutlin-3A.
Int. J. Cancer 121:199-205(2007)
PubMed=17981215; DOI=10.1016/j.cancergencyto.2007.08.003
Selvarajah S., Yoshimoto M., Maire G., Paderova J., Bayani J., Squire J.A., Zielenska M.
Identification of cryptic microaberrations in osteosarcoma by high-definition oligonucleotide array comparative genomic hybridization.
Cancer Genet. Cytogenet. 179:52-61(2007)
PubMed=18359981
Niforou K.N., Anagnostopoulos A.K., Vougas K., Kittas C., Gorgoulis V.G., Tsangaris G.T.
The proteome profile of the human osteosarcoma U2OS cell line.
Cancer Genomics Proteomics 5:63-78(2008)
PubMed=19787792; DOI=10.1002/gcc.20717
Ottaviano L., Schaefer K.-L., Gajewski M., Huckenbeck W., Baldus S.E., Rogel U., Mackintosh C., de Alava E., Myklebost O., Kresse S.H., Meza-Zepeda L.A., Serra M., Cleton-Jansen A.-M., Hogendoorn P.C.W., Buerger H., Aigner T., Gabbert H.E., Poremba C.
Molecular characterization of commonly used cell lines for bone tumor research: a trans-European EuroBoNet effort.
Genes Chromosomes Cancer 49:40-51(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=21519327; DOI=10.1038/labinvest.2011.72
Mohseny A.B., Machado I., Cai Y.-P., Schaefer K.-L., Serra M., Hogendoorn P.C.W., Llombart-Bosch A., Cleton-Jansen A.-M.
Functional characterization of osteosarcoma cell lines provides representative models to study the human disease.
Lab. Invest. 91:1195-1205(2011)
PubMed=22068332; DOI=10.1038/msb.2011.82; PMCID=PMC3261713
Beck M., Schmidt A., Malmstroem J., Claassen M., Ori A., Szymborska A., Herzog F., Rinner O., Ellenberg J., Aebersold R.
The quantitative proteome of a human cell line.
Mol. Syst. Biol. 7:549-549(2011)
PubMed=22278370; DOI=10.1074/mcp.M111.014050; PMCID=PMC3316730
Geiger T., Wehner A., Schaab C., Cox J., Mann M.
Comparative proteomic analysis of eleven common cell lines reveals ubiquitous but varying expression of most proteins.
Mol. Cell. Proteomics 11:M111.014050-M111.014050(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23144859; DOI=10.1371/journal.pone.0048262; PMCID=PMC3492335
Kresse S.H., Rydbeck H., Skarn M., Namlos H.M., Barragan-Polania A.H., Cleton-Jansen A.-M., Serra M., Liestol K., Hogendoorn P.C.W., Hovig E., Myklebost O., Meza-Zepeda L.A.
Integrative analysis reveals relationships of genetic and epigenetic alterations in osteosarcoma.
PLoS ONE 7:E48262-E48262(2012)
PubMed=23649806; DOI=10.1083/jcb.201210031; PMCID=PMC3653305
Kleiblova P., Shaltiel I.A., Benada J., Sevcik J., Pechackova S., Pohlreich P., Voest E.E., Dundr P., Bartek J., Kleibl Z., Medema R.H., Macurek L.
Gain-of-function mutations of PPM1D/Wip1 impair the p53-dependent G1 checkpoint.
J. Cell Biol. 201:511-521(2013)
PubMed=24136885; DOI=10.1002/pbc.24801; PMCID=PMC4154381
Guma S.R., Lee D.A., Yu L., Gordon N., Hughes D., Stewart J., Wang W.-L., Kleinerman E.S.
Natural killer cell therapy and aerosol interleukin-2 for the treatment of osteosarcoma lung metastasis.
Pediatr. Blood Cancer 61:618-626(2014)
PubMed=24618588; DOI=10.1371/journal.pone.0091433; PMCID=PMC3950186
Chernobrovkin A.L., Zubarev R.A.
Detection of viral proteins in human cells lines by xeno-proteomics: elimination of the last valid excuse for not testing every cellular proteome dataset for viral proteins.
PLoS ONE 9:E91433-E91433(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25894527; DOI=10.1371/journal.pone.0121314; PMCID=PMC4404347
Bausch-Fluck D., Hofmann A., Bock T., Frei A.P., Cerciello F., Jacobs A., Moest H., Omasits U., Gundry R.L., Yoon C., Schiess R., Schmidt A., Mirkowska P., Hartlova A.S., Van Eyk J.E., Bourquin J.-P., Aebersold R., Boheler K.R., Zandstra P.W., Wollscheid B.
A mass spectrometric-derived cell surface protein atlas.
PLoS ONE 10:E0121314-E0121314(2015)
PubMed=26351324; DOI=10.1158/1535-7163.MCT-15-0074; PMCID=PMC4636476
Teicher B.A., Polley E.C., Kunkel M., Evans D., Silvers T.E., Delosh R.M., Laudeman J., Ogle C., Reinhart R., Selby M., Connelly J., Harris E., Monks A., Morris J.
Sarcoma cell line screen of oncology drugs and investigational agents identifies patterns associated with gene and microRNA expression.
Mol. Cancer Ther. 14:2452-2462(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29853780; DOI=10.1155/2018/3082526; PMCID=PMC5964616
Salawu A., Wright K., Al-Kathiri A., Wyld L., Reed M.W.R., Sisley K.
Sister chromatid exchange and genomic instability in soft tissue sarcomas: potential implications for response to DNA-damaging treatments.
Sarcoma 2018:3082526.1-3082526.8(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)"
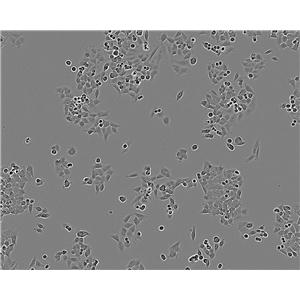





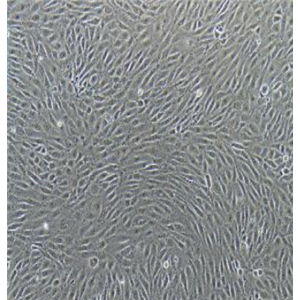
![U-2OS[U2OS]细胞(骨肉瘤细胞STR鉴定正确)LM8C0236](https://img.chemicalbook.com/SupplyImg2/2025-12-01/Large/202512012110008124369.png)
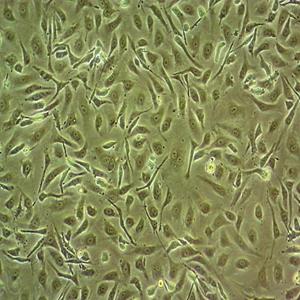

![U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱](/SupplyImg1/2025-02-01/202502011215432874723.jpg)
![U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱](/SupplyImg1/2024-12-26/202412261010053678328.jpg)
![U-2 OS[U2OS]人骨肉瘤细胞代次低|培养基|送STR图谱](/SupplyImg1/2024-12-26/202412261010056126100.jpg)
