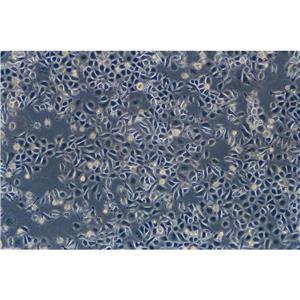
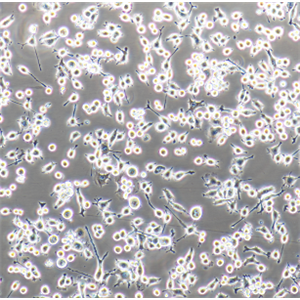
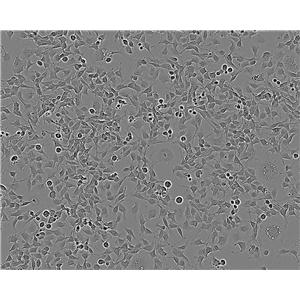
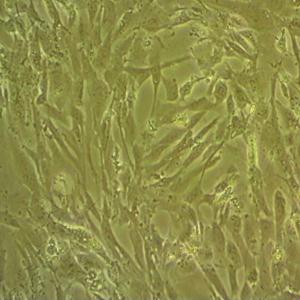
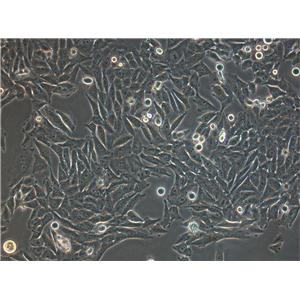
"PANC-1人胰腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
WEHI3B Cells;背景说明:白血病;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:OS-732细胞、HepG2/C3A细胞、Madison细胞
HPMEC Cells;背景说明:肺微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hela-mock细胞、GEO细胞、FAK+/+细胞
LN-229 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:6传代;每周换液2-3次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:RGCs细胞、786-0细胞、hSCC-25细胞
PANC-1人胰腺癌细胞代次低|培养基|送STR图谱
背景信息:这株人胰腺癌细胞株源自于胰腺癌导管细胞,其倍增时间为52小时。染色体研究表明,该细胞染色体众数为63,包括3个独特标记的染色体和1个小环状染色体。该细胞的生长可被1unit/ml的左旋天冬酰胺酶抑制;能在软琼脂上生长;能在裸鼠上成瘤。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
DSMZ菌株保藏中心成立于1969年,是德国的国家菌种保藏中心。该中心一直致力于细菌、真菌、质粒、抗菌素、人体和动物细胞、植物病毒等的分类、鉴定和保藏工作。DSMZ菌种保藏中心是欧洲规模最大的生物资源中心,保藏有动物细胞500多株。Riken BRC成立于1920年,是英国的国家菌种保藏中心。该中心一直致力于细菌、真菌、植物病毒等的分类、鉴定和保藏工作。日本Riken BRC(Riken生物资源保藏中心)是全球三大典型培养物收集中心之一。Riken保藏中心提供了很多细胞系。在世界范围内,这些细胞系,都在医学、科学和兽医中具有重要意义。Riken生物资源中心支持了各种学术、健康、食品和兽医机构的研究工作,并在世界各地不同组织的微生物实验室和研究机构中使用。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
PANC-1人胰腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
MOLT4 Cells;背景说明:MOLT-4与MOLT-3来源于一名19岁的男性急性淋巴细胞性白血病的复发患者,该患者前期接受过多种药物联合化疗。MOLT-4细胞系为T淋巴细胞起源,p53基因的第248位密码子有一个G→A突变,不表达p53,不表达免疫球蛋白或EB病毒;可产生高水平的末端脱氧核糖转移酶;表达CD1(49%),CD2(35%),CD3A(26%)B(33%)C(34%),CD4(55%),CD5(72%),CD6(22%),CD7(77%)。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;圆形;相关产品有:HME-1细胞、VeroE6细胞、Medical University of Graz-Chordoma 1细胞
BSC-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COV 504细胞、Tn5B1-4细胞、RBMVEC细胞
HNE-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SK ES 01细胞、SK 1细胞、HOS-MNNG细胞
LM3 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HCC1500细胞、UCLA-SO-M14细胞、H-35细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞复苏相关注意事项:1.取细胞的过程中注意带HAO防冻手套,护目镜。此项尤为重要,细胞冻存管可能漏入,解冻时冻存管中的气温急剧上升,可导致爆炸。2.冻存的问题:冻存的配置已是常识,在这里不作详述,但二甲基亚砜(DMSO )对细胞不是完全无毒副作用,在常温下,二甲基亚砜对细胞的毒副作较大,因此,必须在1-2min内使冻存完全融化。如果复苏温度太慢,会造成细胞的损伤,二甲基亚砜(DMSO)ZuiHAO选择进口产品。3.离心前须加入少量培养。细胞解冻后二甲基亚砜浓度较GAO,注意加入少量培养可稀释其浓度,以减少对细胞的损伤。4.离心问题:目前主要有两种见解。一种是解冻后的细胞悬直接吹打均匀后分装到培养瓶中进行培养,第二天换。因为离心的目的是两个,去除DMSO,去除死细胞,这个是标准流程,但对一般人来说,把握不HAO离心转速和时间,转的不够活细胞沉底的少,细胞就全被扔掉了,转过了活细胞会受压过大,死亡。此外在操作过程中容易污染,所以不推荐。另一种说法为细胞悬中含有二甲基亚砜(DMSO),DMSO对细胞有一定的毒副作用,所以须将离心后的体前倒净,且一定倒干净。我在试验中按照常规的离心分装的方法进行复苏,结果无异常。5.细胞贴壁少的问题:教科书中说明冻存细胞解冻时1ml细胞要加10ml-15ml培养,而在我的试验中的经验总结为培养基越少细胞越容易贴附。6.复苏细胞分装的问题:试验中我的经验总结为复苏1管细胞一般可分装到1-2只培养瓶中,分装过多,细胞浓度过低,不利于细胞的贴壁。7.加培养基的量放入问题:这个量的多少的把握主要涉及到的问题DMSO的浓度,从如果你加培养基的太少,那么DMSO的浓度就会比较大,就会影响细胞生长,从以前的资料来看,DMSO的浓度在小于0.5%的时候对一般细胞没有什么影响,还有一个说法是1%。所以如果你的冻存的浓度是10%DMSO的话那么加10ml以上的培养基就恰HAO稀释到了无害浓度。
LWnt3A Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H2195细胞、NCI-H2330细胞、SGC-7901细胞
LS1034 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,每周2-3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:NRK-52E细胞、NCL-H548细胞、SK-N-F1细胞
MDA 435 Cells;背景说明:乳腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SP2细胞、A-375.S2细胞、T47D细胞
NB9 Cells;背景说明:详见相关文献介绍;传代方法:1:10 1:50每2 - 3周;每周换液2-3次。;生长特性:贴壁生长;形态特性:成神经细胞;相关产品有:WM451细胞、A-9细胞、MPC11细胞
C-32 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:EST81细胞、Cloudman M3细胞、UK Pan-1细胞
alphaTC1 Clone 6 Cells;背景说明:胰岛素瘤;a细胞;C57BL/6xDBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CCRF.CEM细胞、HBE135-E6E7细胞、ZR-75-1细胞
H-747 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HPC-Y5细胞、H727细胞、VMRC-LCD细胞
373 MG Cells;背景说明:胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:OEC19细胞、MAVER1细胞、NCIH295R细胞
Mac-1 Cells;背景说明:皮肤T淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U87细胞、SNU761细胞、3396细胞
NB1RGB Cells;背景说明:皮肤;成纤维细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-MEL-2-LUC细胞、H-1819细胞、SHIN-3细胞
D341Med Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮生长;形态特性:髓母细胞样;相关产品有:NCI-H2405细胞、U20S细胞、MDA-MB-157细胞
Panc_04_03 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:YD38细胞、J 111细胞、SNU-739细胞
FHs74Int Cells;背景说明:小肠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:D341细胞、KP-N-RT-BM细胞、CAL85-1细胞
769P Cells;背景说明:该细胞系1975年建系,源自一位63岁白人女性的初期透明细胞腺癌组织,细胞呈圆形且边界不清,核浆比大,有微绒毛及桥粒。该细胞可在软琼脂上生长。 ;传代方法:1:4—1:12传代,2—3天换液一次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:AHH1细胞、AU-565细胞、H-1770细胞
My-La 2059 Cells;背景说明:皮肤;T淋巴细胞瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:U-266 AR1细胞、H345细胞、MIMVEC细胞
AAV-293 Cells;背景说明:我们推荐使用AAV-293细胞株繁殖腺病毒相关重组病毒。 AAV-293源自普遍使用的 HEK293细胞株,但产生的病毒滴度更高。 HEK293细胞是剪切过的腺病毒5型DNA转染的人胚肾细胞。 跟HEK293细胞一样,AAV-293细胞反式表达腺病毒E1基因,当共转染三个AAV助质粒(一个含ITR的质粒,pAAV-RC, 和E1缺失助质粒)时,可以产生有感染力的腺病毒-相关病毒颗粒。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:P19细胞、A1847细胞、HECV细胞
ECC-12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RCC10细胞、SCL1细胞、Lec1细胞
13008 Cells(提供STR鉴定图谱)
Abcam HeLa CDKN1A KO Cells(提供STR鉴定图谱)
AG15898 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRE275 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG125 Cells(提供STR鉴定图谱)
C0024 Cells(提供STR鉴定图谱)
CW10005 Cells(提供STR鉴定图谱)
DA05560 Cells(提供STR鉴定图谱)
GHOST(3).CCR1 Cells(提供STR鉴定图谱)
B16-F10 Cells;背景说明:B16-F10是B16-F0的亚系。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Sp2/mIL-6细胞、HEK293-A细胞、EU-4细胞
PANC-1人胰腺癌细胞代次低|培养基|送STR图谱
Mv.1.Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:YAC细胞、BC-027细胞、LICCF细胞
Sol8 Cells;背景说明:骨骼肌;C3H;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RAW2647细胞、Oregon J-111细胞、U-118MG细胞
253J B-V Cells;背景说明:膀胱癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MNNG/HOS Cl #5细胞、HEC-1A细胞、H838细胞
H-2291 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:SuDHL 4细胞、BCaP-37细胞、HeLa/DDP细胞
LNCaP-Clone-FGC Cells;背景说明:人前列腺癌细胞LNCaP克隆FGC是从一位50岁白人男性(血型B+)的左锁骨淋巴结针刺活检中分离,该患者经确诊为前列腺癌转移。 这株细胞对5-α-二睾酮(生长调节子和酸性脂酶产物)有响应。这株细胞并不形成一致的单层,而是形成集落,在传代时可以用滴管反复吹吸打碎。它们仅仅轻轻地吸附在基底上,不形成汇合,很快使培养基变酸。生长很慢。传代后48小时内不应扰动。当培养瓶封包后,多数细胞从培养瓶底分离,悬浮在培养基中。收到后,在通常培养单层细胞的条件下培养24到48小时,以合细胞再贴壁。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:H1954细胞、3T6 Swiss Albino细胞、Tj-905细胞
1H7G10D10F7 Cells(提供STR鉴定图谱)
HNEpC Cells;背景说明:鼻粘膜;上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Jurkat-77细胞、M-NFS-60细胞、C4-I细胞
H2085 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代 ;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Epstein-Barr-1细胞、MC3T3, E1 subgroup-4 clone细胞、H1618细胞
WEHI-3B Cells;背景说明:白血病;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Okayama University Medical School-23细胞、H1385细胞、GA-10-Clone-4细胞
NK-92MI Cells;背景说明:NK细胞;淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SLMT1细胞、SUPB-15细胞、NCI-SNU-407细胞
SK-MEL24 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:星形的;相关产品有:IGROV 1细胞、EFM192B细胞、COLO-1细胞
NCIH847 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SUPM2细胞、Pa16C细胞、H23细胞
SW-403 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:6传代,每周换液2-3次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:TF-1a细胞、HSC5细胞、Lewis-Lung细胞
BT-B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:HET1A细胞、Eph4 1424细胞、G-292 clone A141B1细胞
HAP1 APOBEC3A (-) 2 Cells(提供STR鉴定图谱)
HAP1 SLC26A2 (-) 2 Cells(提供STR鉴定图谱)
TE8 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:RASMC细胞、NKM-1细胞、PLA-801D细胞
H446 Cells;背景说明:该细胞是1982年由CarneyD和GazdarAF等从一位小细胞肺癌患者的胸腔积液中建立的。细胞的原始形态并不具有小细胞肺癌特征。这个细胞株是小细胞肺癌的生化和形态学上的变种,表达神经元特有的烯醇酶和脑型肌酸激酶同工酶;左旋多巴脱羧酶、蚕素、抗利尿激素、催产素或胃泌激素释放肽未达到可检测水平。与正常细胞相比,该细胞c-mycDNA序列扩增约20倍,RNA增加15倍。最初传代培养基用含有5%FBS的RPMI1640,另外添加10nM化可的松、0.005mg/ml胰岛素、0.01mg/ml转铁;传代方法:1:2传代;生长特性:贴壁/悬浮生长,混合;形态特性:上皮样;相关产品有:SUIT 2细胞、OV-CA 432细胞、SF 767细胞
A-375 Cells;背景说明:A375源自一位54岁女性,是Giard DJ等人建立的一系列细胞株中的一株。该细胞可在免疫抑制小鼠上成瘤,在琼脂上形成克隆。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:H647ell细胞、A.704细胞、Hs683T细胞
COR L279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LAN-1细胞、BLO 11细胞、MDA-157细胞
T_T_ Cells;背景说明:详见相关文献介绍;传代方法:10^5 cells/60mm dish;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:BN-CL2细胞、NCIH82细胞、LP1细胞
MDCK-2 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,3-4天传1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Ku812F细胞、HS578T细胞、Swiss/3T3细胞
CHG5 Cells;背景说明:少突神经胶质瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:BE2-M17细胞、OCI-Ly1细胞、UCLA-SO-M20细胞
BEL 7404 Cells;背景说明:用Northernblot方法,未能检测到细胞中1.3kbLFIRE-1/HFREP-1mRNA的表达。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Colo205细胞、JiyoyeP-2003细胞、HIMEC细胞
IAR 2-28 Cells(提供STR鉴定图谱)
LG-1 Cells(提供STR鉴定图谱)
NBCCS-1AW Cells(提供STR鉴定图谱)
PACS1001i-GM27160 Cells(提供STR鉴定图谱)
S004925 Cells(提供STR鉴定图谱)
Ubigene A-549 INSR KO Cells(提供STR鉴定图谱)
UQi001-A Cells(提供STR鉴定图谱)
HAP1 STIM1 (-) 4 Cells(提供STR鉴定图谱)
IOSE 80 Cells;背景说明:卵巢;上皮细胞;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2330细胞、NALM-6细胞、FHC细胞
Fetal Rhesus Kidney-4 Cells;背景说明:胚胎;肾;自发永生;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IGR-OV1细胞、HOS TE85细胞、LAN-6细胞
KP4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NS653细胞、F9细胞、VP229细胞
FHs 74 Int Cells;背景说明:小肠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KPL1细胞、H-510细胞、HEC-251细胞
NCI/ADR-RES Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FM-88细胞、DC 2.4细胞、L-cell细胞
NCI/ADR-RES Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FM-88细胞、DC 2.4细胞、L-cell细胞
Hs739T Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:混合型;相关产品有:OCI-Ly 7细胞、B-cell with DC Morphology细胞、H-2171细胞
MD Anderson-Metastatic Breast-468 Cells;背景说明:该细胞是1977年由CailleauR等从一位患有转移性乳腺癌的51岁黑人女性的胸腔积液中分离得到的。虽然供体组织的G6PD等位基因杂合,但此细胞株始终表现为G6PDA表型。P53基因273位密码子存在G→A突变,从而导致Arg→His替代。每个细胞上存在1×106个EGF受体。;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:YH-13细胞、SLK细胞、NCIH1869细胞
MOPC Cells;背景说明:少突胶质前体 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:HS-294细胞、GM3570细胞、C57 Mouse Tumor 64细胞
GTL-16 Cells;背景说明:胃癌;肝转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H35 Reuber细胞、SNG-M细胞、Colo205细胞
NCI-841 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:5传代,;生长特性:混合型生长;形态特性:详见产品说明书;相关产品有:AML-2细胞、Rat1细胞、LS180细胞
NCI-H1238 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1668细胞、BAC1.2F5细胞、SKMEL-31细胞
HT 1376 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:PG-4(S+L-)细胞、Panc-327细胞、NCIH1993细胞
SF-767 Cells;背景说明:脑瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SNU251细胞、IOSE-Mar细胞、CT 26细胞
LC-1-sq Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RH35细胞、P30OHK细胞、Panc_03_27细胞
THP1-Dual KI-hSTING-R232 Cells(提供STR鉴定图谱)
JROECL19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:LC1/Sq细胞、U118细胞、BT483细胞
M-14 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代;生长特性:混合生长;形态特性:详见产品说明书;相关产品有:SW626细胞、NIH 3T6细胞、BNL-CL.2细胞
Hs 578Bst Cells;背景说明:乳腺 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCC1171细胞、Tu686细胞、J82细胞
GC1-SPG Cells;背景说明:精原细胞;SV40转化;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SaOS-2细胞、SUM149-PT细胞、H-1048细胞
NCI-H522 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SJCRH30细胞、RIN-m 14B细胞、H2170细胞
B16 Cells;背景说明:该细胞源于C57BL/6J小鼠黑色素瘤,可以产生黑色素,同基因小鼠体内移植可成瘤;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:梭形;相关产品有:Panc-08.13细胞、CCD33Co细胞、Panc-8_13细胞
PANC-1人胰腺癌细胞代次低|培养基|送STR图谱
SCC15 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H1092细胞、NCI-H1694细胞、Colo699细胞
Hepatoma-22 Cells;背景说明:1952年,前苏联医学科学院肿瘤研究所以C3HA小鼠诱发的H22肝癌实体瘤的瘤细胞悬液,昆明种小鼠皮下移植后,转腹水瘤。经检测,该瘤株在Km小鼠、615小鼠、C57BL/6小鼠、BALB/C小鼠体内可以形成实体瘤和腹水瘤。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:淋巴母细胞;相关产品有:NCI.H522细胞、Colo320细胞、B-104细胞
SKNFI Cells;背景说明:详见相关文献介绍;传代方法:1:4传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Acanthosis Nigricans 3rd attempt-CArcinoma细胞、NCI-H1155细胞、WBF344细胞
SNG-M Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:多边形;相关产品有:SKMEL5细胞、PIEC细胞、CNE-1细胞
H2122 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:淋巴母细胞;相关产品有:HTR-8/SV-neo细胞、ANA1细胞、Mel-RM细胞
Bx-PC3 Cells;背景说明:这个细胞株不表达囊肿性纤维化跨膜电导调节子(CFTR)。CFTR阳性的细胞株是Capan-1(ATCCHTB-79)。;传代方法:消化5-10分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Hs832T细胞、B95.8细胞、MX-1细胞
NCIH2286 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:椭圆形;相关产品有:CORL105细胞、SNT8细胞、Y3M细胞
NCI-H1563 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H4-II-EC3细胞、ANA-1细胞、KYSE-410细胞
BayGenomics ES cell line RRB035 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XE323 Cells(提供STR鉴定图谱)
CT43 Cells(提供STR鉴定图谱)
mGC5 Cells(提供STR鉴定图谱)
Sp2/3-3 Cells(提供STR鉴定图谱)
LRG Cells(提供STR鉴定图谱)
" "PubMed=1630814
Ruggeri B.A., Zhang S.-Y., Caamano J., DiRado M., Flynn S.D., Klein-Szanto A.J.P.
Human pancreatic carcinomas and cell lines reveal frequent and multiple alterations in the p53 and Rb-1 tumor-suppressor genes.
Oncogene 7:1503-1511(1992)
PubMed=7809022; DOI=10.1097/00006676-199409000-00018
Sumi S., Beauchamp R.D., Townsend C.M. Jr., Pour P.M., Ishizuka J., Thompson J.C.
Lovastatin inhibits pancreatic cancer growth regardless of RAS mutation.
Pancreas 9:657-661(1994)
PubMed=7961102; DOI=10.1111/j.1349-7006.1994.tb02898.x; PMCID=PMC5919355
Suwa H., Yoshimura T., Yamaguchi N., Kanehira K., Manabe T., Imamura M., Hiai H., Fukumoto M.
K-ras and p53 alterations in genomic DNA and transcripts of human pancreatic adenocarcinoma cell lines.
Jpn. J. Cancer Res. 85:1005-1014(1994)
PubMed=8026879; DOI=10.1002/ijc.2910580207
Berrozpe G., Schaeffer J., Peinado M.A., Real F.X., Perucho M.
Comparative analysis of mutations in the p53 and K-ras genes in pancreatic cancer.
Int. J. Cancer 58:185-191(1994)
PubMed=8194712; DOI=10.1016/0016-5085(94)90422-7
Simon B., Weinel R., Hohne M., Watz J., Schmidt J., Kortner G., Arnold R.
Frequent alterations of the tumor suppressor genes p53 and DCC in human pancreatic carcinoma.
Gastroenterology 106:1645-1651(1994)
PubMed=8286197; DOI=10.1038/bjc.1994.24; PMCID=PMC1968784
Lohr J.-M., Trautmann B., Gottler M., Peters S., Zauner I., Maillet B., Kloppel G.
Human ductal adenocarcinomas of the pancreas express extracellular matrix proteins.
Br. J. Cancer 69:144-151(1994)
PubMed=21607521; DOI=10.3892/or.1.6.1223
Iguchi H., Morita R., Yasuda D., Takayanagi R., Ikeda Y., Takada Y., Shimazoe T., Nawata H., Kono A.
Alterations of the p53 tumor-suppressor gene and ki-ras oncogene in human pancreatic cancer-derived cell-lines with different metastatic potential.
Oncol. Rep. 1:1223-1227(1994)
PubMed=9023415; DOI=10.1006/cimm.1996.1062
Seki N., Hoshino T., Kikuchi M., Hayashi A., Itoh K.
HLA-A locus-restricted and tumor-specific CTLs in tumor-infiltrating lymphocytes of patients with non-small cell lung cancer.
Cell. Immunol. 175:101-110(1997)
PubMed=9788440; DOI=10.1038/sj.onc.1202118
Villanueva A., Garcia C., Paules Blazquez A.B., Vicente M., Megias M., Reyes G., de Villalonga P., Agell N., Lluis F., Bachs O., Capella G.
Disruption of the antiproliferative TGF-beta signaling pathways in human pancreatic cancer cells.
Oncogene 17:1969-1978(1998)
PubMed=10027410; DOI=10.1016/S0002-9440(10)65298-4; PMCID=PMC1850008
Ghadimi B.M., Schrock E., Walker R.L., Wangsa D., Jauho A., Meltzer P.S., Ried T.
Specific chromosomal aberrations and amplification of the AIB1 nuclear receptor coactivator gene in pancreatic carcinomas.
Am. J. Pathol. 154:525-536(1999)
PubMed=11115575; DOI=10.3892/or.8.1.89
Sun C.-L., Yamato T., Furukawa T., Ohnishi Y., Kijima H., Horii A.
Characterization of the mutations of the K-ras, p53, p16, and SMAD4 genes in 15 human pancreatic cancer cell lines.
Oncol. Rep. 8:89-92(2001)
PubMed=11169957; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1014>3.0.CO;2-U
Wallrapp C., Hahnel S., Boeck W., Soder A., Mincheva A., Lichter P., Leder G., Gansauge F., Sorio C., Scarpa A., Gress T.M.
Loss of the Y chromosome is a frequent chromosomal imbalance in pancreatic cancer and allows differentiation to chronic pancreatitis.
Int. J. Cancer 91:340-344(2001)
PubMed=11169959; DOI=10.1002/1097-0215(200002)9999:9999<::AID-IJC1049>3.0.CO;2-C
Sirivatanauksorn V., Sirivatanauksorn Y., Gorman P.A., Davidson J.M., Sheer D., Moore P.S., Scarpa A., Edwards P.A.W., Lemoine N.R.
Non-random chromosomal rearrangements in pancreatic cancer cell lines identified by spectral karyotyping.
Int. J. Cancer 91:350-358(2001)
PubMed=11787853; DOI=10.1007/s004280100474
Moore P.S., Sipos B., Orlandini S., Sorio C., Real F.X., Lemoine N.R., Gress T.M., Bassi C., Kloppel G., Kalthoff H., Ungefroren H., Lohr J.-M., Scarpa A.
Genetic profile of 22 pancreatic carcinoma cell lines. Analysis of K-ras, p53, p16 and DPC4/Smad4.
Virchows Arch. 439:798-802(2001)
PubMed=12692724; DOI=10.1007/s00428-003-0784-4
Sipos B., Moser S., Kalthoff H., Torok V., Lohr J.-M., Kloppel G.
A comprehensive characterization of pancreatic ductal carcinoma cell lines: towards the establishment of an in vitro research platform.
Virchows Arch. 442:444-452(2003)
PubMed=12800145; DOI=10.1002/gcc.10218
Adelaide J., Huang H.-E., Murati A., Alsop A.E., Orsetti B., Mozziconacci M.-J., Popovici C., Ginestier C., Letessier A., Basset C., Courtay-Cahen C., Jacquemier J., Theillet C., Birnbaum D., Edwards P.A.W., Chaffanet M.
A recurrent chromosome translocation breakpoint in breast and pancreatic cancer cell lines targets the neuregulin/NRG1 gene.
Genes Chromosomes Cancer 37:333-345(2003)
PubMed=14695172
Iacobuzio-Donahue C.A., Ashfaq R., Maitra A., Adsay N.V., Shen-Ong G.L.-C., Berg K., Hollingsworth M.A., Cameron J.L., Yeo C.J., Kern S.E., Goggins M.G., Hruban R.H.
Highly expressed genes in pancreatic ductal adenocarcinomas: a comprehensive characterization and comparison of the transcription profiles obtained from three major technologies.
Cancer Res. 63:8614-8622(2003)
PubMed=15126341; DOI=10.1158/0008-5472.CAN-03-3159
Heidenblad M., Schoenmakers E.F.P.M., Jonson T., Gorunova L., Veltman J.A., van Kessel A.G., Hoglund M.
Genome-wide array-based comparative genomic hybridization reveals multiple amplification targets and novel homozygous deletions in pancreatic carcinoma cell lines.
Cancer Res. 64:3052-3059(2004)
PubMed=15367885; DOI=10.1097/00006676-200410000-00004
Loukopoulos P., Kanetaka K., Takamura M., Shibata T., Sakamoto M., Hirohashi S.
Orthotopic transplantation models of pancreatic adenocarcinoma derived from cell lines and primary tumors and displaying varying metastatic activity.
Pancreas 29:193-203(2004)
PubMed=15688027; DOI=10.1038/sj.onc.1208383
Heidenblad M., Lindgren D., Veltman J.A., Jonson T., Mahlamaki E.H., Gorunova L., van Kessel A.G., Schoenmakers E.F.P.M., Hoglund M.
Microarray analyses reveal strong influence of DNA copy number alterations on the transcriptional patterns in pancreatic cancer: implications for the interpretation of genomic amplifications.
Oncogene 24:1794-1801(2005)
PubMed=15770730; DOI=10.3748/wjg.v11.i10.1521; PMCID=PMC4305696
Ma J.-H., Patrut E., Schmidt J., Knaebel H.-P., Buchler M.W., Marten A.
Synergistic effects of interferon-alpha in combination with chemoradiation on human pancreatic adenocarcinoma.
World J. Gastroenterol. 11:1521-1528(2005)
PubMed=16912165; DOI=10.1158/0008-5472.CAN-06-0721
Calhoun E.S., Hucl T., Gallmeier E., West K.M., Arking D.E., Maitra A., Iacobuzio-Donahue C.A., Chakravarti A., Hruban R.H., Kern S.E.
Identifying allelic loss and homozygous deletions in pancreatic cancer without matched normals using high-density single-nucleotide polymorphism arrays.
Cancer Res. 66:7920-7928(2006)
PubMed=18298655; DOI=10.1111/j.1582-4934.2008.00289.x; PMCID=PMC3828895
Pilarsky C., Ammerpohl O., Sipos B., Dahl E., Hartmann A., Wellmann A., Braunschweig T., Lohr J.-M., Jesenofsky R., Friess H., Wente M.N., Kristiansen G., Jahnke B., Denz A., Ruckert F., Schackert H.K., Kloppel G., Kalthoff H., Saeger H.-D., Grutzmann R.
Activation of Wnt signalling in stroma from pancreatic cancer identified by gene expression profiling.
J. Cell. Mol. Med. 12:2823-2835(2008)
PubMed=18380791; DOI=10.1111/j.1349-7006.2008.00779.x; PMCID=PMC11158928
Suzuki A., Shibata T., Shimada Y., Murakami Y., Horii A., Shiratori K., Hirohashi S., Inazawa J., Imoto I.
Identification of SMURF1 as a possible target for 7q21.3-22.1 amplification detected in a pancreatic cancer cell line by in-house array-based comparative genomic hybridization.
Cancer Sci. 99:986-994(2008)
PubMed=18575732; DOI=10.3892/or.20.1.155
Kawaoka T., Oka M., Takashima M., Ueno T., Yamamoto K., Yahara N., Yoshino S., Hazama S.
Adoptive immunotherapy for pancreatic cancer: cytotoxic T lymphocytes stimulated by the MUC1-expressing human pancreatic cancer cell line YPK-1.
Oncol. Rep. 20:155-163(2008)
CLPUB00416
Oberlin L.
Treatment of pancreatic carcinoma cell lines in vitro and vivo with a monoclonal antibody against the transferrin receptor.
Thesis VMD (2009); Justus-Liebig-Universitat Giessen; Giessen; Germany
DOI=10.4172/jpb.1000057
Yamada M., Fujii K., Koyama K., Hirohashi S., Kondo T.
The proteomic profile of pancreatic cancer cell lines corresponding to carcinogenesis and metastasis.
J. Proteomics Bioinformatics 2:1-18(2009)
PubMed=19077451; DOI=10.1159/000178871
Harada T., Chelala C., Crnogorac-Jurcevic T., Lemoine N.R.
Genome-wide analysis of pancreatic cancer using microarray-based techniques.
Pancreatology 9:13-24(2009)
PubMed=20037478; DOI=10.4161/cbt.8.21.9685; PMCID=PMC2824894
Kent O.A., Mullendore M.E., Wentzel E.A., Lopez-Romero P., Tan A.-C., Alvarez H., West K.M., Ochs M.F., Hidalgo M., Arking D.E., Maitra A., Mendell J.T.
A resource for analysis of microRNA expression and function in pancreatic ductal adenocarcinoma cells.
Cancer Biol. Ther. 8:2013-2024(2009)
PubMed=20418756; DOI=10.1097/MPA.0b013e3181c15963; PMCID=PMC2860631
Deer E.L., Gonzalez-Hernandez J., Coursen J.D., Shea J.E., Ngatia J.G., Scaife C.L., Firpo M.A., Mulvihill S.J.
Phenotype and genotype of pancreatic cancer cell lines.
Pancreas 39:425-435(2010)
PubMed=21515691; DOI=10.1074/jbc.M111.226795; PMCID=PMC3121343
McCluskey J.T., Hamid M., Guo-Parke H., McClenaghan N.H., Gomis R., Flatt P.R.
Development and functional characterization of insulin-releasing human pancreatic beta cell lines produced by electrofusion.
J. Biol. Chem. 286:21982-21992(2011)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
PubMed=23325432; DOI=10.1101/gr.147942.112; PMCID=PMC3589544
Varley K.E., Gertz J., Bowling K.M., Parker S.L., Reddy T.E., Pauli-Behn F., Cross M.K., Williams B.A., Stamatoyannopoulos J.A., Crawford G.E., Absher D.M., Wold B.J., Myers R.M.
Dynamic DNA methylation across diverse human cell lines and tissues.
Genome Res. 23:555-567(2013)
PubMed=25167228; DOI=10.1038/bjc.2014.475; PMCID=PMC4453732
Hamidi H., Lu M., Chau K., Anderson L., Fejzo M.S., Ginther C., Linnartz R., Zubel A., Slamon D.J., Finn R.S.
KRAS mutational subtype and copy number predict in vitro response of human pancreatic cancer cell lines to MEK inhibition.
Br. J. Cancer 111:1788-1801(2014)
PubMed=25394408; DOI=10.3892/or.2014.3599
Wang C.-F., Zhang W.-W., Fu M.-J., Yang A.-Q., Huang H.-H., Xie J.-M.
Establishment of human pancreatic cancer gemcitabine-resistant cell line with ribonucleotide reductase overexpression.
Oncol. Rep. 33:383-390(2015)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26216984; DOI=10.1073/pnas.1501605112; PMCID=PMC4538616
Daemen A., Peterson D., Sahu N., McCord R., Du X.-N., Liu B., Kowanetz K., Hong R., Moffat J., Gao M., Boudreau A., Mroue R., Corson L., O'Brien T., Qing J., Sampath D., Merchant M., Yauch R.L., Manning G., Settleman J., Hatzivassiliou G., Evangelista M.
Metabolite profiling stratifies pancreatic ductal adenocarcinomas into subtypes with distinct sensitivities to metabolic inhibitors.
Proc. Natl. Acad. Sci. U.S.A. 112:E4410-E4417(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26884312; DOI=10.1038/srep21648; PMCID=PMC4756684
Gradiz R., Silva H.C., Carvalho L., Botelho M.F., Mota-Pinto A.
MIA PaCa-2 and PANC-1 -- pancreas ductal adenocarcinoma cell lines with neuroendocrine differentiation and somatostatin receptors.
Sci. Rep. 6:21648-21648(2016)
PubMed=27067801; DOI=10.1186/s12885-016-2297-y; PMCID=PMC4828819
Miura K., Kimura K., Amano R., Yamazoe S., Ohira G., Murata A., Nishio K., Hasegawa T., Yashiro M., Nakata B., Ohira M., Hirakawa K.
Establishment and characterization of new cell lines of anaplastic pancreatic cancer, which is a rare malignancy: OCUP-A1 and OCUP-A2.
BMC Cancer 16:268.1-268.13(2016)
PubMed=27259358; DOI=10.1074/mcp.M116.058313; PMCID=PMC4974343
Humphrey E.S., Su S.-P., Nagrial A.M., Hochgrafe F., Pajic M., Lehrbach G.M., Parton R.G., Yap A.S., Horvath L.G., Chang D.K., Biankin A.V., Wu J.-M., Daly R.J.
Resolution of novel pancreatic ductal adenocarcinoma subtypes by global phosphotyrosine profiling.
Mol. Cell. Proteomics 15:2671-2685(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=30156359; DOI=10.1111/cas.13783; PMCID=PMC6215881
Sato T., Muramatsu T., Tanabe M., Inazawa J.
Identification and characterization of transforming growth factor beta induced in circulating tumor cell subline from pancreatic cancer cell line.
Cancer Sci. 109:3623-3633(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31037374; DOI=10.1007/s00216-019-01814-1
Lagies S., Schlimpert M., Braun L.M., Kather M., Plagge J., Erbes T., Wittel U.A., Kammerer B.
Unraveling altered RNA metabolism in pancreatic cancer cells by liquid-chromatography coupling to ion mobility mass spectrometry.
Anal. Bioanal. Chem. 411:6319-6328(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"