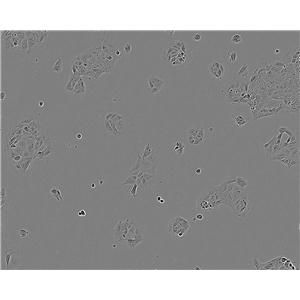
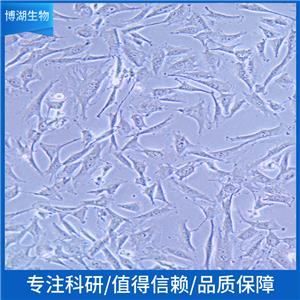
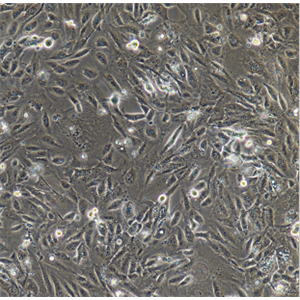
"MDA-MB-453人乳腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
alpha-TC1-6 Cells;背景说明:胰岛素瘤;a细胞;C57BL/6xDBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW-527细胞、CTV-1细胞、SLK细胞
LLC-WRC 256 Cells;背景说明:乳腺癌;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HuNS1细胞、HEEpiC细胞、MHCC-LM3细胞
H-211 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:SUM 190细胞、HT1197细胞、CEM C7细胞
MDA-MB-453人乳腺癌细胞代次低|培养基|送STR图谱
背景信息:该细胞系由CailleauR在1976年从一名48岁的患有转移性乳腺癌的白人女性的心包渗出中分离建立的。该细胞表达FGF的受体。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
MDA-MB-453人乳腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
MHCC 97-H Cells;背景说明:来源于中山医院,用人肝癌细胞株MHCC97-H接种裸鼠,进行3次肺转移筛选,取肺转移瘤建成皮下接种后高度自发性肺转移的肝癌细胞系;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:NIT-1细胞、SW-626细胞、16HBEo-细胞
GL-261 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:Hs-746T细胞、NCI-H1623细胞、CCLP-1细胞
KYSE 180 Cells;背景说明:食管鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H2108细胞、OVCAR-8细胞、SBC-3细胞
SCC 9 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MLE-12细胞、SK-MEL-24细胞、GM03671C细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞传代培养实验:体外培养的原代细胞或细胞株要在体外持续地培养就必须传代,以便获得稳定的细胞株或得到大量的同种细胞,并维持细胞种的延续。培养的细胞形成单层汇合以后,由于密度过大生存空间不足而引起营养枯竭,将培养的细胞分散,从容器中取出,以1:2或1:3以上的比率转移到另外的容器中进行培养,即为传代培养;细胞“一代”指从细胞接种到分离再培养的一段期间,与细胞世代或倍增不同。在一代中,细胞培增3~6次。细胞传代后,一般经过三个阶段:游离期、指数增生期和停止期。常用细胞分裂指数表示细胞增殖的旺盛程度,即细胞群的分裂相数/100个细胞。一般细胞分裂指数介于0.2%~0.5%,肿瘤细胞可达3~5%;细胞接种2~3天分裂增殖旺盛,是活力ZuiHAO时期,称指数增生期(对数生长期),适宜进行各种试验。实验步骤:1.将长成的培养细胞从二氧化碳培养箱中取出,在超净工作台中倒掉瓶内的培养,加入少许消化。(以面盖住细胞为宜),静置5~10分钟。2.在倒置镜下观察被消化的细胞,如果细胞变圆,相互之间不再连接成片,这时应立即在超净台中将消化倒掉,加入3~5ml新鲜培养,吹打,制成细胞悬。3.将细胞悬吸出2ml左右,加到另一个培养瓶中并向每个瓶中分别加3ml左右培养,盖HAO瓶塞,送回二氧化碳培养箱中,继续进行培养。一般情况,传代后的细胞在2小时左右就能附着在培养瓶壁上,2~4天就可在瓶内形成单层,需要再次进行传代。
SW-1116 Cells;背景说明:CSAp阴性(CSAp-)。 结肠抗原3,阴性。 角蛋白免疫过氧化物酶染色阳性。 癌基因c-myc, K-ras, H-ras, myb, sis 和fos的表达呈阳性。 未检测到癌基因N-myc和N-ras的表达。 表达肿瘤特异的核基质蛋白CC-4,CC-5和CC-6。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CL1细胞、Transformed Human Liver Epithelial-3细胞、MODE-K细胞
JM-Jurkat Cells;背景说明:该细胞源自一位14岁患有T淋巴细胞白血病男性的外周血;传代方法:保持细胞密度在3—9×105cells/ml之间,1:5—1:10传代,每周换液2—3次;生长特性:悬浮生长;形态特性:圆形,单个或呈片;相关产品有:PFSK-1细胞、TE85细胞、Hs274T细胞
Vertebral Cancer of the Prostate Cells;背景说明:1997从一位不受激素影响的前列腺癌患者脊椎转移灶中建立了这株细胞。先在小鼠中进行异种移植传代,随后进行体外培养。体内及体外都对雄性激素敏感。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:CT26.WT细胞、JTC-39细胞、MMVECs细胞
OVCA5 Cells;背景说明:卵巢癌;腹水转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PANC-10-05细胞、ML2细胞、MDA-MB-134-VI细胞
MSB-1 Cells;背景说明:淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:WEHI3细胞、MC57细胞、NBL-5细胞
AM38 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:RPMI8226/S细胞、VMRCLCD细胞、Co 205细胞
Swiss 3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:JROECL 19细胞、624细胞、HNE2细胞
C8166-CD4 Cells;背景说明:T淋巴细胞白血病;HTLV-1转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:KRCY细胞、CAL 27细胞、SMMC 7721细胞
NB-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:NCI-HUT-125细胞、HCC-1438细胞、HT clone H9细胞
K562/ADR Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LCMS细胞、PG-4细胞、HSC-I细胞
MAEC Cells;背景说明:主动脉;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH820细胞、TF1细胞、RBL细胞
TCC-PAN2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Suzhou Human Glioma-44细胞、LMH细胞、IOSE-Van细胞
UMR 106 Cells;背景说明:注射放射性同位素磷(32P)诱导产生的可移植性大鼠骨肉瘤克隆建立了UMR-106细胞株。 细胞对PTH,前列腺素及破骨甾体有响应。 对PTH的响应度,UMR-106比相关细胞株UMR-108(ATCC CRL-1663)要高。 蛋白激酶C的活化抑制ATP诱导的胞内水平的升高。 起始骨肉瘤和克隆株都是University of Sheffield的T.J. Martin建立的。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:SW 1990细胞、BSC1细胞、PC-3M-IE8细胞
BHP-10 Cells;背景说明:甲状腺乳头状癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-Mel 2细胞、FCCH1018细胞、OE33细胞
MB157 Cells;背景说明:该细胞源自一位患有乳腺髓样癌的44岁黑人女性,表达WNT7B癌基因,细胞与细胞边界处有细胞桥粒、微绒毛、张力细丝。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SNU-1细胞、18G3.cl 1细胞、SK-ChA-1细胞
M109 Cells;背景说明:肺癌;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JAR细胞、RGC5细胞、Hs895.T细胞
UM-UC-14 Cells;背景说明:肾癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CF-1 MEF细胞、GL261细胞、ZYM-DIEC02细胞
Abcam HEK293T HMGN3 KO Cells(提供STR鉴定图谱)
AG10673 Cells(提供STR鉴定图谱)
BayGenomics ES cell line PST106 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XC832 Cells(提供STR鉴定图谱)
BTL2013 Cells(提供STR鉴定图谱)
CpHi33 Cells(提供STR鉴定图谱)
DA04163 Cells(提供STR鉴定图谱)
Dubca Cells(提供STR鉴定图谱)
GM04280 Cells(提供STR鉴定图谱)
COLO679 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:EPC细胞、Hs-940细胞、SCH细胞
MDA-MB-453人乳腺癌细胞代次低|培养基|送STR图谱
HEL-92 Cells;背景说明:详见相关文献介绍;传代方法:每周2-3次。;生长特性:悬浮生长;形态特性:成淋巴细胞;相关产品有:Hs 737.T细胞、NCIH1184细胞、PT-K75细胞
RWPE-1 Cells;背景说明:肿瘤抑制基因: p53 + [PubMed: 9214605] pRB + [PubMed: 9214605] 一位正常男性前列腺组织切片的周围区域的上皮细胞用单拷贝的人乳头瘤病毒的18(HPV-18)进行转化,建立了RWPE-1 (ATCC CRL-11609) 细胞株 [PubMed: 9214605]. 在三维Matrigel培养时,在雄激素作用下,RWPE-1细胞形成腺胞并向培养基中分泌PSA。[PubMed: 11170142]. 当与Matrigel或基质细胞混合注射雄性裸鼠时,RWPE-1细胞也能形成腺胞[PubMed: 11304724] 并产生PSA。 来源于RWPE-1的细胞再用Kirstin鼠类肿瘤病毒转染Ki-ras基因,建立了能成瘤的RWPE-2细胞株(ATCC CRL-11610) [PubMed: 9214605] 和 RWPE2-W99 (ATCC CRL-2853) 细胞株。 另外,用N--N-(MNU)处理RWPE-1,建立了一系列模拟前列腺癌进程中不同时期的成瘤细胞株。 它们是 WPE1-NA22 (ATCC CRL-2849), WPE1-NB14 (ATCC CRL-2850, WPE1-NB11 (ATCC CRL-2851) 和 WPE1-NB26 (ATCC CRL-2852) 细胞株。 据提供者报道,RWPE-1 细胞株(ATCC CRL-11609)经过检测,乙肝、丙肝、人免疫缺陷病毒都呈阴性。;传代方法:1:3传代,2-3天传一代。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:NCI-H2073细胞、EFM192A细胞、TE3细胞
RSC-96 Cells;背景说明:雪旺细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKRC-52细胞、769-P细胞、SUM 52PE细胞
RGM1 Cells;背景说明:胃黏膜;上皮细胞;自发永生;Wistar;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C1R细胞、HT-29细胞、SKMel-28细胞
UMRC2 Cells;背景说明:肾透明细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:32D/cl3细胞、Malme3M细胞、HBZY-1细胞
76-2-11 Cells(提供STR鉴定图谱)
EO771 Cells;背景说明:恶性乳腺癌;雌性;C57BL/6;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH841细胞、BALB/3T3 cl. A31细胞、Hs 852.T细胞
SCC15 Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H1092细胞、NCI-H1694细胞、Colo699细胞
SUD6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:NCI-H1623细胞、RCS细胞、Walker-256-TC细胞
SJRH 30 Cells;背景说明:肺泡横纹肌肉瘤;骨髓转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCI-H498细胞、GA-10(Clone 4)细胞、HVSMC细胞
ReNcell CX Cells;背景说明:神经干 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HM06.A1细胞、U251MG细胞、P3-x63-Ag8 653细胞
HCC95 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RN-c细胞、Neuro-2a细胞、SV-HUC细胞
HEY-A8 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H1395细胞、SNU520细胞、CHO cell clone K1细胞
GA-10 (Clone 4) Cells;背景说明:详见相关文献介绍;传代方法:每2-3天换液;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:JM细胞、CEM细胞、KMY1022细胞
GM25864 Cells(提供STR鉴定图谱)
HAP1 NSUN5 (-) 1 Cells(提供STR鉴定图谱)
Mv 1 Lu Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Sf-21细胞、C4-1细胞、RL-65细胞
OSA Cells;背景说明:详见相关文献介绍;传代方法:1:5-1:10传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:SHIN3细胞、SUDHL-4细胞、OCI-Ly07细胞
SW-1353 Cells;背景说明:骨肉瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:AZ521细胞、NCIH146细胞、C42细胞
Verda reno Cells;背景说明:Vero细胞株是日本千叶大学的YasumuraY和KawakitaY从正常成年非洲绿猴的肾脏组织中分离建立的。该细胞常作为转染宿主,用于支原体的检测。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Roswell Park Memorial Institute 1788细胞、H9c2(2-1)细胞、KG1A细胞
UMNSAH-DF 1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维母细胞样;相关产品有:EOL1细胞、SK.MEL.2细胞、H2052_MM细胞
Adult Retinal Pigment Epithelial cell line-19 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Mono Mac 6细胞、HOS-MNNG细胞、LN-229细胞
L5178Y-R Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:mIMCD3细胞、RT-BM细胞、HK2细胞
OCI-Ly 19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK-HEP-1细胞、UCLA RO-81-A-1细胞、J 82细胞
Hs 415.Sk Cells(提供STR鉴定图谱)
KMK-2 Cells(提供STR鉴定图谱)
MLC-2-4 Cells(提供STR鉴定图谱)
NSI001-A Cells(提供STR鉴定图谱)
REF-WT1A Cells(提供STR鉴定图谱)
Ubigene HEK293 DGKA KO Cells(提供STR鉴定图谱)
WG0305 Cells(提供STR鉴定图谱)
HCT-116 MTBP-mClover-XLoneGemininT25D Cells(提供STR鉴定图谱)
NCI-H524 Cells;背景说明:该细胞1982年建系,源自非小细胞肺癌男性患者的转移淋巴结。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮生长;形态特性:圆形细胞;相关产品有:DHL8细胞、IMR32细胞、UPCI-SCC090细胞
RA-FLSs Cells;背景说明:关节;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HCC0044细胞、ZR75-30细胞、L6细胞
Panc2.03 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AMJ2-C8细胞、U266BL细胞、C 6细胞
Farage OL Cells;背景说明:Farage细胞源于一名患有弥漫性大B细胞淋巴瘤(DLBCL)白人女性的活检淋巴组织,由HBen-Bassat建系。经IL-4处理,该细胞CD21,CD22,CD54和CD58表达上调,而CD21,CD22,andCD38表达下调。;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:F-36P细胞、SK-RC 52细胞、SK-MEL5细胞
P19 Cells;背景说明:P19细胞株是从C3H/He小鼠中诱导的恶性畸胎瘤中建立的。该细胞在含有0.1mM的培养基中可高效率地克隆。该细胞具有多能性,在500nM维A酸诱导下可以分化成神经和神经胶质样细胞;在0.5%~1.0%二甲亚砜(DMSO)存在下,分化形成心脏和骨骼肌样细胞,但不形成神经或神经胶质样细胞;在DMSO和维A酸同时存在时,细胞的分化与只有维A酸一样。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KYSE140细胞、LN-18细胞、L 363细胞
P19 Cells;背景说明:P19细胞株是从C3H/He小鼠中诱导的恶性畸胎瘤中建立的。该细胞在含有0.1mM的培养基中可高效率地克隆。该细胞具有多能性,在500nM维A酸诱导下可以分化成神经和神经胶质样细胞;在0.5%~1.0%二甲亚砜(DMSO)存在下,分化形成心脏和骨骼肌样细胞,但不形成神经或神经胶质样细胞;在DMSO和维A酸同时存在时,细胞的分化与只有维A酸一样。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KYSE140细胞、LN-18细胞、L 363细胞
NCIH2126 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,每周2-3次。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:GM05862细胞、H1954细胞、HS-766T细胞
NCIH841 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:5传代,;生长特性:混合型生长;形态特性:详见产品说明书;相关产品有:C33-A细胞、HARAB细胞、NCI-SNU-761细胞
A549 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:D-341Med细胞、38C13细胞、CATHa细胞
Anglne Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:COC1/CDDP细胞、HO-8910 PM细胞、GM03573细胞
P3.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ect1/E6E7细胞、Stanford University-Diffuse Histiocytic Lymphoma-2细胞、H446细胞
P3/X63-Ag8 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MHCC 97细胞、MOVAS细胞、HCC-366细胞
HCC-15 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MRMT-1细胞、Swiss 3T3细胞、OCI-Ly1细胞
Anip973 Cells;背景说明:肺腺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:WI-38细胞、COLO-320-DM细胞、MDCKII细胞
OV1/P Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CA46细胞、MDAMB435细胞、CCD18细胞
STBCi094-A Cells(提供STR鉴定图谱)
A-172 MG Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC-827细胞、COLO-824细胞、MEC1细胞
Mahlavu Cells;背景说明:肝癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Sp2/0-Ag14细胞、MB468细胞、HO-1-N-1细胞
Bac 1.2F5 Cells;背景说明:巨噬细胞;SV40转化;BALB/c x A.CA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:130-T细胞、K422细胞、NTERA-2cl.D1细胞
PLB 985 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SHI1细胞、OEC33细胞、FRH-0201细胞
F81 Cells;背景说明:肾;自发永生;雌性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:UPCI-SCC-90细胞、Natural Killer-92细胞、BHK21细胞
CX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MIA-Pa-Ca-2细胞、BV-2细胞、GM03320细胞
MDA-MB-453人乳腺癌细胞代次低|培养基|送STR图谱
HONE-1 Cells;背景说明:鼻咽癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PG-4 S+L-细胞、GM05372E细胞、H1522细胞
OCI-Ly10 Cells;背景说明:弥漫大B细胞淋巴瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RIN-m 14B细胞、Hs 578T细胞、P388.D1细胞
751-NA Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EFO27细胞、HeLa.S3细胞、138MG细胞
WEHI-279 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:P31FUJ细胞、GT1-1细胞、CCD-33Co细胞
CEM/C1 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:H-2106细胞、7404细胞、RL-65细胞
143TK- Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代;每周换液2-3次;生长特性:贴壁生长;形态特性:混合型;相关产品有:BE(2)C细胞、SUM-52细胞、786-0WT细胞
NCI-H1395 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;5-6天传代一次。;生长特性:贴壁生长;形态特性:上皮样;多角形;相关产品有:MLMA细胞、SNU1040细胞、SKNEP细胞
LICR-LON-HN6-R Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Clone 15 HL-60细胞、Hs27F细胞、HPAF-I细胞
BayGenomics ES cell line RRH303 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XH334 Cells(提供STR鉴定图谱)
ES[MC1R(20):tetSix1(15)] Cells(提供STR鉴定图谱)
Na 42/13 Cells(提供STR鉴定图谱)
Ubigene TM3 Gstk1 KO Cells(提供STR鉴定图谱)
mbn-3 Cells(提供STR鉴定图谱)
" "DOI=10.1016/B978-0-12-333530-2.50009-5
Leibovitz A.
Cell lines from human breast.
(In book chapter) Atlas of human tumor cell lines; Hay R.J., Park J.-G., Gazdar A.F. (eds.); pp.161-184; Academic Press; New York; USA (1994)
PubMed=9815641
Wosikowski K., Schuurhuis D.H., Kops G.J.P.L., Saceda M., Bates S.E.
Altered gene expression in drug-resistant human breast cancer cells.
Clin. Cancer Res. 3:2405-2414(1997)
PubMed=9671407; DOI=10.1038/sj.onc.1201814
Sweeney K.J., Swarbrick A., Sutherland R.L., Musgrove E.A.
Lack of relationship between CDK activity and G1 cyclin expression in breast cancer cells.
Oncogene 16:2865-2878(1998)
PubMed=10969801
Forozan F., Mahlamaki E.H., Monni O., Chen Y.-D., Veldman R., Jiang Y., Gooden G.C., Ethier S.P., Kallioniemi A.H., Kallioniemi O.-P.
Comparative genomic hybridization analysis of 38 breast cancer cell lines: a basis for interpreting complementary DNA microarray data.
Cancer Res. 60:4519-4525(2000)
PubMed=11687795; DOI=10.1038/ng754
Snijders A.M., Nowak N.J., Segraves R., Blackwood S., Brown N., Conroy J., Hamilton G., Hindle A.K., Huey B., Kimura K., Law S., Myambo K., Palmer J., Ylstra B., Yue J.P., Gray J.W., Jain A.N., Pinkel D., Albertson D.G.
Assembly of microarrays for genome-wide measurement of DNA copy number.
Nat. Genet. 29:263-264(2001)
PubMed=11789735; DOI=10.1309/4NCM-QJ9W-QM0J-6QJE
Rhodes A., Jasani B., Couturier J., McKinley M.J., Morgan J.M., Dodson A.R., Navabi H., Miller K.D., Balaton A.J.
A formalin-fixed, paraffin-processed cell line standard for quality control of immunohistochemical assay of HER-2/neu expression in breast cancer.
Am. J. Clin. Pathol. 117:81-89(2002)
PubMed=12353263; DOI=10.1002/gcc.10107
Popovici C., Basset C., Bertucci F., Orsetti B., Adelaide J., Mozziconacci M.-J., Conte N., Murati A., Ginestier C., Charafe-Jauffret E., Ethier S.P., Lafage-Pochitaloff M., Theillet C., Birnbaum D., Chaffanet M.
Reciprocal translocations in breast tumor cell lines: cloning of a t(3;20) that targets the FHIT gene.
Genes Chromosomes Cancer 35:204-218(2002)
PubMed=12800145; DOI=10.1002/gcc.10218
Adelaide J., Huang H.-E., Murati A., Alsop A.E., Orsetti B., Mozziconacci M.-J., Popovici C., Ginestier C., Letessier A., Basset C., Courtay-Cahen C., Jacquemier J., Theillet C., Birnbaum D., Edwards P.A.W., Chaffanet M.
A recurrent chromosome translocation breakpoint in breast and pancreatic cancer cell lines targets the neuregulin/NRG1 gene.
Genes Chromosomes Cancer 37:333-345(2003)
PubMed=16142302; DOI=10.3892/ijo.27.4.881
de Longueville F., Lacroix M., Barbuto A.-M., Bertholet V., Gallo D., Larsimont D., Marcq L., Zammatteo N., Boffe S., Leclercq G., Remacle J.
Molecular characterization of breast cancer cell lines by a low-density microarray.
Int. J. Oncol. 27:881-892(2005)
PubMed=16397213; DOI=10.1158/0008-5472.CAN-05-2853
Elstrodt F., Hollestelle A., Nagel J.H.A., Gorin M., Wasielewski M., van den Ouweland A.M.W., Merajver S.D., Ethier S.P., Schutte M.
BRCA1 mutation analysis of 41 human breast cancer cell lines reveals three new deleterious mutants.
Cancer Res. 66:41-45(2006)
PubMed=16541312; DOI=10.1007/s10549-006-9186-z
Wasielewski M., Elstrodt F., Klijn J.G.M., Berns E.M.J.J., Schutte M.
Thirteen new p53 gene mutants identified among 41 human breast cancer cell lines.
Breast Cancer Res. Treat. 99:97-101(2006)
PubMed=17157791; DOI=10.1016/j.ccr.2006.10.008; PMCID=PMC2730521
Neve R.M., Chin K., Fridlyand J., Yeh J., Baehner F.L., Fevr T., Clark L., Bayani N., Coppe J.-P., Tong F., Speed T., Spellman P.T., DeVries S., Lapuk A., Wang N.J., Kuo W.-L., Stilwell J.L., Pinkel D., Albertson D.G., Waldman F.M., McCormick F., Dickson R.B., Johnson M.D., Lippman M.E., Ethier S.P., Gazdar A.F., Gray J.W.
A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes.
Cancer Cell 10:515-527(2006)
PubMed=18516279; DOI=10.1016/j.molonc.2007.02.004; PMCID=PMC2391005
Kenny P.A., Lee G.Y., Myers C.A., Neve R.M., Semeiks J.R., Spellman P.T., Lorenz K., Lee E.H., Barcellos-Hoff M.H., Petersen O.W., Gray J.W., Bissell M.J.
The morphologies of breast cancer cell lines in three-dimensional assays correlate with their profiles of gene expression.
Mol. Oncol. 1:84-96(2007)
PubMed=19582160; DOI=10.1371/journal.pone.0006146; PMCID=PMC2702084
Kao J., Salari K., Bocanegra M., Choi Y.-L., Girard L., Gandhi J., Kwei K.A., Hernandez-Boussard T., Wang P., Gazdar A.F., Minna J.D., Pollack J.R.
Molecular profiling of breast cancer cell lines defines relevant tumor models and provides a resource for cancer gene discovery.
PLoS ONE 4:E6146-E6146(2009)
DOI=10.25904/1912/1434
Morrison B.J.
Breast cancer stem cells: tumourspheres and implications for therapy.
Thesis PhD (2010); Griffith University; Brisbane; Australia
PubMed=19593635; DOI=10.1007/s10549-009-0460-8
Hollestelle A., Nagel J.H.A., Smid M., Lam S., Elstrodt F., Wasielewski M., Ng S.S., French P.J., Peeters J.K., Rozendaal M.J., Riaz M., Koopman D.G., ten Hagen T.L.M., de Leeuw B.H.C.G.M., Zwarthoff E.C., Teunisse A.F.A.S., van der Spek P.J., Klijn J.G.M., Dinjens W.N.M., Ethier S.P., Clevers H.C., Jochemsen A.G., den Bakker M.A., Foekens J.A., Martens J.W.M., Schutte M.
Distinct gene mutation profiles among luminal-type and basal-type breast cancer cell lines.
Breast Cancer Res. Treat. 121:53-64(2010)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=21378333
Ford C.H.J., Al-Bader M., Al-Ayadhi B., Francis I.
Reassessment of estrogen receptor expression in human breast cancer cell lines.
Anticancer Res. 31:521-527(2011)
PubMed=22121396; DOI=10.3892/ol.2011.375; PMCID=PMC3224077
Vranic S., Gatalica Z., Wang Z.-Y.
Update on the molecular profile of the MDA-MB-453 cell line as a model for apocrine breast carcinoma studies.
Oncol. Lett. 2:1131-1137(2011)
PubMed=22414580; DOI=10.1158/0008-5472.CAN-11-3711
Geiger T., Madden S.F., Gallagher W.M., Cox J., Mann M.
Proteomic portrait of human breast cancer progression identifies novel prognostic markers.
Cancer Res. 72:2428-2439(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22585861; DOI=10.1158/2159-8290.CD-11-0224; PMCID=PMC5057396
Marcotte R., Brown K.R., Suarez Saiz F.J., Sayad A., Karamboulas K., Krzyzanowski P.M., Sircoulomb F., Medrano M., Fedyshyn Y., Koh J.L.-Y., van Dyk D., Fedyshyn B., Luhova M., Brito G.C., Vizeacoumar F.J., Vizeacoumar F.S., Datti A., Kasimer D., Buzina A., Mero P., Misquitta C., Normand J., Haider M., Ketela T., Wrana J.L., Rottapel R., Neel B.G., Moffat J.
Essential gene profiles in breast, pancreatic, and ovarian cancer cells.
Cancer Discov. 2:172-189(2012)
PubMed=22719059; DOI=10.1530/ERC-12-0065
Moore N.L., Buchanan G., Harris J.M., Selth L.A., Bianco-Miotto T., Hanson A.R., Birrell S.N., Butler L.M., Hickey T.E., Tilley W.D.
An androgen receptor mutation in the MDA-MB-453 cell line model of molecular apocrine breast cancer compromises receptor activity.
Endocr. Relat. Cancer 19:599-613(2012)
PubMed=23601657; DOI=10.1186/bcr3415; PMCID=PMC3672661
Riaz M., van Jaarsveld M.T.M., Hollestelle A., Prager-van der Smissen W.J.C., Heine A.A.J., Boersma A.W.M., Liu J.-J., Helmijr J.C.A., Ozturk B., Smid M., Wiemer E.A.C., Foekens J.A., Martens J.W.M.
miRNA expression profiling of 51 human breast cancer cell lines reveals subtype and driver mutation-specific miRNAs.
Breast Cancer Res. 15:R33.1-R33.17(2013)
PubMed=24094812; DOI=10.1016/j.ccr.2013.08.020; PMCID=PMC3931310
Timmerman L.A., Holton T., Yuneva M., Louie R.J., Padro M., Daemen A., Hu M., Chan D.A., Ethier S.P., van 't Veer L.J., Polyak K., McCormick F., Gray J.W.
Glutamine sensitivity analysis identifies the xCT antiporter as a common triple-negative breast tumor therapeutic target.
Cancer Cell 24:450-465(2013)
PubMed=24176112; DOI=10.1186/gb-2013-14-10-r110; PMCID=PMC3937590
Daemen A., Griffith O.L., Heiser L.M., Wang N.J., Enache O.M., Sanborn Z., Pepin F., Durinck S., Korkola J.E., Griffith M., Hur J.S., Huh N., Chung J., Cope L., Fackler M.J., Umbricht C.B., Sukumar S., Seth P., Sume V.P., Jakkula L.R., Lu Y.-L., Mills G.B., Cho R.J., Collisson E.A., van 't Veer L.J., Spellman P.T., Gray J.W.
Modeling precision treatment of breast cancer.
Genome Biol. 14:R110.1-R110.14(2013)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25892236; DOI=10.1016/j.celrep.2015.03.050; PMCID=PMC4425736
Lawrence R.T., Perez E.M., Hernandez D., Miller C.P., Haas K.M., Irie H.Y., Lee S.-I., Blau C.A., Villen J.
The proteomic landscape of triple-negative breast cancer.
Cell Rep. 11:630-644(2015)
PubMed=26218769; DOI=10.1016/j.jchromb.2015.07.021
Willmann L., Schlimpert M., Halbach S., Erbes T., Stickeler E., Kammerer B.
Metabolic profiling of breast cancer: differences in central metabolism between subtypes of breast cancer cell lines.
J. Chromatogr. B 1000:95-104(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28889351; DOI=10.1007/s10549-017-4496-x
Saunus J.M., Smart C.E., Kutasovic J.R., Johnston R.L., Kalita-de Croft P., Miranda M., Rozali E.N., Vargas A.C., Reid L.E., Lorsy E., Cocciardi S., Seidens T., McCart Reed A.E., Dalley A.J., Wockner L.F., Johnson J., Sarkar D., Askarian-Amiri M.E., Simpson P.T., Khanna K.K., Chenevix-Trench G., Al-Ejeh F., Lakhani S.R.
Multidimensional phenotyping of breast cancer cell lines to guide preclinical research.
Breast Cancer Res. Treat. 167:289-301(2018)
PubMed=30787054; DOI=10.1158/1055-9965.EPI-18-1132; PMCID=PMC6548687
Hooker S.E. Jr., Woods-Burnham L., Bathina M., Lloyd S., Gorjala P., Mitra R., Nonn L., Kimbro K.S., Kittles R.A.
Genetic ancestry analysis reveals misclassification of commonly used cancer cell lines.
Cancer Epidemiol. Biomarkers Prev. 28:1003-1009(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)"