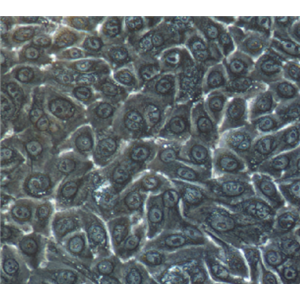
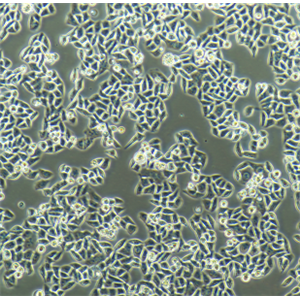
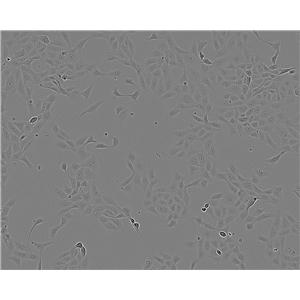
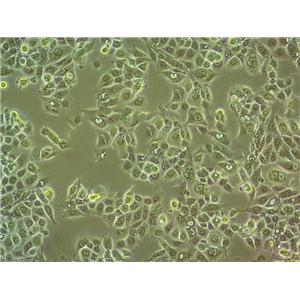
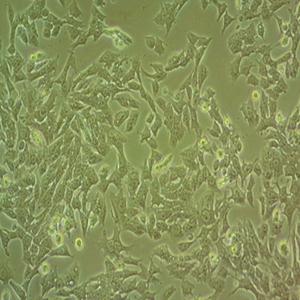
"Hela人宫颈癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
公司细胞系形态漂亮、增殖倍数高、纯度高、功能性强,细胞培养就跟养孩子一个样。养孩子要喂奶,养细胞要加补液,都需要在前期补充足够的营养,初始状态的细胞或刚刚复苏的细胞还要适量加入血清或细胞因子来帮助它们的存活增殖,如果营养物质缺乏,细胞就会不生长甚至死亡。养孩子要从小培养学习,养细胞也得培养宝宝顺利生下来,你会经常抚摸他,给他看各种颜色,刺激他的五感。细胞也是一样,分离后的细胞需要使用特定的细胞因子进行活化、增殖。另外加入因子的种类、因子的浓度、加入时间、加入顺序都会影响细胞最终的结果。养孩子最怕孩子生病,养细胞最怕被污染,平时你会仔细观察宝宝是否呕吐、是否突然哭闹,猜测宝宝是否生病了。对于细胞,我们也需要时刻进行观察的,假如培养液浑浊(污染了),则需要换液后加抗生素;假如细胞增殖不明显,形态变差,则可能是因为营养不足了,对贴壁细胞可以消化后重新用新的培养基接种并加倍加入细胞因子含量;对悬浮细胞增殖能力不强的,则不着急补液,只是先补加血清、细胞因子看是否可以好转。培养时还得全程在无菌的环境,一个小小的偏差,细胞就会死亡。
换液周期:每周2-3次
H-719 Cells;背景说明:小细胞肺癌;骨髓转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:Caki2细胞、H1155细胞、HuT 102细胞
BC-3-H-I Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CHP126细胞、Lewis Lung细胞、NBL-3细胞
DU-145 Cells;背景说明:DU 145 是从一位有3年淋巴细胞白血病史的前列腺癌患者的脑部转移灶中建立的。该细胞系未检测到激素敏感性,酸性酶阳性,单个的细胞可在软琼脂中形成集落。对此细胞和原始肿瘤的亚显微结构分析可见微绒毛、微丝、细胞桥粒、线粒体、发达的高尔基体和异质溶酶体。该细胞不表达前列腺抗原。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:HRGEC细胞、Human Pancreatic Duct Epithelial细胞、Hx-147细胞
Hela人宫颈癌细胞代次低|培养基|送STR图谱
背景信息:HeLa是第一个来自人体组织经连续培养获得的非整倍体上皮样细胞系,它由GeyGO等在1951年从31岁女性黑人的宫颈癌组织建立。经原始组织切片重新观察,Jones等将其诊断为腺癌。已知该细胞系含有人乳头状瘤病毒HPV18序列,需在2级生物安全防护台操作。该细胞角蛋白阳性,p53表达量较低,但表达正常水平的pRB(视网膜母细胞瘤抑制因子)。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
贴壁细胞的传代培养,详细步骤如下:首先倒掉培养基,在这一步骤可以收集一些细胞上清做支原体检测;加入胰蛋白酶,一般T25是加2mL,盖好瓶盖,摇晃T25培养瓶,使胰蛋白酶均匀覆盖在细胞表面,放入培养箱2-3min,期间可在显微镜下观察,看到大部分细胞变圆,即可放入超净台,加入2倍的完全培养基,这里就是加4mL培养基,终止消化;将含有胰蛋白酶,细胞和培养基一起转移到离心管中,1000rpm/3min离心,去掉上清;新鲜的完全培养基重悬,根据细胞的生长特性和后续的实验需求进行传代,比如我养的Hepa1-6就长的比较快,不是着急用的话,我就会按1E6个细胞/T75培养瓶进行传代;但如果后两天要用,就会适当多传一点;还可通过显微镜计数后,直接用于细胞铺板,继续后续的实验。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
Hela人宫颈癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
COLO 320 DM Cells;背景说明:该细胞可产生5-羟色胺、去甲、、ACTH和甲状旁腺素。角蛋白、波形蛋白弱阳性。培养条件: RPMI 1640 10%FBS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮+贴壁;形态特性:淋巴细胞;相关产品有:HT 1197.T细胞、WPMY1细胞、SK-MEL-2-LUC细胞
C28/I2 Cells;背景说明:软骨;SV40转化;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CHO/dhFr-细胞、Hos TE-85细胞、526 mel细胞
HLEB-3 Cells;背景说明:晶状体;Ad12-SV40转化;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MM1细胞、Ramos-2G6-4C10细胞、U266B1细胞
CCRF/CEM/0 Cells;背景说明:G.E. Foley 等人建立了类淋巴母细胞细胞株CCRF-CEM。 细胞是1964年11月从一位四岁白人女性急性淋巴细胞白血病患者的外周血白血球衣中得到。此细胞系从香港收集而来。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:H9c2(2-1)细胞、HCC-1419细胞、H661细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞复苏后贴壁细胞较少的问题分析:总结1:复苏过程没有问题,是否是从拿出直接放入温水,还有培养箱,二氧化碳浓度,培养基、PH值等环节。要么加GAO浓度FBS 15-20%,看看能否帮助贴壁,当然也需要考虑血清问题,还有确信拿来的细胞没问题。总结2:首先应该怀疑冻存,实际上复苏出问题的可能非常小,因为操作简单,而且死板。1、你冻存的时候是不是消化的时间过长,这是一般人所注意不到的,即使书上也不讲这个问题,太长的消化时间会让细胞复苏时失去贴壁能力,表现为先贴后死,原因是在你复苏的时候细胞已进入凋亡程序,不可逆转的死亡。2、你的冻存HAO不HAO,是什么,甘油还是DMSO,质量非常重要,否则也会死亡。3、你的冻存的量加的是不是太多,AC推荐是不超过7%,大于5%,太多也不HAO。4、你在冻存的时候是不是把DMSO混均匀,这个有一些影响,但不算太大。5、你的冻存是否按部就班,就是所温度梯度是不是把握严格,很多人容易忘却这个事情,因为这个东西流程长。6、如果你细胞污染,你是否能很快看到,我比我的导师能早一天看到污染。从这个角度讲建议去除离心这步。7、你的细胞在冻存前是否过密。还有,不赞成孵箱污染这个概念的,所有在一个孵箱里的细胞都污染一个细菌的话,这个细菌是源于孵箱的,但这不代表孵箱污染,因为孵箱无论你如何处理都有大量的细菌,问题在操作。每次污染的原因都要尽可能的找,以后就不犯同样的问题,这个很重要,不能靠猜,否则你就有可能细胞养绝Zui后换课题,这个见得太多了,别不当会事。
WEHI3 Cells;背景说明:白血病;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Hs294T细胞、RPMI-7951细胞、SGC7901/DDP细胞
Colo16 Cells;背景说明:皮肤鳞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HBMEC细胞、NCIH520细胞、Wills Eye Research Institute-Retinoblastoma-1细胞
SU8686 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:BRL3A细胞、PANC-28细胞、LuCL4细胞
Tu-212 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:SNU-475细胞、NCI-H526细胞、V79-4细胞
MDA MB 361 Cells;背景说明:该细胞源自40岁女性乳腺癌的脑转移组织。;传代方法: 1:2—1:6传代,每周换液2—3次;生长特性:松散贴壁生长;形态特性:上皮细胞样;相关产品有:H3255_DA细胞、Renal Proximal Tubule Epithelial Cells/TERT-immortalized 1细胞、HFF-1细胞
H-1688 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MDA330细胞、LLC-PK-1细胞、Moorfields/Institute of Ophthalmology-Muller 1细胞
Vero 76 clone E6 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:EFO 27细胞、HMCB细胞、PaTu-8988s细胞
B104 [Rat neuroblastoma] Cells;背景说明:神经母细胞瘤;BDIX;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C57/B6-L细胞、HHL5细胞、C166细胞
GM02132C Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:HR-8348细胞、GLC82细胞、MC57G细胞
LC1/Sq Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs 822.T细胞、Be-Wo细胞、HCC-1806细胞
aTC1 Clone 6 Cells;背景说明:胰岛素瘤;a细胞;C57BL/6xDBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:D407 RPE细胞、NCTC 1469细胞、4T1细胞
CZ-1 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:ST细胞、EFO-27细胞、HMC细胞
GM03320 Cells;背景说明:该细胞是1967年4月由NicholsWW,LeeJ和DwightS建立,来源于一名13月龄白人男婴腹部肿块,临床诊断为神经母细胞瘤,伴有极少部位的类器官样分化。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:存在两种细胞类型,小的神经母细胞样细胞和大的透明成纤维样细胞;相关产品有:SK.OV.3细胞、675T细胞、L-02细胞
NCI N87 Cells;背景说明:NCI-N87细胞表达表面糖蛋白癌胚抗原(CEA)和TAG72,并且没有左旋多巴胺脱羧酶(DDC)活性。它们的血管活性的肠肽(VIP)受体活性极低并缺乏胃泌激素受体。它们表达蕈毒碱胆碱受体。没有证据表明存在N-myc,L-myc,myb和EGF受体基因的重组。这个细胞株表达的c-myc和c-erb-B2RNA水平与其它细胞株相当。以下基因不表达:N-myc,L-myc,c-cis,IGF-2,或胃泌激素释放肽。报道NCI-N87细胞的植板率为4.3%。;传代方法:消化15-20分钟。1:2。4-5天长满。;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:MGC-803细胞、MV4-11细胞、NCIH208细胞
H-1238 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ARH77细胞、Anip[973]细胞、THLE-3细胞
Rat Fetal Lung-6 Cells;背景说明:胚肺;成纤维细胞;SD大鼠;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JB6 [Mouse]细胞、HUV-EC-C细胞、SK-MEL-2细胞
SkChA1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:REC1细胞、H-146细胞、LU165细胞
143B pML BK TK Cells(提供STR鉴定图谱)
Abcam HeLa FXR1 KO Cells(提供STR鉴定图谱)
AG21276 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRF229 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XG446 Cells(提供STR鉴定图谱)
C0189 Cells(提供STR鉴定图谱)
CW60066 Cells(提供STR鉴定图谱)
DTE 1-6-4 Cells(提供STR鉴定图谱)
GM04208 Cells(提供STR鉴定图谱)
NCI-H165 Cells;背景说明:非小细胞肺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H69C细胞、MC-4细胞、HPF细胞
Hela人宫颈癌细胞代次低|培养基|送STR图谱
RPMI No. 1846 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H2081细胞、MDA-MB-231 #4175细胞、SK-OV-3细胞
A9(Hamprecht) Cells;背景说明:皮下结缔组织;自发永生;雄性;C3H/An;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SCC-15细胞、HCC-1359细胞、PC-14细胞
Panc 08.13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:AZ 521细胞、HKF细胞、HSAS1细胞
Wien 133 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCI-H1404细胞、KLM-1细胞、MNNG细胞
NT2D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GAK细胞、NL20SV细胞、D10细胞
7265PDA Cells(提供STR鉴定图谱)
HIT.T15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:M3 Clone M-3细胞、H7721细胞、Capan 1细胞
CX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MIA-Pa-Ca-2细胞、BV-2细胞、GM03320细胞
SW954 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:KNS81细胞、MIA-Pa-Ca-2细胞、HCC15细胞
PLC/PRF5 Cells;背景说明:该细胞系分泌乙肝病毒表面抗原(HBsAg)。 此细胞系原先被支原体污染,后用BM-cycline去除支原体;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KYSE 510细胞、SUPB15细胞、LC1sq细胞
KRC Y Cells;背景说明:肾透明细胞癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:L540细胞、B-95-8细胞、McG-1细胞
Hs-27 Cells;背景说明:包皮;成纤维细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SKNBE2细胞、NTERA2-D1细胞、SNU5细胞
OVCA 420 Cells;背景说明:卵巢癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:IOSE 80细胞、RT-112细胞、NK92MI细胞
SUDHL-2 Cells;背景说明:弥漫性大细胞淋巴瘤;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:LP-1细胞、SK-N-BE-2细胞、8226细胞
GM21889 Cells(提供STR鉴定图谱)
HAP1 MARK3 (-) 1 Cells(提供STR鉴定图谱)
TE-32 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:P3-Jiyoye细胞、hTERT-RPE细胞、EVSAT细胞
PG [Human lung carcinoma] Cells;背景说明:巨细胞肺癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NCIH2009细胞、LWnt3A细胞、CL11细胞
UMRC2 Cells;背景说明:肾透明细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:32D/cl3细胞、Malme3M细胞、HBZY-1细胞
HCC2108 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BNCL2细胞、MXI细胞、SW1783细胞
HM1900 Cells;背景说明:小胶质 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs 636.T细胞、RFL6细胞、Immortal Pig Intestinal-2I细胞
8226/S Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:LCD细胞、HCC-4006细胞、293细胞
HIT T-15 Cells;背景说明:胰岛β细胞;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CORL23细胞、SGC 7901细胞、SK RC 20细胞
YH-13 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:HEp-2细胞、NT2细胞、SHEE细胞
HPSI0616i-beyk_6 Cells(提供STR鉴定图谱)
KAUSTi007-A Cells(提供STR鉴定图谱)
MEO clone 515 Cells(提供STR鉴定图谱)
NHEM680 Cells(提供STR鉴定图谱)
Raji CD19 Low Cells(提供STR鉴定图谱)
U-87MG ATCC ECE1 KO Cells(提供STR鉴定图谱)
UM29-3 PGD Cells(提供STR鉴定图谱)
HG01367 Cells(提供STR鉴定图谱)
HCC1143 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:Biopsy xenograft of Pancreatic Carcinoma line-3细胞、NL-20细胞、MDCK-II细胞
RPMI1788 Cells;背景说明:B淋巴细胞;EBV转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCI-H1436细胞、UM-UC14细胞、NCI-H1436细胞
HC11 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MM6细胞、HEC-251细胞、NUGC4细胞
K299 Cells;背景说明:间变性大细胞淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CL34细胞、H-2347细胞、NK-92细胞
U14 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:IFRS1细胞、DR2R 1610细胞、H-1838细胞
U14 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:IFRS1细胞、DR2R 1610细胞、H-1838细胞
Chang Cells Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Z138细胞、G-361细胞、KYSE450细胞
130 T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:H2106细胞、3396细胞、MDAMB231细胞
Th17 Cells;背景说明:辅助性T Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:MIN-6细胞、SHIN3细胞、Eph4 1424细胞
PL-12 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MOLT-3细胞、NCIH747细胞、NIE-115细胞
U251-MG Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:Leukemia L1210细胞、DMS-153细胞、CCD33Co细胞
U-251 MG Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:AML-EOL-1细胞、AMO1细胞、PC615.3细胞
UWB1.289 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:118MG细胞、NCIH196细胞、FD-LSC-1细胞
KHYG-1 Cells;背景说明:NK细胞淋巴瘤/白血病;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:Y3细胞、GM00346细胞、BALB 3T3 clone A31细胞
HBVP Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH1793细胞、Panc-8_13细胞、Reuber H35细胞
ST90/MGH12 Cells(提供STR鉴定图谱)
FAK+/+ Cells;背景说明:详见相关文献介绍;传代方法:1:4-1:8传代; ;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:NCI-H1869细胞、Melanoma 14细胞、RD-ES细胞
NCIH322 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:SW-1116细胞、H-865细胞、Henrietta Lacks cells细胞
J111 Cells;背景说明:单核细胞白血病;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCIH650细胞、Hs819T细胞、University of Michigan-Urothelial Carcinoma-14细胞
B16-F10 Cells;背景说明:B16-F10是B16-F0的亚系。;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Sp2/mIL-6细胞、HEK293-A细胞、EU-4细胞
B16 subline B78 Cells;背景说明:该细胞源于C57BL/6J小鼠黑色素瘤,可以产生黑色素,同基因小鼠体内移植可成瘤;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:梭形;相关产品有:SKMEL-2细胞、DMS-273细胞、CALU1细胞
BC-028 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:FHC细胞、H446细胞、IAR20细胞
Hela人宫颈癌细胞代次低|培养基|送STR图谱
CAL 12 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PTK2细胞、HO8910/PM细胞、IPLB-SF 21细胞
HeLaS3 Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KP-N-RT-BM细胞、HCC0202细胞、AC29细胞
BT 549 Cells;背景说明:该细胞1978年由W.G.Coutinho和E.Y.Lasfargues建系,源自一位72岁患有乳腺导管癌的白人女性,来源组织包括乳头及浸润导管。该细胞形态包括上皮样细胞及多核巨细胞,可分泌一种粘性物质。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:DU4475细胞、HS-729细胞、Mouse podocyte细胞
KE-39 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs706T细胞、LAD-2细胞、Wills Eye Research Institute-Retinoblastoma-1细胞
H1666_DA Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2次。;生长特性:贴壁生长,松散;形态特性:上皮细胞;相关产品有:RK13细胞、RT-BM 1细胞、ECC 12细胞
WEHI164 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RMS 13细胞、MDA-MB436细胞、BC-3-H-I细胞
HuT 78 Cells;背景说明:皮肤;T淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:RPMI no 8226细胞、EHEB细胞、HR-8348细胞
PCI-4B Cells;背景说明:喉鳞癌;淋巴结转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KYSE-410细胞、Rca-B细胞、NCIH1755细胞
BayGenomics ES cell line RRR019 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YHC444 Cells(提供STR鉴定图谱)
HIL12R1.2B10 Cells(提供STR鉴定图谱)
PCRP-ELF3-8F6 Cells(提供STR鉴定图谱)
COS1NR Cells(提供STR鉴定图谱)
HPS1330 Cells(提供STR鉴定图谱)
" "PubMed=13261081
Moore A.E., Sabachewsky L., Toolan H.W.
Culture characteristics of four permanent lines of human cancer cells.
Cancer Res. 15:598-602(1955)
PubMed=16589695; DOI=10.1073/pnas.41.7.432; PMCID=PMC528114
Puck T.T., Marcus P.I.
A rapid method for viable cell titration and clone production with HeLa cells in tissue culture: the use of X-irradiated cells to supply conditioning factors.
Proc. Natl. Acad. Sci. U.S.A. 41:432-437(1955)
PubMed=14031339
Katzberg A.A.
The effect of space flights on living human cells aboard Discoverer XVIII.
Tech. Doc. Rep. SAMTDR USAF Sch. Aerosp. Med. 62:43.1-43.4(1962)
PubMed=14185313; DOI=10.1126/science.146.3641.241
Cell Culture Collection Committee
Animal cell strains. The Cell Culture Collection Committee has assembled and certified 23 strains of animal cells.
Science 146:241-243(1964)
PubMed=5668122; DOI=10.3181/00379727-128-33119
Peterson W.D. Jr., Stulberg C.S., Swanborg N.K., Robinson A.R.
Glucose-6-phosphate dehydrogenase isoenzymes in human cell cultures determined by sucrose-agar gel and cellulose acetate zymograms.
Proc. Soc. Exp. Biol. Med. 128:772-776(1968)
DOI=10.1007/BF02618370
Stulberg C.S., Coriell L.L., Kniazeff A.J., Shannon J.E.
The animal cell culture collection.
In Vitro 5:1-16(1970)
PubMed=4942173; DOI=10.1097/00006250-197112000-00028
Jones H.W. Jr., McKusick V.A., Harper P.S., Wuu K.-D.
George Otto Gey (1899-1970). The HeLa cell and a reappraisal of its origin.
Obstet. Gynecol. 38:945-949(1971)
DOI=10.1007/978-1-4757-1647-4_13
Biedler J.L.
Chromosome abnormalities in human tumor cells in culture.
(In book chapter) Human tumor cells in vitro; Fogh J. (eds.); pp.359-394; Springer; New York; USA (1975)
PubMed=1246620; DOI=10.1126/science.1246620
Hsu S.H., Schacter B.Z., Delaney N.L., Miller T.B., McKusick V.A., Kennett R.H., Bodmer J.G., Young D., Bodmer W.F.
Genetic characteristics of the HeLa cell.
Science 191:392-394(1976)
PubMed=77569; DOI=10.1111/j.1399-0039.1978.tb01259.x
Espmark J.A., Ahlqvist-Roth L., Sarne L., Persson A.
Tissue typing of cells in culture. III. HLA antigens of established human cell lines. Attempts at typing by the mixed hemadsorption technique.
Tissue Antigens 11:279-286(1978)
PubMed=6256643; DOI=10.1038/288724a0
Day R.S. 3rd, Ziolkowski C.H.J., Scudiero D.A., Meyer S.A., Lubiniecki A.S., Girardi A.J., Galloway S.M., Bynum G.D.
Defective repair of alkylated DNA by human tumour and SV40-transformed human cell strains.
Nature 288:724-727(1980)
PubMed=6935474; DOI=10.1093/jnci/66.2.239
Wright W.C., Daniels W.P., Fogh J.
Distinction of seventy-one cultured human tumor cell lines by polymorphic enzyme analysis.
J. Natl. Cancer Inst. 66:239-247(1981)
PubMed=6358339; DOI=10.1093/jhmas/38.4.415
Brown R.W., Henderson J.H.M.
The mass production and distribution of HeLa cells at Tuskegee Institute, 1953-55.
J. Hist. Med. Allied Sci. 38:415-431(1983)
PubMed=6401685; DOI=10.1007/BF02617989
Halton D.M., Peterson W.D. Jr., Hukku B.
Cell culture quality control by rapid isoenzymatic characterization.
In Vitro 19:16-24(1983)
PubMed=6825208; DOI=10.1093/carcin/4.2.199
Yarosh D.B., Foote R.S., Mitra S., Day R.S. 3rd
Repair of O6-methylguanine in DNA by demethylation is lacking in Mer- human tumor cell strains.
Carcinogenesis 4:199-205(1983)
PubMed=2983228; DOI=10.1038/314111a0
Schwarz E., Freese U.-K., Gissmann L., Mayer W., Roggenbuck B., Stremlau A., zur Hausen H.
Structure and transcription of human papillomavirus sequences in cervical carcinoma cells.
Nature 314:111-114(1985)
PubMed=2990217; PMCID=PMC1888002
Yee C., Krishnan-Hewlett I., Baker C.C., Schlegel R., Howley P.M.
Presence and expression of human papillomavirus sequences in human cervical carcinoma cell lines.
Am. J. Pathol. 119:361-366(1985)
CLPUB00262
Gold M.
A conspiracy of cells. One woman's immortal legacy and the medical scandal it caused.
(In book) ISBN 9780887060991; pp.1-171; State University of New York Press; Albany; USA (1986)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=3028716; DOI=10.1159/000132342
Popescu N.C., DiPaolo J.A., Amsbaugh S.C.
Integration sites of human papillomavirus 18 DNA sequences on HeLa cell chromosomes.
Cytogenet. Cell Genet. 44:58-62(1987)
PubMed=3180844; DOI=10.1159/000132579
Chen T.-R.
Re-evaluation of HeLa, HeLa S3, and HEp-2 karyotypes.
Cytogenet. Cell Genet. 48:19-24(1988)
PubMed=3371749; DOI=10.1016/0090-8258(88)90029-7
Grenman S.E., Shapira A., Carey T.E.
In vitro response of cervical cancer cell lines CaSki, HeLa, and ME-180 to the antiestrogen tamoxifen.
Gynecol. Oncol. 30:228-238(1988)
PubMed=1522048; DOI=10.1007/BF02634140
Kataoka E., Honma M., Ohnishi K., Sofuni T., Mizusawa H.
Application of highly polymorphic DNA markers to the identification of HeLa cell sublines.
In Vitro Cell. Dev. Biol. Anim. 28:553-556(1992)
PubMed=8380785; DOI=10.1006/gyno.1993.1016
Iwasaka T., Oh-uchida M., Matsuo N., Yokoyama M., Fukuda K., Hara K., Fukuyama K., Hori K., Sugimori H.
Correlation between HPV positivity and state of the p53 gene in cervical carcinoma cell lines.
Gynecol. Oncol. 48:104-109(1993)
PubMed=9892199
Macville M.V.E., Schrock E., Padilla-Nash H.M., Keck C., Ghadimi B.M., Zimonjic D.B., Popescu N.C., Ried T.
Comprehensive and definitive molecular cytogenetic characterization of HeLa cells by spectral karyotyping.
Cancer Res. 59:141-150(1999)
PubMed=10423141; DOI=10.1099/0022-1317-80-7-1725
Meissner J.D.
Nucleotide sequences and further characterization of human papillomavirus DNA present in the CaSki, SiHa and HeLa cervical carcinoma cell lines.
J. Gen. Virol. 80:1725-1733(1999)
PubMed=11416159; DOI=10.1073/pnas.121616198; PMCID=PMC35459
Masters J.R.W., Thomson J.A., Daly-Burns B., Reid Y.A., Dirks W.G., Packer P., Toji L.H., Ohno T., Tanabe H., Arlett C.F., Kelland L.R., Harrison M., Virmani A.K., Ward T.H., Ayres K.L., Debenham P.G.
Short tandem repeat profiling provides an international reference standard for human cell lines.
Proc. Natl. Acad. Sci. U.S.A. 98:8012-8017(2001)
PubMed=11668190; DOI=10.1177/002215540104901105
Quentmeier H., Osborn M., Reinhardt J., Zaborski M., Drexler H.G.
Immunocytochemical analysis of cell lines derived from solid tumors.
J. Histochem. Cytochem. 49:1369-1378(2001)
PubMed=12001993; DOI=10.1038/nrc775
Masters J.R.W.
HeLa cells 50 years on: the good, the bad and the ugly.
Nat. Rev. Cancer 2:315-319(2002)
PubMed=12793746; DOI=10.1269/jrr.43.s133
Ohnishi T., Ohnishi K., Takahashi A., Taniguchi Y., Sato M., Nakano T., Nagaoka S.
Detection of DNA damage induced by space radiation in Mir and space shuttle.
J. Radiat. Res. 43 Suppl. 1:S133-S136(2002)
PubMed=12661003; DOI=10.1002/gcc.10196
Seitz S., Wassmuth P., Plaschke J., Schackert H.K., Karsten U., Santibanez-Koref M.F., Schlag P.M., Scherneck S.
Identification of microsatellite instability and mismatch repair gene mutations in breast cancer cell lines.
Genes Chromosomes Cancer 37:29-35(2003)
PubMed=15302935; DOI=10.1073/pnas.0404720101; PMCID=PMC514446
Beausoleil S.A., Jedrychowski M.P., Schwartz D., Elias J.E., Villen J., Li J.-X., Cohn M.A., Cantley L.C., Gygi S.P.
Large-scale characterization of HeLa cell nuclear phosphoproteins.
Proc. Natl. Acad. Sci. U.S.A. 101:12130-12135(2004)
PubMed=15531914; DOI=10.1038/sj.onc.1208235
Baldus S.E., Schwarz E., Lohrey C., Zapatka M., Landsberg S., Hahn S.A., Schmidt D., Dienes H.-P., Schmiegel W.H., Schwarte-Waldhoff I.
Smad4 deficiency in cervical carcinoma cells.
Oncogene 24:810-819(2005)
PubMed=15901131; DOI=10.1016/j.prp.2005.01.002
Murai Y., Hayashi S., Takahashi H., Tsuneyama K., Takano Y.
Correlation between DNA alterations and p53 and p16 protein expression in cancer cell lines.
Pathol. Res. Pract. 201:109-115(2005)
PubMed=17311676; DOI=10.1186/1471-2164-8-53; PMCID=PMC1805756
Kloth J.N., Oosting J., van Wezel T., Szuhai K., Knijnenburg J., Gorter A., Kenter G.G., Fleuren G.J., Jordanova E.S.
Combined array-comparative genomic hybridization and single-nucleotide polymorphism-loss of heterozygosity analysis reveals complex genetic alterations in cervical cancer.
BMC Genomics 8:53.1-53.13(2007)
PubMed=19450234; DOI=10.2144/000113089; PMCID=PMC2696096
Rahbari R., Sheahan T., Modes V., Collier P., Macfarlane C.M., Badge R.M.
A novel L1 retrotransposon marker for HeLa cell line identification.
BioTechniques 46:277-284(2009)
PubMed=19722756; DOI=10.5858/133.9.1463
Lucey B.P., Nelson-Rees W.A., Hutchins G.M.
Henrietta Lacks, HeLa cells, and cell culture contamination.
Arch. Pathol. Lab. Med. 133:1463-1467(2009)
CLPUB00377
Skloot R.L.
The immortal life of Henrietta Lacks.
(In book) ISBN 9781400052172; pp.1-400; Random House; New York; USA (2010)
CLPUB00468
Javitt G.H.
Why not take all of me? Reflections on the immortal life of Henrietta Lacks and the status of participants in research using human specimens.
Minn. J. Law Sci. Technol. 11:713-755(2010)
PubMed=19941903; DOI=10.1016/j.jviromet.2009.11.022
Karger A., Bettin B., Lenk M., Mettenleiter T.C.
Rapid characterisation of cell cultures by matrix-assisted laser desorption/ionisation mass spectrometric typing.
J. Virol. Methods 164:116-121(2010)
PubMed=21269460; DOI=10.1186/1752-0509-5-17; PMCID=PMC3039570
Burkard T.R., Planyavsky M., Kaupe I., Breitwieser F.P., Burckstummer T., Bennett K.L., Superti-Furga G., Colinge J.
Initial characterization of the human central proteome.
BMC Syst. Biol. 5:17.1-17.13(2011)
PubMed=22068331; DOI=10.1038/msb.2011.81; PMCID=PMC3261714
Nagaraj N., Wisniewski J.R., Geiger T., Cox J., Kircher M., Kelso J., Paabo S., Mann M.
Deep proteome and transcriptome mapping of a human cancer cell line.
Mol. Syst. Biol. 7:548-548(2011)
PubMed=21937730; DOI=10.1074/mcp.M111.011429; PMCID=PMC3316722
Boisvert F.-M., Ahmad Y., Gierlinski M., Charriere F., Lamont D., Scott M., Barton G., Lamond A.I.
A quantitative spatial proteomics analysis of proteome turnover in human cells.
Mol. Cell. Proteomics 11:M111.011429-M111.011429(2012)
PubMed=22278370; DOI=10.1074/mcp.M111.014050; PMCID=PMC3316730
Geiger T., Wehner A., Schaab C., Cox J., Mann M.
Comparative proteomic analysis of eleven common cell lines reveals ubiquitous but varying expression of most proteins.
Mol. Cell. Proteomics 11:M111.014050-M111.014050(2012)
PubMed=22412903; DOI=10.1371/journal.pone.0032667; PMCID=PMC3296745
Vazquez-Mena O., Medina-Martinez I., Juarez-Torres E., Barron V., Espinosa A., Villegas-Sepulveda N., Gomez-Laguna L., Nieto-Martinez K., Orozco L., Roman-Bassaure E., Munoz Cortez S., Borges Ibanez M., Venegas-Vega C.A., Guardado-Estrada M., Rangel-Lopez A., Kofman S., Berumen J.
Amplified genes may be overexpressed, unchanged, or downregulated in cervical cancer cell lines.
PLoS ONE 7:E32667-E32667(2012)
CLPUB00507
Pultarova T.
HeLa cells: immortal space travellers.
Space Saf. Mag. 7:10-12(2013)
PubMed=23205564; DOI=10.1021/pr300859k
Malerod H., Graham R.L.J., Sweredoski M.J., Hess S.
Comprehensive profiling of N-linked glycosylation sites in HeLa cells using hydrazide enrichment.
J. Proteome Res. 12:248-259(2013)
PubMed=23336012; DOI=10.1371/journal.pone.0054672; PMCID=PMC3545996
Horvat T., Dezeljin M., Redzic I., Barisic D., Herak Bosnar M., Lauc G., Zoldos V.
Reversibility of membrane N-glycome of HeLa cells upon treatment with epigenetic inhibitors.
PLoS ONE 8:E54672-E54672(2013)
PubMed=23925224; DOI=10.1038/500141a; PMCID=PMC5101952
Hudson K.L., Collins F.S.
Biospecimen policy: family matters.
Nature 500:141-142(2013)
PubMed=23925245; DOI=10.1038/nature12064; PMCID=PMC3740412
Adey A.C., Burton J.N., Kitzman J.O., Hiatt J.B., Lewis A.P., Martin B.K., Qiu R.-L., Lee C., Shendure J.
The haplotype-resolved genome and epigenome of the aneuploid HeLa cancer cell line.
Nature 500:207-211(2013)
PubMed=24134916; DOI=10.1186/1755-8166-6-44; PMCID=PMC3879223
McCormack A., Fan J.-L., Duesberg M., Bloomfield M., Fiala C., Duesberg P.H.
Individual karyotypes at the origins of cervical carcinomas.
Mol. Cytogenet. 6:44.1-44.23(2013)
PubMed=24618588; DOI=10.1371/journal.pone.0091433; PMCID=PMC3950186
Chernobrovkin A.L., Zubarev R.A.
Detection of viral proteins in human cells lines by xeno-proteomics: elimination of the last valid excuse for not testing every cellular proteome dataset for viral proteins.
PLoS ONE 9:E91433-E91433(2014)
PubMed=24696503; DOI=10.1074/mcp.M113.035170; PMCID=PMC4047476
Guo X.-F., Trudgian D.C., Lemoff A., Yadavalli S., Mirzaei H.
Confetti: a multiprotease map of the HeLa proteome for comprehensive proteomics.
Mol. Cell. Proteomics 13:1573-1584(2014)
PubMed=24908793
Gilgenkrantz S.
Sixty years of HeLa cell cultures.
Hist. Sci. Med. 48:139-144(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
CLPUB00376
Verspaget C.J.
Unruly bodies: monstrous readings of biotechnology.
Thesis PhD (2015); Curtin University; Perth; Australia
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25807930; DOI=10.1002/anie.201500342; PMCID=PMC4471546
Broncel M., Serwa R.A., Ciepla P., Krause E., Dallman M.J., Magee A.I., Tate E.W.
Multifunctional reagents for quantitative proteome-wide analysis of protein modification in human cells and dynamic profiling of protein lipidation during vertebrate development.
Angew. Chem. Int. Ed. Engl. 54:5948-5951(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25894527; DOI=10.1371/journal.pone.0121314; PMCID=PMC4404347
Bausch-Fluck D., Hofmann A., Bock T., Frei A.P., Cerciello F., Jacobs A., Moest H., Omasits U., Gundry R.L., Yoon C., Schiess R., Schmidt A., Mirkowska P., Hartlova A.S., Van Eyk J.E., Bourquin J.-P., Aebersold R., Boheler K.R., Zandstra P.W., Wollscheid B.
A mass spectrometric-derived cell surface protein atlas.
PLoS ONE 10:E0121314-E0121314(2015)
PubMed=26554430; DOI=10.1021/acs.analchem.5b03639
Dimayacyac-Esleta B.R.T., Tsai C.-F., Kitata R.B., Lin P.-Y., Choong W.-K., Lin T.-D., Wang Y.-T., Weng S.-H., Yang P.-C., Arco S.D., Sung T.-Y., Chen Y.-J.
Rapid high-pH reverse phase stagetip for sensitive small-scale membrane proteomic profiling.
Anal. Chem. 87:12016-12023(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28078501; DOI=10.1007/s11626-016-0128-8
Ambrose C.T.
The Tissue Culture Laboratory of Dr. George Otto Gey 60 yrs ago as recalled by a former student.
In Vitro Cell. Dev. Biol. Anim. 53:467-473(2017)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28261610; DOI=10.1155/2017/4180703; PMCID=PMC5316418
Kontostathi G., Zoidakis J., Makridakis M., Lygirou V., Mermelekas G., Papadopoulos T., Vougas K., Vlamis-Gardikas A., Drakakis P., Loutradis D., Vlahou A., Anagnou N.P., Pappa K.I.
Cervical cancer cell line secretome highlights the roles of transforming growth factor-beta-induced protein ig-h3, peroxiredoxin-2, and NRF2 on cervical carcinogenesis.
BioMed Res. Int. 2017:4180703.1-4180703.15(2017)
PubMed=28601559; DOI=10.1016/j.cels.2017.05.009; PMCID=PMC5493283
Bekker-Jensen D.B., Kelstrup C.D., Batth T.S., Larsen S.C., Haldrup C., Bramsen J.B., Sorensen K.D., Hoyer S., Orntoft T.F., Lindbjerg Andersen C., Nielsen M.L., Olsen J.V.
An optimized shotgun strategy for the rapid generation of comprehensive human proteomes.
Cell Syst. 4:587-599.e4(2017)
PubMed=29156801; DOI=10.18632/oncotarget.21174; PMCID=PMC5689691
Kalu N.N., Mazumdar T., Peng S.-H., Shen L., Sambandam V., Rao X.-Y., Xi Y.-X., Li L.-R., Qi Y., Gleber-Netto F.O., Patel A., Wang J., Frederick M.J., Myers J.N., Pickering C.R., Johnson F.M.
Genomic characterization of human papillomavirus-positive and -negative human squamous cell cancer cell lines.
Oncotarget 8:86369-86383(2017)
PubMed=30175587; DOI=10.1021/acs.jproteome.8b00392
Robin T., Bairoch A., Muller M., Lisacek F., Lane L.
Large-scale reanalysis of publicly available HeLa cell proteomics data in the context of the Human Proteome Project.
J. Proteome Res. 17:4160-4170(2018)
PubMed=30778230; DOI=10.1038/s41587-019-0037-y
Liu Y.-S., Mi Y., Mueller T., Kreibich S., Williams E.G., Van Drogen A., Borel C., Frank M., Germain P.-L., Bludau I., Mehnert M., Seifert M., Emmenlauer M., Sorg I., Bezrukov F., Sloan-Bena F., Zhou H., Dehio C., Testa G., Saez-Rodriguez J., Antonarakis S.E., Hardt W.-D., Aebersold R.
Multi-omic measurements of heterogeneity in HeLa cells across laboratories.
Nat. Biotechnol. 37:314-322(2019)
PubMed=30787054; DOI=10.1158/1055-9965.EPI-18-1132; PMCID=PMC6548687
Hooker S.E. Jr., Woods-Burnham L., Bathina M., Lloyd S., Gorjala P., Mitra R., Nonn L., Kimbro K.S., Kittles R.A.
Genetic ancestry analysis reveals misclassification of commonly used cancer cell lines.
Cancer Epidemiol. Biomarkers Prev. 28:1003-1009(2019)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)
PubMed=31433507; DOI=10.15252/embj.2018100847; PMCID=PMC6826212
Hardman G., Perkins S., Brownridge P.J., Clarke C.J., Byrne D.P., Campbell A.E., Kalyuzhnyy A., Myall A., Eyers P.A., Jones A.R., Eyers C.E.
Strong anion exchange-mediated phosphoproteomics reveals extensive human non-canonical phosphorylation.
EMBO J. 38:e100847.1-e100847.22(2019)
PubMed=31790455; DOI=10.1371/journal.pone.0225466; PMCID=PMC6886862
Hu W.-E., Zhang X., Guo Q.-F., Yang J.-W., Yang Y., Wei S.-C., Su X.-D.
HeLa-CCL2 cell heterogeneity studied by single-cell DNA and RNA sequencing.
PLoS ONE 14:E0225466-E0225466(2019)
PubMed=33389257; DOI=10.1007/s10096-020-04106-0; PMCID=PMC7778494
Wurtz N., Penant G., Jardot P., Duclos N., La Scola B.
Culture of SARS-CoV-2 in a panel of laboratory cell lines, permissivity, and differences in growth profile.
Eur. J. Clin. Microbiol. Infect. Dis. 40:477-484(2021)"