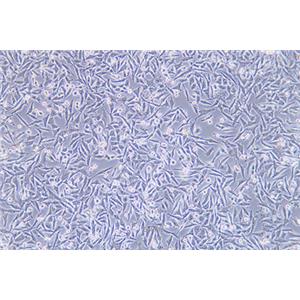
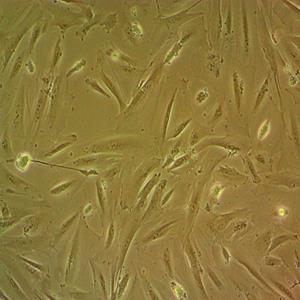
"A-375人恶性黑色素瘤细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
NCIH661 Cells;背景说明:该细胞1982年建系,源自一位患有大细胞肺癌的男性的胸腔积液。该细胞角蛋白、波形蛋白阳性。;传代方法:1:3—1:5传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:YTMLC-90细胞、H1618细胞、HUVEC-C[HUVEC]细胞
Jeko1 Cells;背景说明:一位套细胞淋巴瘤患者的巨细胞变种显示白血病转变,从其外周血单核细胞出发建立了MCL细胞株JeKo-1。 JeKo-1细胞EB病毒阴性,并表达一种B细胞表型的IgM。 细胞过表达cyclin D1, Bcl-2, c-Myc 及 Rb 蛋白。 Bcl-1/J(H)基因重排得到了PCR证实。 JeKo-1细胞在SCID小鼠中高成瘤。 [PubMed: 9753063];传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:Mel-RM细胞、MMVECs细胞、ChaGo-K1细胞
NB.4 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:FHL 124细胞、OV-90细胞、FDCP1细胞
A-375人恶性黑色素瘤细胞代次低|培养基|送STR图谱
背景信息:A375源自一位54岁女性,是Giard DJ等人建立的一系列细胞株中的一株。该细胞可在免疫抑制小鼠上成瘤,在琼脂上形成克隆。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
绝大部分细胞消化只要用胰酶润洗一遍即可:吸去胰酶后,残留的那些无法计算体积的附着在细胞表面的微量胰酶在37℃一般不到2min足够消化细胞(绝大部分1min不到)。对于这些细胞原则上不要用胰酶孵育细胞,连续这样传代,对细胞伤害很大。简单的程序是PBS润洗吸去,胰酶润洗吸去,然后37℃消化。什么算是消化好了呢?不需要把细胞全部消化成间隔分布很离散的单个圆形才算消化好了,一般你肉眼观察贴壁细胞层,只要能移动了,多半呈沙状移动,其实已经是可以了。一般能移动了,说明细胞与培养基质材料的附着已经消失了,细胞之间的附着也已经消失了,细胞已经独立分布了(虽然没有呈现很广的离散分布)。这个时候应该停止消化,不要等到看到镜下所有细胞都分离得非常好,间隙很大,才停止。细胞系在贴壁的过程中仍然会聚集,这个是贴壁培养的细胞,尤其是肿瘤细胞的一个特性,你可以尝试,准备100%的单个细胞悬液,贴壁后观察细胞,仍然是几个几个细胞聚集在一起。一些悬浮培养细胞也是如此,容易聚集,不要过几个小时就拿出来吹打成单细胞悬液。细胞只要能从基质上脱离下来,即使是成片的(比如Calu-3细胞),吹打不超过20次(一般10次即可),成小规模聚集(10个细胞左右)是正常的,不要再去延长消化时间,等待单细胞悬液出现。比较难消化的细胞:润洗方法5min还不能消化,以结肠癌细胞为例,比如:HCT15、LS411和KM12细胞,胰酶消化,一般10 cm培养皿,一次加入300ul-500ul就足够了。即使这样难消化的细胞,一般不超过5min,即可见细胞成片移动,就应该停止消化。一些正常细胞也会有难消化的时候,比如tsDC细胞,用胰酶孵育,3min左右即可看到成片沙状移动。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
A-375人恶性黑色素瘤细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
HDQP1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Tissue Culture-1细胞、HuP-T4细胞、Ect1/E6E7细胞
LICR-LON-HN6 Cells;背景说明:舌鳞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NW-38细胞、HOP-62细胞、H2023细胞
DMS-53 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代,每周2-3次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HPDEC细胞、Hs 695.T细胞、HL-7702细胞
NCI-522 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SR细胞、He La细胞、TE14细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞株(系)的使用,为医学研究和测试工作带来了大的方便。但细胞的传代是有限制的,长期连续传代的细胞,不仅消耗大量的人力和物力,而且细胞的生长与形态等会有一定退变或转化,因而细胞失去原有的遗传性,有时还会由于细胞污染而造成传代中断,种子丢失。因此,在实际工作中常需冻存一定数量的细胞,以备替换使用。细胞冷冻与复苏是细胞培养 室的常规工作和通用技术。目前,细胞冻存Zui常用的技术是冷冻保存法,主要采用加适量保护剂的缓慢冷冻法冻存细胞。细胞在不加任何保护剂的情况下直接冷冻,细胞内外的水分会很快形成冰晶,从而引起一系列不良反应。如细胞脱水使局部电解质浓度增GAO,pH值改变,部分蛋白质由于上述原因而变性,引起细胞内部空间结构紊乱,溶酶体膜由此遭到损伤而释放出溶酶体酶,使细胞内结构成分造成破坏,线粒体肿胀,功能丢失,并造成能量代谢障碍。胞膜上的类脂蛋白复合体也易破坏引起细胞膜通透性的改变,使细胞内容物丢失。如果细胞内冰晶形成较多,随冷冻温度的降低,冰晶体积膨胀造成细胞核DNA空间构型发生不可逆的损伤,而致细胞死亡。因此,细胞冷冻技术的关键是尽可能地减少细胞内水分,减少细胞内冰晶的形成。采用甘油或二甲基亚砜作保护剂,这两种物质分子量小,溶解度大,易穿透细胞,可以使冰点下降,提GAO细胞膜对水的通透性,且对细胞无明显毒性。慢速冷冻方法又可使细胞内的水分渗出细胞外,减少胞内形成冰结晶的机会,从而减少冰晶对细胞的损伤。
SNU-1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮聚集;形态特性:上皮细胞;相关产品有:MDA436细胞、U-138MG细胞、U373-MG细胞
no-11 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MiaPaca.2细胞、KMB-17细胞、HRIF细胞
MEG01 Cells;背景说明:MEG-01细胞株源自一位CML患者成巨核细胞转换期的骨髓细胞。细胞质因子VIII和表面球蛋白IIb/IIIa,-Schiff(PAS)反应,α醋酸酯酶和酸性酶阳性。髓过氧化物酶,α酯酶,化醋酸AS-D酯酶和碱性酶阴性。用单克隆抗体BA-1(抗B细胞,粒性白细胞),HPL-3(抗球蛋白IIb/IIIa)和20.3(抗单核细胞,血小板)染色成阳性。其他淋巴和骨髓类抗体成阴性。;传代方法:1:2传代。3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:OVCAR4细胞、Hs706T细胞、Murine Long bone Osteocyte-Y4细胞
U-87 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LLC-WRC 256细胞、TE-12细胞、McArdle RH-7777细胞
NRK Cells;背景说明:肾;自发永生;Osborne-Mendel;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:DC2.4细胞、Sp2/O-Ag14细胞、NCTC-3960细胞
Central Adrenergic TH-expressing a Cells;背景说明:神经;SV40转化;C57BL/6 x DBA/2;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MPC-11细胞、H-128细胞、LAPC4细胞
FRTL-5 Cells;背景说明:甲状腺;自发永生;Fischer;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Hs-675-T细胞、P3X63Ag8.653细胞、SO-RB50细胞
OSKM-1 Cells;背景说明:诱导型多能干 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CMEC/D3细胞、Hs 839.T细胞、CCD-841-CoTr细胞
DMS273 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:CF PAC-1细胞、CC-LP-I细胞、P-815细胞
MHCC97L Cells;背景说明:来源于中山医院,生长较缓慢;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MESSADX5细胞、COLO 741细胞、CEM-C7细胞
RKO_AS45 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCL-H548细胞、X63-Ag8.653细胞、Saos2细胞
Simpson Cells;背景说明:来源于一位61岁的男性浆细胞瘤患者;可产生免疫球蛋白轻链,未检测到重链。;传代方法:按1:2传代,5-6小时可以看到细胞分裂;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:RERFLCMS细胞、TE-9细胞、UMRC-2细胞
U251-MG Cells;背景说明:U-251 MG分离至一位患者的胶质母细胞瘤组织。;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:成纤维细胞样;相关产品有:Leukemia L1210细胞、DMS-153细胞、CCD33Co细胞
COLO_320DM Cells;背景说明:该细胞可产生5-羟色胺、去甲、、ACTH和甲状旁腺素。角蛋白、波形蛋白弱阳性。培养条件: RPMI 1640 10%FBS;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮+贴壁;形态特性:淋巴细胞;相关产品有:HANK-1细胞、SUDHL-8细胞、H22细胞
OCI-Ly 19 Cells;背景说明:弥漫大B淋巴瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:SK-HEP-1细胞、UCLA RO-81-A-1细胞、J 82细胞
RL-95-2 Cells;背景说明:这些细胞有α角蛋白,定义明确的连接复合体,张力丝和表面微绒毛。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SK-ES-1细胞、Y3-Ag123细胞、SUDHL2细胞
Glioma-261 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:BHP 10-3细胞、H2141细胞、COLO 205细胞
1181N1 Cells(提供STR鉴定图谱)
Abcam HeLa ATG16L2 KO Cells(提供STR鉴定图谱)
AG14828 Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRE017 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XF425 Cells(提供STR鉴定图谱)
BY00749 Cells(提供STR鉴定图谱)
CTUi001-A Cells(提供STR鉴定图谱)
F180 Cells(提供STR鉴定图谱)
GM07492 iPSC Cells(提供STR鉴定图谱)
MEF Cells;背景说明:取孕9天的615小鼠胚胎,去除脑、心脏等培养建立。该细胞可用作饲养层细胞,支持胚胎干细胞ES的生长并维持ES未分化的状态。当作为饲养层细胞时,MEF需经处理停止生长。建议作为ES细胞的饲养层时,MEF不要超过6代。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:OKAC1细胞、SNT-8细胞、C8-D1A细胞
A-375人恶性黑色素瘤细胞代次低|培养基|送STR图谱
NCIH1341 Cells;背景说明:详见相关文献介绍;传代方法:3-4天换液1次。;生长特性:悬浮生长;形态特性:圆形细胞;相关产品有:L5178-R细胞、WRL68细胞、HCC-827细胞
HFL Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:HeLaS3细胞、hTERT RPE-1细胞、SKLU-1细胞
CNE-2Z Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Centre Antoine Lacassagne-27细胞、WM35细胞、Line 870 Emory University-4细胞
U 266 Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,2-3天传一代;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:SUDHL-4细胞、AML-EOL-1细胞、HCS-2/8细胞
RSC 96 Cells;背景说明:雪旺细胞;自发永生;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Ej138细胞、SKMEL-31细胞、IEC 18细胞
AZc6#25 Cells(提供STR鉴定图谱)
MLE12 Cells;背景说明:肺;上皮细胞;SV40转化;FVB/N;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:KATO 3细胞、Hs 606.T细胞、SK-ES1细胞
A9 (Hamprecht) Cells;背景说明:皮下结缔组织;自发永生;雄性;C3H/An;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HLF细胞、CFSC-2G细胞、HEK293S细胞
CHP 100 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PANC3.27细胞、RWPE-1细胞、Hs-611-T细胞
NPC-TW01 Cells;背景说明:鼻咽癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Pfeiffer细胞、H1770细胞、C57/B6-L细胞
BCP-1 Cells;背景说明:详见相关文献介绍;传代方法:维持细胞浓度在2×105/ml-1×106/ml;根据细胞浓度每2-3天补液1次。;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:P3X63Ag8-6-5-3细胞、High-5细胞、H-810细胞
CAL-120 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ishikawa细胞、RAT2细胞、MBT-2细胞
CAL-120 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Ishikawa细胞、RAT2细胞、MBT-2细胞
Daudi Cells;背景说明:1967年,该细胞系KleinE和KleinG建系,源于一名16岁患有Burkitt's淋巴瘤的黑人男性,beta-2-微球蛋白阴性,表达EBNA,VCA,sIg。该细胞携带EB病毒,是一个典型的B淋巴母细胞系,可用于白血病发病机制的研究。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:SW 837细胞、SK-Mel 1细胞、NKM1细胞
GM10376 Cells(提供STR鉴定图谱)
HAP1 CBX1 (-) 2 Cells(提供STR鉴定图谱)
S3 HeLa Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MDA-MB 231细胞、MPVECs细胞、Melan-a细胞
P3/NSI/1-AG4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:SW 1088细胞、Alexander细胞、CAL-148细胞
Swiss-3T3 Cells;背景说明:3T3细胞株是1962年Todaro G和Green H从分离的瑞士小鼠胚胎中建立的;该细胞的生长受接触性抑制,汇合状态的单层细胞密度为40000个细胞/平方厘米;检测结果显示该细胞鼠痘病毒阴性;在中生长较好,在某些玻璃表面上可能状态不佳;细胞生长饱和时其密度可以达到约50000 cells/cm2。;传代方法:1:3传代;3-4天1次。;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:H-1688细胞、Capan2细胞、HCC1395细胞
hCMEC/D3 Cells;背景说明:脑微血管;内皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SACC-83细胞、EBTr细胞、MN9D细胞
ML-2 Cells;背景说明:急性髓单核细胞白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CAMA细胞、H2452细胞、NCIH508细胞
143B Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:5传代;每周换液2-3次;生长特性:贴壁生长;形态特性:混合型;相关产品有:JVM2细胞、Hs-695-T细胞、V 79-4细胞
526 Cells;背景说明:黑色素瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SK-RC-52细胞、LUDLU-1细胞、H548细胞
Jurkat E6.1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:H-1869细胞、H2291细胞、NCI522细胞
HG03760 Cells(提供STR鉴定图谱)
IGRT-27 Cells(提供STR鉴定图谱)
LS801 Cells(提供STR鉴定图谱)
ND00182 Cells(提供STR鉴定图谱)
PC-6/SN2-5 Cells(提供STR鉴定图谱)
TM-791 Cells(提供STR鉴定图谱)
UCT0144 Cells(提供STR鉴定图谱)
HEV0284 Cells(提供STR鉴定图谱)
BI-Mel Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:KHYG-1细胞、SkChA1细胞、SW.620细胞
SUP-T1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:N-2a细胞、A-431细胞、SN12C细胞
NCI-H441-4 Cells;背景说明:NCI-H441建系于1982年(A.F.Gazdar,etal.)。该细胞分离自一名肺腺癌患者的心包液。该细胞能在半固体琼脂糖中形成克隆,并能表达肺泡表面活性蛋白A。该细胞在有血清培养基中倍增时间为58小时,在无血清培养基中倍增时间为99-138小时。;传代方法:1:3传代,2-3天传一代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:H1975细胞、BNL.1ME A.7R.1细胞、Human Corneal Endothelial Cells-12细胞
T/GHA-VSMC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SGC-7901细胞、NCIH441细胞、KOSC2细胞
UCLA NPA-87-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SNU-16细胞、MHCC97-L细胞、HCC78细胞
UCLA NPA-87-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SNU-16细胞、MHCC97-L细胞、HCC78细胞
Leukemia 1210 Cells;背景说明:该细胞源于用0.2%甲基胆蒽(溶解)涂抹雌性小鼠的皮肤诱发的肿瘤,鼠痘病毒阴性。;传代方法:1:2传代;生长特性:悬浮生长;形态特性:淋巴母细胞样;相关产品有:293T细胞、CL 1-0细胞、U-2OS细胞
P3/NS-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:3LL细胞、SUM-102PT细胞、IPLB-Sf21细胞
MEF Cells;背景说明:取孕9天的615小鼠胚胎,去除脑、心脏等培养建立。该细胞可用作饲养层细胞,支持胚胎干细胞ES的生长并维持ES未分化的状态。当作为饲养层细胞时,MEF需经处理停止生长。建议作为ES细胞的饲养层时,MEF不要超过6代。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:成纤维细胞样;相关产品有:OKAC1细胞、SNT-8细胞、C8-D1A细胞
MDAMB435S Cells;背景说明:MDA-MB-435S是一种纺锤形的细胞,1976年由其亲本(435)中筛选得到。435是从31岁的转移性乳腺导管腺癌女性患者胸水中分离得到。当用荧光染料对微管蛋白进行染色时亲本细胞显现散布特征(II型)。最近通过cDNA阵列研究表明,亲本(MDA-MB-435)可归入黑素瘤起源。;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:纺锤形;相关产品有:Caco-2/BBe 1细胞、TOV21细胞、NCI-SNU-520细胞
UCLA SO M21 Cells;背景说明:黑色素瘤;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MAVER-1细胞、Mono Mac 6细胞、SF-295细胞
U-hth74 Cells;背景说明:未分化甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MEL-526细胞、HuT-78细胞、P3HR1细胞
JROECL21 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OsA-CL细胞、HCMEC(BL12-H)细胞、EBC-1/original细胞
GDM-1 Cells;背景说明:详见相关文献介绍;传代方法:2-3天换液1次。;生长特性:悬浮生长;形态特性:淋巴母细胞样 ;相关产品有:MCF7-GFP细胞、B16 melanoma F10细胞、UWB1289细胞
HT-55 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:BT-474细胞、NS1/1-Ag4.1细胞、HLEB-3细胞
SCD16-2E Cells(提供STR鉴定图谱)
LK-2 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:RAEC细胞、COLO 829细胞、A1847细胞
VMM39 Cells;背景说明:黑色素瘤;神经节转移;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:A498细胞、SK-ML2细胞、KCL22细胞
Roswell Park Memorial Institute 6666 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:BC-019细胞、UM2细胞、LU-65M细胞
TU 686 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:NF639细胞、KOPN8细胞、N-Tera-2细胞
NCI-H1838 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:4传代;每周换液2次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:A3细胞、Strain V细胞、B16细胞
NS-1-Ag4-1 Cells;背景说明:这是P3X63Ag8(ATCCTIB-9)的一个不分泌克隆。Kappa链合成了但不分泌。能抗0.1mM8-氮杂鸟嘌呤但不能在HAT培养基中生长。据报道它是由于缺失了3-酮类固醇还原酶活性的胆固醇营养缺陷型。检测表明肢骨发育畸形病毒(鼠痘)阴性。;传代方法:1:2传代,3天内可长满。;生长特性:悬浮生长;形态特性:淋巴母细胞;相关产品有:H661细胞、Tb1Lu细胞、OUMS27细胞
A-375人恶性黑色素瘤细胞代次低|培养基|送STR图谱
HCT FET Cells;背景说明:结肠癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NBLAN5T细胞、MDCK supertube细胞、HO1-N-1细胞
HB 611 Cells;背景说明:肝母细胞癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:RPMI #1846细胞、CAL12T细胞、H2405细胞
PC3-M Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:Hs 611.T细胞、H-647细胞、HO-8910细胞
QGY-7701 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟,1:2,3天内可长满;生长特性:贴壁生长;形态特性:上皮样;相关产品有:CMT 93细胞、SK-N-BE2C细胞、MUVEC细胞
OV 1063 Cells;背景说明:卵巢上皮细胞癌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Pt K2 (NBL-5)细胞、SK-MES1细胞、IR 983F细胞
ISHI Cells;背景说明:详见相关文献介绍;传代方法:1:3传代,3-4天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:SF17细胞、HUASMC细胞、H322细胞
CZ-1 Cells;背景说明:骨髓瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:B16F0细胞、RT-4细胞、Mel RM细胞
COLO-829 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:成纤维细胞;相关产品有:SR-786细胞、PBMC细胞、MHHCALL2细胞
BayGenomics ES cell line RRA122 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XE220 Cells(提供STR鉴定图谱)
CT.4R Cells(提供STR鉴定图谱)
Mfn1/Mfn2-null MEFs Cells(提供STR鉴定图谱)
Sog9 Cells(提供STR鉴定图谱)
LR-414 Cells(提供STR鉴定图谱)
" "PubMed=833871; DOI=10.1093/jnci/58.2.209
Fogh J., Wright W.C., Loveless J.D.
Absence of HeLa cell contamination in 169 cell lines derived from human tumors.
J. Natl. Cancer Inst. 58:209-214(1977)
DOI=10.1007/BF00199208
Bruggen J., Sorg C., Macher E.
Membrane associated antigens of human malignant melanoma V: Serological typing of cell lines using antisera from nonhuman primates.
Cancer Immunol. Immunother. 5:53-62(1978)
PubMed=77569; DOI=10.1111/j.1399-0039.1978.tb01259.x
Espmark J.A., Ahlqvist-Roth L., Sarne L., Persson A.
Tissue typing of cells in culture. III. HLA antigens of established human cell lines. Attempts at typing by the mixed hemadsorption technique.
Tissue Antigens 11:279-286(1978)
PubMed=375235; DOI=10.1073/pnas.76.3.1288; PMCID=PMC383236
Sherwin S.A., Sliski A.H., Todaro G.J.
Human melanoma cells have both nerve growth factor and nerve growth factor-specific receptors on their cell surfaces.
Proc. Natl. Acad. Sci. U.S.A. 76:1288-1292(1979)
PubMed=22282976; DOI=10.1093/carcin/1.1.21
Day R.S. 3rd, Ziolkowski C.H.J., Scudiero D.A., Meyer S.A., Mattern M.R.
Human tumor cell strains defective in the repair of alkylation damage.
Carcinogenesis 1:21-32(1980)
DOI=10.1007/BF00205883
Bruggen J., Macher E., Sorg C.
Expression of surface antigens and its relation to parameters of malignancy in human malignant melanoma.
Cancer Immunol. Immunother. 10:121-127(1981)
PubMed=6954533; DOI=10.1073/pnas.79.7.2194; PMCID=PMC346157
Westin E.H., Gallo R.C., Arya S.K., Eva A., Souza L.M., Baluda M.A., Aaronson S.A., Wong-Staal F.
Differential expression of the amv gene in human hematopoietic cells.
Proc. Natl. Acad. Sci. U.S.A. 79:2194-2198(1982)
PubMed=6220172
Dracopoli N.C., Fogh J.
Polymorphic enzyme analysis of cultured human tumor cell lines.
J. Natl. Cancer Inst. 70:469-476(1983)
PubMed=6500159; DOI=10.1159/000163283
Gershwin M.E., Lentz D., Owens R.B.
Relationship between karyotype of tissue culture lines and tumorigenicity in nude mice.
Exp. Cell Biol. 52:361-370(1984)
PubMed=6584666; DOI=10.1093/jnci/72.4.913
Kozlowski J.M., Hart I.R., Fidler I.J., Hanna N.
A human melanoma line heterogeneous with respect to metastatic capacity in athymic nude mice.
J. Natl. Cancer Inst. 72:913-917(1984)
PubMed=3518877; DOI=10.3109/07357908609038260
Fogh J.
Human tumor lines for cancer research.
Cancer Invest. 4:157-184(1986)
PubMed=1832891; DOI=10.1016/0277-5379(91)90277-K
Hansson J., Fichtinger-Schepman A.M.J., Edgren M.R., Ringborg U.
Comparative study of two human melanoma cell lines with different sensitivities to mustine and cisplatin.
Eur. J. Cancer 27:1039-1045(1991)
PubMed=9670966; DOI=10.4049/jimmunol.161.2.877
Bettinotti M.P., Kim C.J., Lee K.-H., Roden M., Cormier J.N., Panelli M.C., Parker K.K., Marincola F.M.
Stringent allele/epitope requirements for MART-1/Melan A immunodominance: implications for peptide-based immunotherapy.
J. Immunol. 161:877-889(1998)
PubMed=9973934; DOI=10.1016/S0165-4608(98)00122-8
Nelson M.A., Ariza M.E., Yang J.-M., Thompson F.H., Taetle R., Trent J.M., Wymer J., Massey-Brown K.S., Broome-Powell M., Easton J., Lahti J.M., Kidd V.J.
Abnormalities in the p34cdc2-related PITSLRE protein kinase gene complex (CDC2L) on chromosome band 1p36 in melanoma.
Cancer Genet. Cytogenet. 108:91-99(1999)
PubMed=10497214; DOI=10.1074/jbc.274.40.28505
Ariza M.E., Broome-Powell M., Lahti J.M., Kidd V.J., Nelson M.A.
Fas-induced apoptosis in human malignant melanoma cell lines is associated with the activation of the p34(cdc2)-related PITSLRE protein kinases.
J. Biol. Chem. 274:28505-28513(1999)
PubMed=12068308; DOI=10.1038/nature00766
Davies H.R., Bignell G.R., Cox C., Stephens P.J., Edkins S., Clegg S., Teague J.W., Woffendin H., Garnett M.J., Bottomley W., Davis N., Dicks E., Ewing R., Floyd Y., Gray K., Hall S., Hawes R., Hughes J., Kosmidou V., Menzies A., Mould C., Parker A., Stevens C., Watt S., Hooper S., Wilson R., Jayatilake H., Gusterson B.A., Cooper C.S., Shipley J.M., Hargrave D., Pritchard-Jones K., Maitland N.J., Chenevix-Trench G., Riggins G.J., Bigner D.D., Palmieri G., Cossu A., Flanagan A.M., Nicholson A., Ho J.W.C., Leung S.Y., Yuen S.T., Weber B.L., Seigler H.F., Darrow T.L., Paterson H.F., Marais R., Marshall C.J., Wooster R., Stratton M.R., Futreal P.A.
Mutations of the BRAF gene in human cancer.
Nature 417:949-954(2002)
PubMed=14871852; DOI=10.1158/0008-5472.CAN-03-2209
Hogan K.T., Coppola M.A., Gatlin C.L., Thompson L.W., Shabanowitz J., Hunt D.F., Engelhard V.H., Ross M.M., Slingluff C.L. Jr.
Identification of novel and widely expressed cancer/testis gene isoforms that elicit spontaneous cytotoxic T-lymphocyte reactivity to melanoma.
Cancer Res. 64:1157-1163(2004)
PubMed=15009714; DOI=10.1046/j.0022-202X.2004.22243.x; PMCID=PMC2586668
Tsao H., Goel V., Wu H., Yang G., Haluska F.G.
Genetic interaction between NRAS and BRAF mutations and PTEN/MMAC1 inactivation in melanoma.
J. Invest. Dermatol. 122:337-341(2004)
PubMed=15467732; DOI=10.1038/sj.onc.1208152
Tanami H., Imoto I., Hirasawa A., Yuki Y., Sonoda I., Inoue J., Yasui K., Misawa-Furihata A., Kawakami Y., Inazawa J.
Involvement of overexpressed wild-type BRAF in the growth of malignant melanoma cell lines.
Oncogene 23:8796-8804(2004)
PubMed=17308088; DOI=10.1158/0008-5472.CAN-06-3311
Shields J.M., Thomas N.E., Cregger M., Berger A.J., Leslie M., Torrice C., Hao H.-L., Penland S., Arbiser J.L., Scott G.A., Zhou T., Bar-Eli M., Bear J.E., Der C.J., Kaufmann W.K., Rimm D.L., Sharpless N.E.
Lack of extracellular signal-regulated kinase mitogen-activated protein kinase signaling shows a new type of melanoma.
Cancer Res. 67:1502-1512(2007)
PubMed=18172304; DOI=10.1158/0008-5472.CAN-07-1939
Sabatino M., Zhao Y.-D., Voiculescu S., Monaco A., Robbins P.F., Karai L., Nickoloff B.J., Maio M., Selleri S., Marincola F.M., Wang E.
Conservation of genetic alterations in recurrent melanoma supports the melanoma stem cell hypothesis.
Cancer Res. 68:122-131(2008)
PubMed=19727395; DOI=10.1371/journal.pone.0006888; PMCID=PMC2731225
Wadlow R.C., Wittner B.S., Finley S.A., Bergquist H., Upadhyay R., Finn S.P., Loda M., Mahmood U., Ramaswamy S.
Systems-level modeling of cancer-fibroblast interaction.
PLoS ONE 4:E6888-E6888(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=20215515; DOI=10.1158/0008-5472.CAN-09-3458; PMCID=PMC2881662
Rothenberg S.M., Mohapatra G., Rivera M.N., Winokur D., Greninger P., Nitta M., Sadow P.M., Sooriyakumar G., Brannigan B.W., Ulman M.J., Perera R.M., Wang R., Tam A., Ma X.-J., Erlander M., Sgroi D.C., Rocco J.W., Lingen M.W., Cohen E.E.W., Louis D.N., Settleman J., Haber D.A.
A genome-wide screen for microdeletions reveals disruption of polarity complex genes in diverse human cancers.
Cancer Res. 70:2158-2164(2010)
PubMed=21343389; DOI=10.1158/0008-5472.CAN-10-2958; PMCID=PMC3070783
Rose A.E., Poliseno L., Wang J.-H., Clark M., Pearlman A., Wang G.-M., Vega y Saenz de Miera E.C., Medicherla R., Christos P.J., Shapiro R., Pavlick A., Darvishian F., Zavadil J., Polsky D., Hernando E., Ostrer H., Osman I.
Integrative genomics identifies molecular alterations that challenge the linear model of melanoma progression.
Cancer Res. 71:2561-2571(2011)
PubMed=21424129; DOI=10.3892/or.2011.1220
Manca A., Sini M.C., Izzo F., Ascierto P.A., Tatangelo F., Botti G., Gentilcore G., Capone M., Mozzillo N., Rozzo C., Cossu A., Tanda F., Palmieri G.
Induction of arginosuccinate synthetase (ASS) expression affects the antiproliferative activity of arginine deiminase (ADI) in melanoma cells.
Oncol. Rep. 25:1495-1502(2011)
PubMed=21673604; DOI=10.1097/CMR.0b013e32834495c3; PMCID=PMC3131479
Orgaz J.L., Benguria A., Sanchez-Martinez C., Ladhani O., Volpert O.V., Jimenez B.
Changes in the gene expression profile of A375 human melanoma cells induced by overexpression of multifunctional pigment epithelium-derived factor.
Melanoma Res. 21:285-297(2011)
PubMed=21857157; DOI=10.4161/cc.10.17.17068; PMCID=PMC5479465
Caputo E., Maiorana L., Vasta V., Pezzino F.M., Sunkara S., Wynne K., Elia G., Marincola F.M., McCubrey J.A., Libra M., Travali S., Kane M.
Characterization of human melanoma cell lines and melanocytes by proteome analysis.
Cell Cycle 10:2924-2936(2011)
PubMed=22178978; DOI=10.1016/j.freeradbiomed.2011.11.019
Swalwell H., Latimer J., Haywood R.M., Birch-Machin M.A.
Investigating the role of melanin in UVA/UVB- and hydrogen peroxide-induced cellular and mitochondrial ROS production and mitochondrial DNA damage in human melanoma cells.
Free Radic. Biol. Med. 52:626-634(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=23039341; DOI=10.1186/1476-4598-11-75; PMCID=PMC3554420
Byron S.A., Loch D.C., Wellens C.L., Wortmann A., Wu J.-Y., Wang J., Nomoto K., Pollock P.M.
Sensitivity to the MEK inhibitor E6201 in melanoma cells is associated with mutant BRAF and wildtype PTEN status.
Mol. Cancer 11:75.1-75.15(2012)
PubMed=24581590; DOI=10.1016/j.jdermsci.2014.01.006
Gehrke S., Otsuka A., Huber R., Meier B., Kistowska M., Fenini G., Cheng P., Dummer R., Kerl K., Contassot E., French L.E.
Metastatic melanoma cell lines do not secrete IL-1beta but promote IL-1beta production from macrophages.
J. Dermatol. Sci. 74:167-169(2014)
PubMed=25056119; DOI=10.1158/0008-5472.CAN-14-1232-T; PMCID=PMC4167745
Watson I.R., Li L.-R., Cabeceiras P.K., Mahdavi M., Gutschner T., Genovese G., Wang G.-C., Fang Z.-N., Tepper J.M., Stemke-Hale K., Tsai K.Y., Davies M.A., Mills G.B., Chin L.
The RAC1 P29S hotspot mutation in melanoma confers resistance to pharmacological inhibition of RAF.
Cancer Res. 74:4845-4852(2014)
PubMed=25960936; DOI=10.4161/21624011.2014.954893; PMCID=PMC4355981
Boegel S., Lower M., Bukur T., Sahin U., Castle J.C.
A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines.
OncoImmunology 3:e954893.1-e954893.12(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=26405815; DOI=10.1371/journal.pone.0138210; PMCID=PMC4583389
Capaldo B.J., Roller D.G., Axelrod M.J., Koeppel A.F., Petricoin E.F. 3rd, Slingluff C.L. Jr., Weber M.J., Mackey A.J., Gioeli D., Bekiranov S.
Systems analysis of adaptive responses to MAP kinase pathway blockade in BRAF mutant melanoma.
PLoS ONE 10:E0138210-E0138210(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26673621; DOI=10.18632/oncotarget.6548; PMCID=PMC4823068
Roller D.G., Capaldo B.J., Bekiranov S., Mackey A.J., Conaway M.R., Petricoin E.F. 3rd, Gioeli D., Weber M.J.
Combinatorial drug screening and molecular profiling reveal diverse mechanisms of intrinsic and adaptive resistance to BRAF inhibition in V600E BRAF mutant melanomas.
Oncotarget 7:2734-2753(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=29275043; DOI=10.1016/j.jprot.2017.12.013
Liberato T., Pessotti D.S., Fukushima I., Kitano E.S., Serrano S.M.T., Zelanis A.
Signatures of protein expression revealed by secretome analyses of cancer associated fibroblasts and melanoma cell lines.
J. Proteomics 174:1-8(2018)
PubMed=29492214; DOI=10.18632/oncotarget.23989; PMCID=PMC5823576
Sini M.C., Doneddu V., Paliogiannis P., Casula M., Colombino M., Manca A., Botti G., Ascierto P.A., Lissia A., Cossu A., Palmieri G.
Genetic alterations in main candidate genes during melanoma progression.
Oncotarget 9:8531-8541(2018)
PubMed=29605720; DOI=10.1016/j.neo.2018.02.009; PMCID=PMC5915992
Mologni L., Costanza M., Sharma G.G., Viltadi M., Massimino L., Citterio S., Purgante S., Raman H., Pirola A., Zucchetti M., Piazza R., Gambacorti-Passerini C.
Concomitant BCORL1 and BRAF mutations in vemurafenib-resistant melanoma cells.
Neoplasia 20:467-477(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=30971826; DOI=10.1038/s41586-019-1103-9
Behan F.M., Iorio F., Picco G., Goncalves E., Beaver C.M., Migliardi G., Santos R., Rao Y., Sassi F., Pinnelli M., Ansari R., Harper S., Jackson D.A., McRae R., Pooley R., Wilkinson P., van der Meer D.J., Dow D., Buser-Doepner C.A., Bertotti A., Trusolino L., Stronach E.A., Saez-Rodriguez J., Yusa K., Garnett M.J.
Prioritization of cancer therapeutic targets using CRISPR-Cas9 screens.
Nature 568:511-516(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"