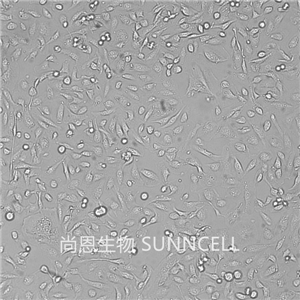
"786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱
传代比例:1:2-1:4(首次传代建议1:2)
生长特性:贴壁生长
【细胞培养经验分享】启蒙老师的重要性:一般进实验室都有师兄师姐带着做,他们就是你做细胞的启蒙老师。他们的操作手法、细节、理论讲解就成了你操作的准则,如营养液、细胞瓶的摆放位置、灭菌处理程序、开盖手法、细胞吹打手法等等。要学会他们的正确操作,在第一次的时候就要重视。像养孩子一样养细胞,细胞有时真的很脆弱,最好每天都去看看它,以防止出现培养箱缺水、缺二氧化碳、停电、温度不够等异常现象,也好及时解决这些意外,避免重复实验带来的更大痛苦。好细胞要及时保种:细胞要分批传代,这样即使有一批出了问题,还有一批备用的。像后者一般人可能不容易做到。但这是我血的教训,有一次细胞污染了,全军覆没。当时可后悔没有保种。细胞跟人一样,不同的细胞,培养特性是不一样的。培养过程中要细细体会,不同细胞系使用不同的培养基和血清。
换液周期:每周2-3次
Mevo Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:5传代,2-3天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:Sci-1细胞、BxPC-3细胞、MDA2B细胞
OVCA-432 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:MRC-5细胞、NCI-H209细胞、SCC90细胞
EFM-192A Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H524细胞、Anip 973细胞、Mv1Lu细胞
786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱
背景信息:该细胞源自一位原发性肾透明细胞癌患者。该细胞有微绒毛和桥粒,能在软琼脂上生长。此细胞生成一种PTH(甲状旁腺素)样的多肽,与乳癌和肺癌中生成的肽相似,其N端序列与PTH相似,具有PTH样活性,分子量为6000D。
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
ATCC细胞库(American Type Culture Colection),该中心一直致力于细胞分类、鉴定和保藏工作。ATCC是全球最大的生物资源保藏中心,ATCC通过行业标准产品、服务和创新解决方案支持全球学术、政府、生物技术、制药、食品、农业和工业领域的科学进步。ATCC提供的服务和定制解决方案包括细胞和微生物培养、鉴定、生物衍生物的开发和生产、性能测试和生物资源保藏服务。美国国家标准协会(ANSI)认可了ATCC标准开发组织,并制定了标准协议,以确保生物材料的可靠性和可重复性。ATCC的使命是为了获取、鉴定、保存、开发、标准化和分发生物资源和生物信息,以提高和应用生物科学知识。
产品包装:复苏发货:T25培养瓶(一瓶)或冻存发货:1ml冻存管(两支)
来源说明:细胞主要来源ATCC、ECACC、DSMZ、RIKEN等细胞库
786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱
物种来源:人源、鼠源等其它物种来源
HTSMC Cells;背景说明:气管平滑肌;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:LIM-1215细胞、Ej138细胞、HTori-3细胞
NU-GC-4 Cells;背景说明:胃癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:半贴壁;形态特性:详见产品说明书;相关产品有:TK-1细胞、Ramos G6.C10细胞、BERH-2细胞
BGC823 Cells;背景说明:建自一位62岁的胃癌患者;传代方法:1:3传代,2-3天换液一次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:KM12SM细胞、Hep G2/C3A细胞、MLMA细胞
RK 13 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:NCIH2405细胞、HNE3细胞、SNU601细胞
┈订┈购(技术服务)┈热┈线:1┈3┈6┈4┈1┈9┈3┈0┈7┈9┈1【微信同号】┈Q┈Q:3┈1┈8┈0┈8┈0┈7┈3┈2┈4;
形态特性:上皮细胞样
细胞复苏相关注意事项:1.取细胞的过程中注意带HAO防冻手套,护目镜。此项尤为重要,细胞冻存管可能漏入,解冻时冻存管中的气温急剧上升,可导致爆炸。2.冻存的问题:冻存的配置已是常识,在这里不作详述,但二甲基亚砜(DMSO )对细胞不是完全无毒副作用,在常温下,二甲基亚砜对细胞的毒副作较大,因此,必须在1-2min内使冻存完全融化。如果复苏温度太慢,会造成细胞的损伤,二甲基亚砜(DMSO)ZuiHAO选择进口产品。3.离心前须加入少量培养。细胞解冻后二甲基亚砜浓度较GAO,注意加入少量培养可稀释其浓度,以减少对细胞的损伤。4.离心问题:目前主要有两种见解。一种是解冻后的细胞悬直接吹打均匀后分装到培养瓶中进行培养,第二天换。因为离心的目的是两个,去除DMSO,去除死细胞,这个是标准流程,但对一般人来说,把握不HAO离心转速和时间,转的不够活细胞沉底的少,细胞就全被扔掉了,转过了活细胞会受压过大,死亡。此外在操作过程中容易污染,所以不推荐。另一种说法为细胞悬中含有二甲基亚砜(DMSO),DMSO对细胞有一定的毒副作用,所以须将离心后的体前倒净,且一定倒干净。我在试验中按照常规的离心分装的方法进行复苏,结果无异常。5.细胞贴壁少的问题:教科书中说明冻存细胞解冻时1ml细胞要加10ml-15ml培养,而在我的试验中的经验总结为培养基越少细胞越容易贴附。6.复苏细胞分装的问题:试验中我的经验总结为复苏1管细胞一般可分装到1-2只培养瓶中,分装过多,细胞浓度过低,不利于细胞的贴壁。7.加培养基的量放入问题:这个量的多少的把握主要涉及到的问题DMSO的浓度,从如果你加培养基的太少,那么DMSO的浓度就会比较大,就会影响细胞生长,从以前的资料来看,DMSO的浓度在小于0.5%的时候对一般细胞没有什么影响,还有一个说法是1%。所以如果你的冻存的浓度是10%DMSO的话那么加10ml以上的培养基就恰HAO稀释到了无害浓度。
RTMC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:PL 45细胞、J-111细胞、KMH-2细胞
COLO679 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:EPC细胞、Hs-940细胞、SCH细胞
NCI-H1651 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:8传代,每周换液2次;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:Caco-2/ATCC细胞、NCI/ADRRES细胞、ARO 81-1细胞
HHUA Cells;背景说明:子宫内膜癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:U266BL细胞、Hs-578Bst细胞、NK92-MI细胞
NU-GC-3 Cells;背景说明:详见相关文献介绍;传代方法:1:6传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:MGC803细胞、ACCM细胞、MC 116细胞
NT2-D1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Lewis Lung细胞、SK-MES细胞、A253细胞
BALB/3T3 Cells;背景说明:胚胎;成纤维;自发永生;雄性;BALB/c;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:GM03573细胞、HEL 92.1.7细胞、T9细胞
KFB Cells;背景说明:肾;成纤维 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:YAC-1细胞、KYSE0030细胞、TK-10细胞
RS1 Cells;背景说明:该细胞系来源于一大鼠的皮肤组织。2007年由中国科学院昆明细胞库建立。;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:HT 144细胞、MSTO-211细胞、Namalwa细胞
NCI-H28 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代,每周换液2-3次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:Nittby-Salford 1细胞、L929(NCTC)细胞、H774细胞
T 98 G Cells;背景说明:详见相关文献介绍;传代方法:按1:3传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:FHs74Int细胞、MLA144细胞、MPC-83细胞
Metastatic Variant 522 Cells;背景说明:肺腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:ZR-75-30细胞、MV3细胞、MKN-74细胞
NCI-H520 Cells;背景说明:详见相关文献介绍;传代方法:1:3-1:6传代;2-3天换液1次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs888T细胞、DC2.4细胞、HIEC-6细胞
Hs 578.T Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:OUMS-23细胞、CT26WT细胞、UMUC-14细胞
HRA 19 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Rat Lung Epithelial-6-T-antigen Negative细胞、NCI-H2444细胞、L02细胞
H69 Cells;背景说明:详见相关文献介绍;传代方法:1:2—1:4传代,每周换液2次;生长特性:悬浮生长,聚团;形态特性:聚团悬浮;相关产品有:KYSE-50细胞、Ontario Cancer Institute-Acute Myeloid Leukemia-4细胞、PG [Human lung carcinoma]细胞
OVCAR.3 Cells;背景说明:该细胞1982年由T.C. Hamilton等建系,源自一位60卵巢腺癌的腹水,是卵巢癌抗药性研究的模型。;传代方法:1:2—1:4传代,每周换液2—3次;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:Vero C1008细胞、SKNEP1细胞、Madin Darby Canine Kidney细胞
Aag2 eGFP-Piwi5 Cells(提供STR鉴定图谱)
Abcam Raji FDX1 KO Cells(提供STR鉴定图谱)
BALB/Edeltat Cells(提供STR鉴定图谱)
BayGenomics ES cell line RRR478 Cells(提供STR鉴定图谱)
BayGenomics ES cell line YTA014 Cells(提供STR鉴定图谱)
CH1 [Mouse lymphoma] Cells(提供STR鉴定图谱)
DA01643 Cells(提供STR鉴定图谱)
DD0279 Cells(提供STR鉴定图谱)
GM01680 Cells(提供STR鉴定图谱)
CCD19Lu Cells;背景说明:肺成纤维细胞;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:Kasumi-1细胞、McA-RH7777细胞、Kit-225细胞
786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱
L23/P Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:ETCC007细胞、Mink细胞、Primary Liver Carcinoma/Poliomyelitis Research Foundation/5细胞
N2a Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MPP89细胞、Gejiu Lung Carcinoma-82细胞、SNU16细胞
L6565 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:GM00637F细胞、MB157细胞、DU145细胞
MX-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HFE-145细胞、Stanford University-Diffuse Histiocytic Lymphoma-8细胞、N-2a细胞
HO8910PM Cells;背景说明:高转移卵巢癌 Cells;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:TGBC-11-TKB细胞、SK.MEL.5细胞、NCI-H220细胞
3e Cells(提供STR鉴定图谱)
SUDHL-6 Cells;背景说明:详见相关文献介绍;传代方法:1:3—1:6传代,3—4天换液1次;生长特性:悬浮生长 ;形态特性:淋巴母细胞样;相关产品有:MCF-10 Attached细胞、University of Michigan-Renal Carcinoma-2细胞、RGC-5细胞
MALME.3M Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2天换液1次。;生长特性:混合生长;形态特性:成纤维细胞;相关产品有:P3J细胞、TALL-1 [Human adult T-ALL]细胞、SP-2细胞
KYSE140 Cells;背景说明:食管鳞癌细胞;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:H1573细胞、BJ1细胞、H1299细胞
MESSA Dx5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:8传代;每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞样 ;相关产品有:PASMCS细胞、CCD19-Lu细胞、U251细胞
OCI-AML3 Cells;背景说明:急性髓系白血病;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:HCC0078细胞、UPCI:SCC90细胞、P3X63Ag8.653细胞
50.B1 Cells;背景说明:角膜上皮;Ad12-SV40;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TOV21G细胞、WIL2 S细胞、SW579细胞
50.B1 Cells;背景说明:角膜上皮;Ad12-SV40;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:TOV21G细胞、WIL2 S细胞、SW579细胞
MDA2B Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,2-3天换液1次。;生长特性:贴壁生长;形态特性:上皮细胞;相关产品有:WC00079细胞、LC1/Sq细胞、NCTC clone 1469细胞
GM50332 Cells(提供STR鉴定图谱)
HAP1 PIK3CD (-) 1 Cells(提供STR鉴定图谱)
HeLa S-3 Cells;背景说明:该细胞是1955年由PuckTT,MarcusPI和CieciuraSJ建系的,含HPV-18序列;角蛋白阳性;可用于与染色体突变、细胞营养、集落形成相关的哺乳动物细胞的克隆分析。;传代方法:1:2传代;生长特性:贴壁生长;形态特性:上皮样;相关产品有:HeLa 229细胞、RFL 6细胞、Factor Dependent Cell-Paterson 1细胞
ARO Cells;背景说明:甲状腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:2PK3细胞、MOLM-14细胞、A375.S2细胞
R3/1 Cells;背景说明:肺;Ⅰ 型上皮 Cells;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:JS1细胞、NK-10A细胞、HSC2细胞
SUM190 Cells;背景说明:乳腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:SW 780细胞、JURKAT E-61细胞、SCC15细胞
KMBC Cells;背景说明:胆管癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:MKN-28细胞、TE 85 ClF-5细胞、Neukoplast细胞
H283 Cells;背景说明:详见相关文献介绍;传代方法:每周换液2-3次。;生长特性:悬浮细胞的多细胞聚集体,和一些贴壁 Cells;形态特性:上皮细胞;相关产品有:U266 Bl细胞、beta TC6细胞、Gerner 7666细胞
NS-20Y Cells;背景说明:神经母细胞瘤;雄性;A/J;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CCRF CEM细胞、VK2细胞、U2932细胞
KMS11 Cells;背景说明:多发性骨髓瘤;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:B 95-8细胞、HCA-7细胞、RBMEC细胞
HSkMEC.2 Cells(提供STR鉴定图谱)
KOT52 Cells(提供STR鉴定图谱)
MoOV-36 Cells(提供STR鉴定图谱)
NYSCF-10005-617-617-Skin-mR-iPSC Cells(提供STR鉴定图谱)
RGE1-03 Cells(提供STR鉴定图谱)
Ubigene HEK293T RNASEL KO Cells(提供STR鉴定图谱)
WSGO Cells(提供STR鉴定图谱)
HAP1 SPIRE1 (-) 2 Cells(提供STR鉴定图谱)
Bac1 2F5 Cells;背景说明:巨噬细胞;SV40转化;BALB/c x A.CA;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:X63Ag8.653细胞、PanC1细胞、Hu-P-T4细胞
Menschliche Und Tierische Zellkulture-1 Cells;背景说明:骨髓增生异常综合征;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:NCIH1838细胞、H157细胞、SK 1细胞
SUM52 Cells;背景说明:乳腺癌;胸腔积液转移;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:PC-12细胞、NCIH2081细胞、NCI-H524细胞
L-132 Cells;背景说明:胚肺;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:CAL851细胞、EVSA/T细胞、OCI/AML5细胞
WSUDLCL2 Cells;背景说明:弥漫大B淋巴瘤;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:悬浮;形态特性:详见产品说明书;相关产品有:CCC-HPF-1细胞、TE 32细胞、LAN5细胞
HL-1 Cells;背景说明:心肌;SV40转化;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:HK-2 [Human kidney]细胞、H1792细胞、OCI-Ly7细胞
MRAEC Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LNCaP C4-2B细胞、LS-123细胞、RAMSCs细胞
GM07404 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长 ;形态特性:详见产品说明书;相关产品有:639-V细胞、Pa017C细胞、OCI/AML-5细胞
P3X63Ag8.653 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:SW 48细胞、CMT-167细胞、Beta-TC-6细胞
HDQ-P1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCT.116细胞、A-673细胞、HIEC细胞
Hi-5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HCC1438细胞、SNU-C2A细胞、As-PC1细胞
HK2 Cells;背景说明:该细胞属源于正常肾的近曲小管细胞,通过导入HPV-16 E6/E7基因而获得永生化。将含有HPV-16 E6/E7基因的重组的逆转录病毒载体pLXSN 16 E6/E7转染外生包装细胞Psi-2,Psi-2细胞产生的病毒再去感染兼嗜性包装细胞系PA317,最后将PA317产生的病毒颗粒导入正常的肾皮质近曲小管细胞。尽管pLXSN 16 E6/E7中含有新霉素抗性,但未用G418筛选转导克隆。Southern和FISH分析显示HK-2细胞来源于单克隆。PCR检测证实HK-2细胞基因组中含有E6/E7基因。;传代方法:1:4传代;2-3天换液1次;生长特性:贴壁生长;形态特性:上皮样;相关产品有:OCI-Ly 10细胞、2E8细胞、H-4-II-E细胞
J774 A1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:Hs 895.T细胞、C-26细胞、OCIAML3细胞
NHEK Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:MDA 435细胞、526 mel细胞、OCI-Ly 18细胞
MESSADX5 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:8传代;每周2-3次。;生长特性:贴壁生长;形态特性:成纤维细胞样 ;相关产品有:MALME 3M细胞、CHL/IU细胞、P3-X63.Ag8.653细胞
SUm/C Cells(提供STR鉴定图谱)
HuCC-T1 Cells;背景说明:详见相关文献介绍;传代方法:1:4传代;生长特性:贴壁生长;形态特性:上皮细胞样;相关产品有:HOP 62细胞、P3HR1-BL细胞、H-209细胞
NE-1 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:159 PT细胞、MGC803细胞、HSC-6细胞
V79-4 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:LAN5细胞、Tissue Culture-1细胞、HPAFII细胞
BEL7405 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:上皮细胞样;相关产品有:GDM1细胞、MD Anderson-Metastatic Breast-361细胞、CAL33细胞
HONE1 Cells;背景说明:鼻咽癌;男性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:T2 (174 x CEM.T2)细胞、TR-146细胞、Rin-M-5F细胞
JJN-3 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:HOC1细胞、CW2细胞、UMUC14细胞
786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱
EFO 27 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:详见产品说明书;相关产品有:TU 686细胞、Kit-225细胞、JB6 [Mouse]细胞
KP-2 Cells;背景说明:胰腺癌;女性;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:NBL-S细胞、Colo-206F细胞、NCI-SNU-16细胞
TE-9 Cells;背景说明:详见相关文献介绍;传代方法:消化3-5分钟。1:2。3天内可长满。;生长特性:贴壁生长;形态特性:上皮样;相关产品有:DMS-79细胞、MCF/Adr细胞、ECC10细胞
SK ES 01 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:5传代;每周换液2-3次;生长特性:贴壁或悬浮,详见产品说明书部分;形态特性:上皮样;相关产品有:MKN45细胞、Jurkat (clone E6-1)细胞、AML-EOL-1细胞
DMS 79 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代;每周换液2-3次。;生长特性:悬浮生长;形态特性:详见产品说明书;相关产品有:SH-SY5Y Parental细胞、GM 2132细胞、SW 620细胞
HEK-AD 293 Cells;背景说明:详见相关文献介绍;传代方法:1:2传代;生长特性:贴壁生长;形态特性:详见产品说明书;相关产品有:H184A1细胞、NPC-TW 01细胞、NS-20Y细胞
SK RC 52 Cells;背景说明:肾癌;纵隔膜转移;传代方法:1:2-1:3传代;每周换液2-3次。;生长特性:贴壁;形态特性:详见产品说明书;相关产品有:C38细胞、95C细胞、SK-MES-1细胞
TE 32 Cells;背景说明:详见相关文献介绍;传代方法:1:2-1:4传代,3-4天换液1次。;生长特性:贴壁生长;形态特性:梭型和大的多核细胞;相关产品有:NCIH810细胞、HFF-1细胞、CHO-ori细胞
BayGenomics ES cell line KST046 Cells(提供STR鉴定图谱)
BayGenomics ES cell line XB726 Cells(提供STR鉴定图谱)
CMS-5 Cells(提供STR鉴定图谱)
MANEX7374H-8G1 Cells(提供STR鉴定图谱)
s1A Cells(提供STR鉴定图谱)
YS-6MP-R-TC Cells(提供STR鉴定图谱)
" "PubMed=2041050; DOI=10.1093/jnci/83.11.757
Monks A., Scudiero D.A., Skehan P., Shoemaker R.H., Paull K.D., Vistica D.T., Hose C.D., Langley J., Cronise P., Vaigro-Wolff A., Gray-Goodrich M., Campbell H., Mayo J.G., Boyd M.R.
Feasibility of a high-flux anticancer drug screen using a diverse panel of cultured human tumor cell lines.
J. Natl. Cancer Inst. 83:757-766(1991)
PubMed=7915601; DOI=10.1038/ng0594-85
Gnarra J.R., Tory K., Weng Y.-K., Schmidt L.S., Wei M.H., Li H., Latif F., Liu S., Chen F., Duh F.-M., Lubensky I.A., Duan D.-S.R., Florence C., Pozzatti R., Walther M.M., Bander N.H., Grossman H.B., Brauch H., Pomer S., Brooks J.D., Isaacs W.B., Lerman M.I., Zbar B., Linehan W.M.
Mutations of the VHL tumour suppressor gene in renal carcinoma.
Nat. Genet. 7:85-90(1994)
PubMed=10700174; DOI=10.1038/73432
Ross D.T., Scherf U., Eisen M.B., Perou C.M., Rees C., Spellman P.T., Iyer V.R., Jeffrey S.S., van de Rijn M., Waltham M.C., Pergamenschikov A., Lee J.C.F., Lashkari D., Shalon D., Myers T.G., Weinstein J.N., Botstein D., Brown P.O.
Systematic variation in gene expression patterns in human cancer cell lines.
Nat. Genet. 24:227-235(2000)
PubMed=15585611; DOI=10.1158/1078-0432.CCR-04-0072
Tykodi S.S., Warren E.H., Thompson J.A., Riddell S.R., Childs R.W., Otterud B.E., Leppert M.F., Storb R., Sandmaier B.M.
Allogeneic hematopoietic cell transplantation for metastatic renal cell carcinoma after nonmyeloablative conditioning: toxicity, clinical response, and immunological response to minor histocompatibility antigens.
Clin. Cancer Res. 10:7799-7811(2004)
PubMed=15748285; DOI=10.1186/1479-5876-3-11; PMCID=PMC555742
Adams S., Robbins F.-M., Chen D., Wagage D., Holbeck S.L., Morse H.C. 3rd, Stroncek D., Marincola F.M.
HLA class I and II genotype of the NCI-60 cell lines.
J. Transl. Med. 3:11.1-11.8(2005)
PubMed=17088437; DOI=10.1158/1535-7163.MCT-06-0433; PMCID=PMC2705832
Ikediobi O.N., Davies H.R., Bignell G.R., Edkins S., Stevens C., O'Meara S., Santarius T., Avis T., Barthorpe S., Brackenbury L., Buck G., Butler A.P., Clements J., Cole J., Dicks E., Forbes S., Gray K., Halliday K., Harrison R., Hills K., Hinton J., Hunter C., Jenkinson A., Jones D., Kosmidou V., Lugg R., Menzies A., Miroo T., Parker A., Perry J., Raine K.M., Richardson D., Shepherd R., Small A., Smith R., Solomon H., Stephens P.J., Teague J.W., Tofts C., Varian J., Webb T., West S., Widaa S., Yates A., Reinhold W.C., Weinstein J.N., Stratton M.R., Futreal P.A., Wooster R.
Mutation analysis of 24 known cancer genes in the NCI-60 cell line set.
Mol. Cancer Ther. 5:2606-2612(2006)
PubMed=19372543; DOI=10.1158/1535-7163.MCT-08-0921; PMCID=PMC4020356
Lorenzi P.L., Reinhold W.C., Varma S., Hutchinson A.A., Pommier Y., Chanock S.J., Weinstein J.N.
DNA fingerprinting of the NCI-60 cell line panel.
Mol. Cancer Ther. 8:713-724(2009)
PubMed=20164919; DOI=10.1038/nature08768; PMCID=PMC3145113
Bignell G.R., Greenman C.D., Davies H.R., Butler A.P., Edkins S., Andrews J.M., Buck G., Chen L., Beare D., Latimer C., Widaa S., Hinton J., Fahey C., Fu B.-Y., Swamy S., Dalgliesh G.L., Teh B.T., Deloukas P., Yang F.-T., Campbell P.J., Futreal P.A., Stratton M.R.
Signatures of mutation and selection in the cancer genome.
Nature 463:893-898(2010)
PubMed=22068913; DOI=10.1073/pnas.1111840108; PMCID=PMC3219108
Gillet J.-P., Calcagno A.M., Varma S., Marino M., Green L.J., Vora M.I., Patel C., Orina J.N., Eliseeva T.A., Singal V., Padmanabhan R., Davidson B., Ganapathi R., Sood A.K., Rueda B.R., Ambudkar S.V., Gottesman M.M.
Redefining the relevance of established cancer cell lines to the study of mechanisms of clinical anti-cancer drug resistance.
Proc. Natl. Acad. Sci. U.S.A. 108:18708-18713(2011)
PubMed=22185343; DOI=10.1186/1471-2407-11-523; PMCID=PMC3292516
Matsuura K., Nakada C., Mashio M., Narimatsu T., Yoshimoto T., Tanigawa M., Tsukamoto Y., Hijiya N., Takeuchi I., Nomura T., Sato F., Mimata H., Seto M., Moriyama M.
Downregulation of SAV1 plays a role in pathogenesis of high-grade clear cell renal cell carcinoma.
BMC Cancer 11:523.1-523.10(2011)
PubMed=22347499; DOI=10.1371/journal.pone.0031628; PMCID=PMC3276511
Ruan X.-Y., Kocher J.-P.A., Pommier Y., Liu H.-F., Reinhold W.C.
Mass homozygotes accumulation in the NCI-60 cancer cell lines as compared to HapMap trios, and relation to fragile site location.
PLoS ONE 7:E31628-E31628(2012)
PubMed=22384151; DOI=10.1371/journal.pone.0032096; PMCID=PMC3285665
Lee J.-S., Kim Y.K., Kim H.J., Hajar S., Tan Y.L., Kang N.-Y., Ng S.H., Yoon C.N., Chang Y.-T.
Identification of cancer cell-line origins using fluorescence image-based phenomic screening.
PLoS ONE 7:E32096-E32096(2012)
PubMed=22460905; DOI=10.1038/nature11003; PMCID=PMC3320027
Barretina J.G., Caponigro G., Stransky N., Venkatesan K., Margolin A.A., Kim S., Wilson C.J., Lehar J., Kryukov G.V., Sonkin D., Reddy A., Liu M., Murray L., Berger M.F., Monahan J.E., Morais P., Meltzer J., Korejwa A., Jane-Valbuena J., Mapa F.A., Thibault J., Bric-Furlong E., Raman P., Shipway A., Engels I.H., Cheng J., Yu G.-Y.K., Yu J.-J., Aspesi P. Jr., de Silva M., Jagtap K., Jones M.D., Wang L., Hatton C., Palescandolo E., Gupta S., Mahan S., Sougnez C., Onofrio R.C., Liefeld T., MacConaill L.E., Winckler W., Reich M., Li N.-X., Mesirov J.P., Gabriel S.B., Getz G., Ardlie K., Chan V., Myer V.E., Weber B.L., Porter J., Warmuth M., Finan P., Harris J.L., Meyerson M.L., Golub T.R., Morrissey M.P., Sellers W.R., Schlegel R., Garraway L.A.
The Cancer Cell Line Encyclopedia enables predictive modelling of anticancer drug sensitivity.
Nature 483:603-607(2012)
PubMed=22628656; DOI=10.1126/science.1218595; PMCID=PMC3526189
Jain M., Nilsson R., Sharma S., Madhusudhan N., Kitami T., Souza A.L., Kafri R., Kirschner M.W., Clish C.B., Mootha V.K.
Metabolite profiling identifies a key role for glycine in rapid cancer cell proliferation.
Science 336:1040-1044(2012)
PubMed=22949125; DOI=10.1002/ijc.27822
Pawlowski R., Muhl S.M., Sulser T., Krek W., Moch H., Schraml P.
Loss of PBRM1 expression is associated with renal cell carcinoma progression.
Int. J. Cancer 132:E11-E17(2013)
PubMed=23856246; DOI=10.1158/0008-5472.CAN-12-3342; PMCID=PMC4893961
Abaan O.D., Polley E.C., Davis S.R., Zhu Y.-L.J., Bilke S., Walker R.L., Pineda M.A., Gindin Y., Jiang Y., Reinhold W.C., Holbeck S.L., Simon R.M., Doroshow J.H., Pommier Y., Meltzer P.S.
The exomes of the NCI-60 panel: a genomic resource for cancer biology and systems pharmacology.
Cancer Res. 73:4372-4382(2013)
PubMed=23933261; DOI=10.1016/j.celrep.2013.07.018
Moghaddas Gholami A., Hahne H., Wu Z.-X., Auer F.J., Meng C., Wilhelm M., Kuster B.
Global proteome analysis of the NCI-60 cell line panel.
Cell Rep. 4:609-620(2013)
PubMed=24279929; DOI=10.1186/2049-3002-1-20; PMCID=PMC4178206
Dolfi S.C., Chan L.L.-Y., Qiu J., Tedeschi P.M., Bertino J.R., Hirshfield K.M., Oltvai Z.N., Vazquez A.
The metabolic demands of cancer cells are coupled to their size and protein synthesis rates.
Cancer Metab. 1:20.1-20.13(2013)
PubMed=24670534; DOI=10.1371/journal.pone.0092047; PMCID=PMC3966786
Varma S., Pommier Y., Sunshine M., Weinstein J.N., Reinhold W.C.
High resolution copy number variation data in the NCI-60 cancer cell lines from whole genome microarrays accessible through CellMiner.
PLoS ONE 9:E92047-E92047(2014)
PubMed=25984343; DOI=10.1038/sdata.2014.35; PMCID=PMC4432652
Cowley G.S., Weir B.A., Vazquez F., Tamayo P., Scott J.A., Rusin S., East-Seletsky A., Ali L.D., Gerath W.F.J., Pantel S.E., Lizotte P.H., Jiang G.-Z., Hsiao J., Tsherniak A., Dwinell E., Aoyama S., Okamoto M., Harrington W., Gelfand E.T., Green T.M., Tomko M.J., Gopal S., Wong T.C., Li H.-B., Howell S., Stransky N., Liefeld T., Jang D., Bistline J., Meyers B.H., Armstrong S.A., Anderson K.C., Stegmaier K., Reich M., Pellman D., Boehm J.S., Mesirov J.P., Golub T.R., Root D.E., Hahn W.C.
Parallel genome-scale loss of function screens in 216 cancer cell lines for the identification of context-specific genetic dependencies.
Sci. Data 1:140035-140035(2014)
PubMed=25485619; DOI=10.1038/nbt.3080
Klijn C., Durinck S., Stawiski E.W., Haverty P.M., Jiang Z.-S., Liu H.-B., Degenhardt J., Mayba O., Gnad F., Liu J.-F., Pau G., Reeder J., Cao Y., Mukhyala K., Selvaraj S.K., Yu M.-M., Zynda G.J., Brauer M.J., Wu T.D., Gentleman R.C., Manning G., Yauch R.L., Bourgon R., Stokoe D., Modrusan Z., Neve R.M., de Sauvage F.J., Settleman J., Seshagiri S., Zhang Z.-M.
A comprehensive transcriptional portrait of human cancer cell lines.
Nat. Biotechnol. 33:306-312(2015)
PubMed=25877200; DOI=10.1038/nature14397
Yu M., Selvaraj S.K., Liang-Chu M.M.Y., Aghajani S., Busse M., Yuan J., Lee G., Peale F.V., Klijn C., Bourgon R., Kaminker J.S., Neve R.M.
A resource for cell line authentication, annotation and quality control.
Nature 520:307-311(2015)
PubMed=25894527; DOI=10.1371/journal.pone.0121314; PMCID=PMC4404347
Bausch-Fluck D., Hofmann A., Bock T., Frei A.P., Cerciello F., Jacobs A., Moest H., Omasits U., Gundry R.L., Yoon C., Schiess R., Schmidt A., Mirkowska P., Hartlova A.S., Van Eyk J.E., Bourquin J.-P., Aebersold R., Boheler K.R., Zandstra P.W., Wollscheid B.
A mass spectrometric-derived cell surface protein atlas.
PLoS ONE 10:E0121314-E0121314(2015)
PubMed=26589293; DOI=10.1186/s13073-015-0240-5; PMCID=PMC4653878
Scholtalbers J., Boegel S., Bukur T., Byl M., Goerges S., Sorn P., Loewer M., Sahin U., Castle J.C.
TCLP: an online cancer cell line catalogue integrating HLA type, predicted neo-epitopes, virus and gene expression.
Genome Med. 7:118.1-118.7(2015)
PubMed=26972028; DOI=10.1016/j.jprot.2016.03.008
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Identification of glycosylphosphatidylinositol-anchored proteins and omega-sites using TiO2-based affinity purification followed by hydrogen fluoride treatment.
J. Proteomics 139:77-83(2016)
PubMed=27141528; DOI=10.1016/j.dib.2016.04.001; PMCID=PMC4838930
Masuishi Y., Kimura Y., Arakawa N., Hirano H.
Data for identification of GPI-anchored peptides and omega-sites in cancer cell lines.
Data Brief 7:1302-1305(2016)
PubMed=27377824; DOI=10.1038/sdata.2016.52; PMCID=PMC4932877
Mestdagh P., Lefever S., Volders P.-J., Derveaux S., Hellemans J., Vandesompele J.
Long non-coding RNA expression profiling in the NCI60 cancer cell line panel using high-throughput RT-qPCR.
Sci. Data 3:160052-160052(2016)
PubMed=27397505; DOI=10.1016/j.cell.2016.06.017; PMCID=PMC4967469
Iorio F., Knijnenburg T.A., Vis D.J., Bignell G.R., Menden M.P., Schubert M., Aben N., Goncalves E., Barthorpe S., Lightfoot H., Cokelaer T., Greninger P., van Dyk E., Chang H., de Silva H., Heyn H., Deng X.-M., Egan R.K., Liu Q.-S., Miroo T., Mitropoulos X., Richardson L., Wang J.-H., Zhang T.-H., Moran S., Sayols S., Soleimani M., Tamborero D., Lopez-Bigas N., Ross-Macdonald P., Esteller M., Gray N.S., Haber D.A., Stratton M.R., Benes C.H., Wessels L.F.A., Saez-Rodriguez J., McDermott U., Garnett M.J.
A landscape of pharmacogenomic interactions in cancer.
Cell 166:740-754(2016)
PubMed=27807467; DOI=10.1186/s13100-016-0078-4; PMCID=PMC5087121
Zampella J.G., Rodic N., Yang W.R., Huang C.R.L., Welch J., Gnanakkan V.P., Cornish T.C., Boeke J.D., Burns K.H.
A map of mobile DNA insertions in the NCI-60 human cancer cell panel.
Mob. DNA 7:20.1-20.11(2016)
PubMed=27993170; DOI=10.1186/s12943-016-0565-8; PMCID=PMC5168717
Brodaczewska K.K., Szczylik C., Fiedorowicz M., Porta C., Czarnecka A.M.
Choosing the right cell line for renal cell cancer research.
Mol. Cancer 15:83.1-83.15(2016)
PubMed=28196595; DOI=10.1016/j.ccell.2017.01.005; PMCID=PMC5501076
Li J., Zhao W., Akbani R., Liu W.-B., Ju Z.-L., Ling S.-Y., Vellano C.P., Roebuck P., Yu Q.-H., Eterovic A.K., Byers L.A., Davies M.A., Deng W.-L., Gopal Y.N.V., Chen G., von Euw E.M., Slamon D.J., Conklin D., Heymach J.V., Gazdar A.F., Minna J.D., Myers J.N., Lu Y.-L., Mills G.B., Liang H.
Characterization of human cancer cell lines by reverse-phase protein arrays.
Cancer Cell 31:225-239(2017)
PubMed=28489074; DOI=10.1038/ncomms15165; PMCID=PMC5436135
Sinha R., Winer A.G., Chevinsky M.S., Jakubowski C., Chen Y.-B., Dong Y.-Y., Tickoo S.K., Reuter V.E., Russo P., Coleman J.A., Sander C., Hsieh J.J.-D., Hakimi A.A.
Analysis of renal cancer cell lines from two major resources enables genomics-guided cell line selection.
Nat. Commun. 8:15165.1-15165.10(2017)
PubMed=30260228; DOI=10.1021/acs.jproteome.8b00538
Knott M.E., Manzi M., Zabalegui N., Salazar M.O., Puricelli L.I., Monge M.E.
Metabolic footprinting of a clear cell renal cell carcinoma in vitro model for human kidney cancer detection.
J. Proteome Res. 17:3877-3888(2018)
PubMed=30894373; DOI=10.1158/0008-5472.CAN-18-2747; PMCID=PMC6445675
Dutil J., Chen Z.-H., Monteiro A.N.A., Teer J.K., Eschrich S.A.
An interactive resource to probe genetic diversity and estimated ancestry in cancer cell lines.
Cancer Res. 79:1263-1273(2019)
PubMed=31068700; DOI=10.1038/s41586-019-1186-3; PMCID=PMC6697103
Ghandi M., Huang F.W., Jane-Valbuena J., Kryukov G.V., Lo C.C., McDonald E.R. 3rd, Barretina J.G., Gelfand E.T., Bielski C.M., Li H.-X., Hu K., Andreev-Drakhlin A.Y., Kim J., Hess J.M., Haas B.J., Aguet F., Weir B.A., Rothberg M.V., Paolella B.R., Lawrence M.S., Akbani R., Lu Y.-L., Tiv H.L., Gokhale P.C., de Weck A., Mansour A.A., Oh C., Shih J., Hadi K., Rosen Y., Bistline J., Venkatesan K., Reddy A., Sonkin D., Liu M., Lehar J., Korn J.M., Porter D.A., Jones M.D., Golji J., Caponigro G., Taylor J.E., Dunning C.M., Creech A.L., Warren A.C., McFarland J.M., Zamanighomi M., Kauffmann A., Stransky N., Imielinski M., Maruvka Y.E., Cherniack A.D., Tsherniak A., Vazquez F., Jaffe J.D., Lane A.A., Weinstock D.M., Johannessen C.M., Morrissey M.P., Stegmeier F., Schlegel R., Hahn W.C., Getz G., Mills G.B., Boehm J.S., Golub T.R., Garraway L.A., Sellers W.R.
Next-generation characterization of the Cancer Cell Line Encyclopedia.
Nature 569:503-508(2019)"
![786-O[786-0]细胞](https://img.chemicalbook.com/SupplyImg/2023-09-28/Large/202309281112590996262.jpg)

![786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱](/SupplyImg1/2025-02-01/202502011215432874723.jpg)
![786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱](/SupplyImg1/2024-12-26/202412261010053678328.jpg)
![786-O[786-0]人肾透明细胞腺癌细胞代次低|培养基|送STR图谱](/SupplyImg1/2024-12-26/202412261010056126100.jpg)
![786-O [786-0]](https://img.chemicalbook.com/SupplyImg/2020-01-13/Large/105251542695163495.JPEG)
![786-O [786-0]](https://img.chemicalbook.com/SupplyImg/2020-01-15/Large/105187508727486592.JPEG)
![786-O [786-0]](https://img.chemicalbook.com/SupplyImg/2020-01-12/Large/105183280198157961.JPEG)
![786-O [786-0]](https://img.chemicalbook.com/SupplyImg/2019-12-18/Large/105255377846226095.JPEG)
