
4YR企业会员
发布人:上海优宁维生物科技股份有限公司
发布日期:2025/2/10 14:07:01
在处理光谱流式所产生的高维单细胞数据集时,传统流式细胞术里手动门控这种常规做法已难以满足需求,开展高维分析才是充分挖掘数据价值的关键所在。不过,当前光谱流式数据的高维分析因存在诸多尚未攻克的技术陷阱而困难重重。本文不仅对这些技术陷阱展开了深入探究,还精心构建了一套工作流程,旨在为光谱流式细胞术数据进行高维分析做好充分准备,并配套提供相应的分析工具。文中以健康对照数据为依托,详细阐释了数据质控、数据清理、转换、批次效应校正、子采样、聚类以及数据集成等一系列重要概念。此外,基于此前已发布的软件包,整合形成了一个基于 R 的集合以供使用。该工作流程能够有力地辅助光谱流式细胞术的使用者收获有效且具备可重复性的分析成果。
光谱流式数据分析工作流程图
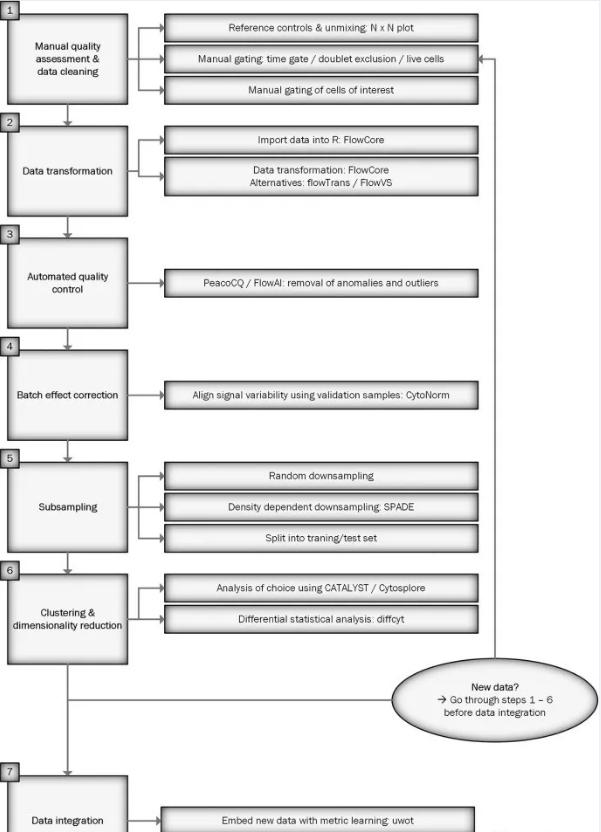
手动质检和清洁数据
挑选适宜的单染参照光谱对照以精准解混光谱数据,抑或进一步实施手动微调矫正操作。
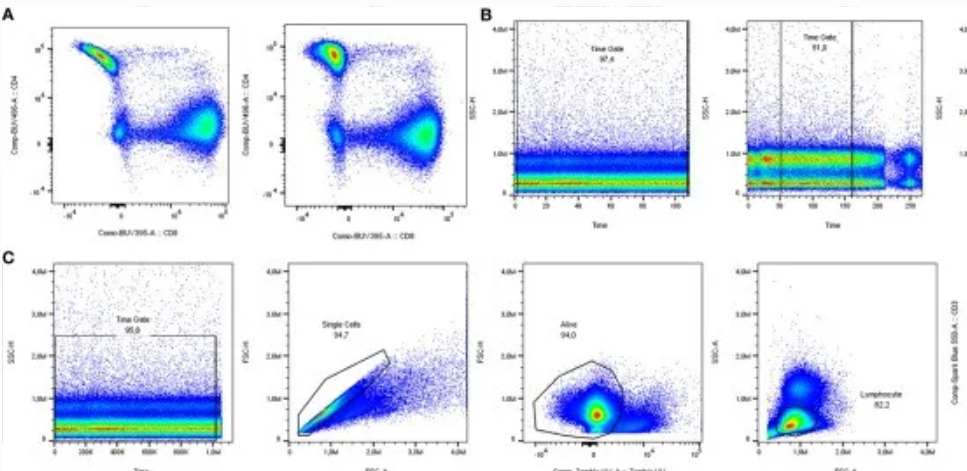
在数据清理环节,可利用时间门来剔除因流速不一致而产生的噪声。除此之外,还需把双连体以及死细胞排除在外,对于那些活性较低且流速变化幅度较大的样品数据,同样要将其排除在后续的进一步分析范围之外。
本文着重分析的是冻存外周血单个核细胞(PBMC)中的 T 细胞,在选择活细胞比例时,要求至少达到 80%,而这一临界值的确定会受到样品、靶细胞以及标记物类型等因素的影响。
随后,把靶细胞群的数据导出,以便用于后续的自动分析流程当中。在本文的研究中,靶细胞为 CD3 + T 细胞(具体筛选条件为时间门、单细胞、活细胞、淋巴细胞以及 CD3 + 标记),并将这些符合条件的靶细胞数据保存为新的 FCS 3.1 文件。
导入和转换数据
将导出的 FCS 3.1 文件借助 FlowCore 软件包导入至 R 语言环境之中。
在 “Read.FCS” 与 “Read.FlowSet” 函数里合理设置相关选项参数,以此避免极阳荧光强度值的数据出现截断现象。
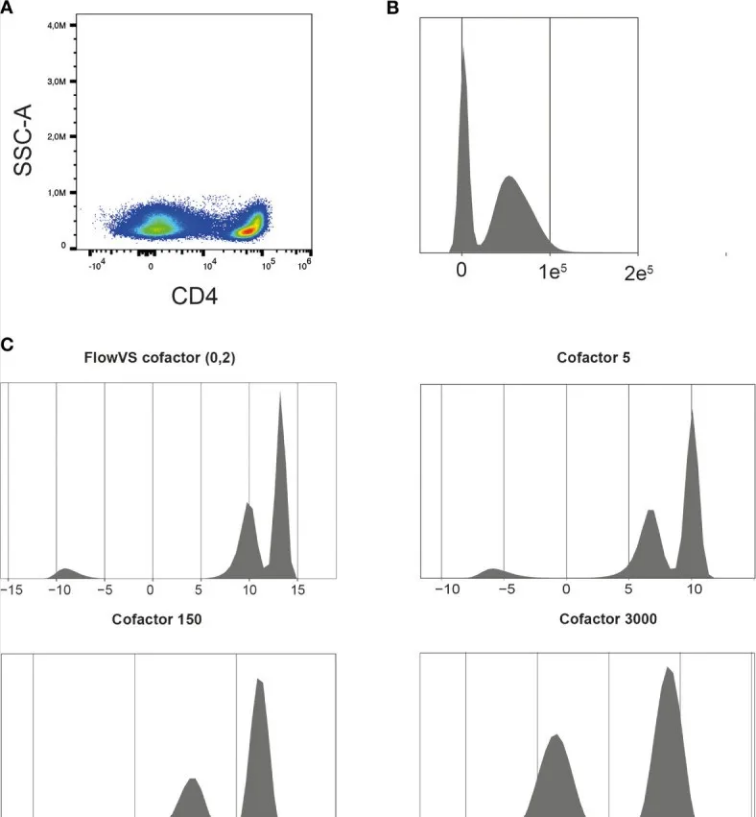
如同传统流式细胞术一般,光谱流式数据也会受到信号方差不均匀性的作用影响,即随着平均荧光强度的升高,荧光信号的方差也会增大。
通过数据转换能够使细胞群之间的这种差异趋于稳定,有助于对细胞群进行有效区分,进而提升聚类分析的结果质量。
FlowCore 软件包具备可应用不同转换方式的函数,例如双指数(bi - exponential)、Box - Cox 或者反正弦双曲(arcsinh)转换等,同时也可以尝试使用诸如 flowTrans 或者 FlowVS 等其他软件包,从而为数据探寻到最为适宜的转换方式。
生物学领域的专业知识能够为转换方式的选择以及数据的适应性调整提供有力的指导依据。
在流式细胞术数据的计算分析过程中,arcsinh 转换被广泛运用。
FlowVS 软件包能够针对每个荧光染料计算出合适的辅助因子,进而对数据开展 arcsinh 变换操作。
这里的辅助因子属于稳定群内方差的关键参数。
计算每个辅助因子往往需要耗费大量的时间,倘若只是需要对数据进行快速浏览查看,那么可以暂且采用固定的辅助因子。
一个理想的辅助因子应当能够产生两个清晰可辨的峰形,其中阴性峰应处于零值的附近区域。
自动质检和清洁数据
在 Bioconductor 平台中,为清洁流式数据提供了多个不同的软件包,像 FlowAI、FlowClean 以及 PeacoQC 等。
FlowAI 软件包具备消除流速、信号采集以及异常值事件等相关异常情况的功能。它所运用的是基于广义极值学生偏离测试和二进制分割的算法,正因为采用了这样的算法,使得该软件包在处理数据时标准十分严格。而 FlowAI 的一大突出优点在于,它额外提供了一种交互式的方法来消除异常情况,这为使用者带来了很大的便利。
PeacoQC 软件包则是借助根据隔离树(IT)中细胞所处的位置以及平均绝对偏差距离(MAD)来制定的算法,实现对异常情况的消除。使用者能够在若干个样本上对相关参数进行细微的调整,如此一来,该软件包在大型数据集中使用时就会变得更加容易上手。PeacoQC 软件包的优势重点体现在对样品的处理方面,例如当样品中出现两个堵塞物干扰流速以及信号采集的情况时,它能够从中选择多个相互不相交的区域来进行异常去除操作,从而有效提升数据的质量。
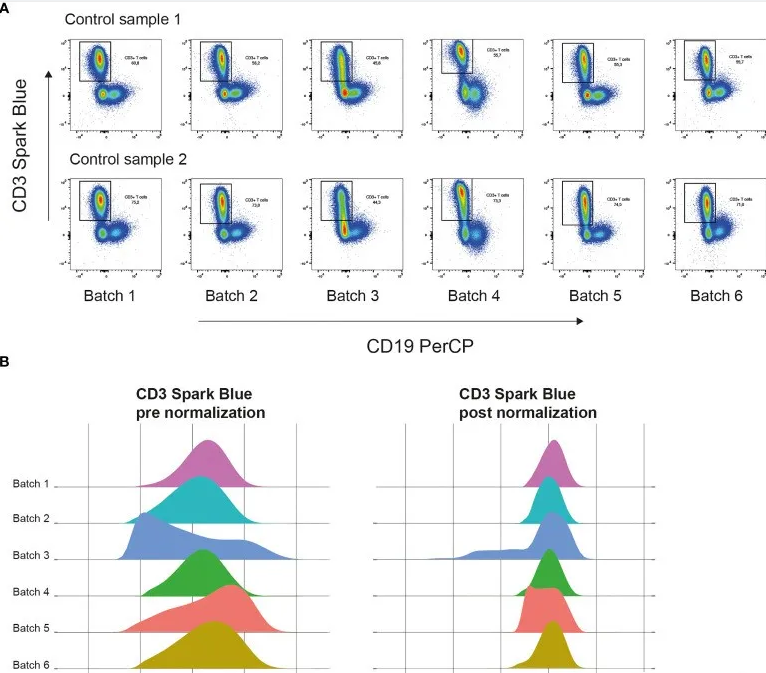
批次效应
无论是样品处理环节、染色程序步骤,还是仪器性能表现,均有可能引发批次之间存在的技术差异。针对多个批次里同一个对照样品的全部标记物构建密度图,这对于精准识别那些对批次效应较为敏感的标记物而言极具帮助。
CytoNorm 方法巧妙地借助对照样品数据以及验证样品数据来实现对批次效应的校正工作,从而确保信号变异性能够维持在一致的水平。在具体操作过程中,CytoNorm 方法会运用 FlowSOM 针对不同批次的对照样品开展聚类分析。由于在理想状态下,对照样品中所发现的细胞簇原本不应受到批次效应的干扰。所以,后续便能够计算出每个细胞簇中每个标记物的分位数,并对这些分位数进行对齐处理,以此完成批次效应的校正流程,保障数据在不同批次间的可比性与准确性,为后续深入的数据分析奠定坚实可靠的基础。
子采样(Subsampling)
一般而言,光谱流式实验往往会涉及众多标记物,进而采集到海量的数据,有时所涵盖的样品数量也颇为可观。为了能够更高效地在大型数据集中展开数据探索工作,较为明智的做法是在针对完整数据集进行深入分析之前,先对数据实施子采样处理,这样做的主要目的在于节省宝贵的计算时间以及内存资源。
针对子采样操作,可以采用两种不同的策略。其一为随机选取 n 个细胞(即随机下采样,random downsampling);其二是依据密度依赖性来挑选 n 个细胞(也就是密度依赖下采样,density - dependent downsampling)。在 R 脚本当中,已经包含了一个专门用于随机子采样的函数(Downsampling_Flowset)。当需要特意保留稀有细胞群的时候,密度依赖子采样则是更为理想的选择,因为它能够通过对主要细胞群执行欠采样操作,或者对次要细胞群开展过采样操作,从而有效地平衡细胞分布情况,使得数据在后续分析中能够更全面地反映各类细胞群体的特征。
在实际操作过程中,建议测试对不同数量的细胞进行聚类分析(例如 20,000 至 200,000 个细胞),这是由于部分研究成果表明样本量的大小可能会对聚类结果以及细胞群的发现产生极为关键的影响。另外一种可行的方式是,若要对所有样本均进行子采样处理,还可以首先将数据拆分成规模较小的部分(也叫做训练数据集)以及规模较大的测试集。在正式使用完整数据集之前,可以借助小型训练数据集来探寻最为适宜的聚类算法以及确定合适的聚类数量。在本文所阐述的工作流程脚本(Subsampling_Flowset)里,就包含了一个用于拆分数据的函数。当利用子采样数据完成数据探索工作之后,如果要使用全部数据来生成最终的分析结果,那么此时就需要借助性能强大的计算机硬件资源,以确保能够顺利完成复杂且大规模的数据处理任务,从而获得精准且具有深度的光谱流式数据分析结论,为相关研究领域提供有力的数据支持与理论依据。
通过聚类和降维探索数据
在光谱流式数据分析领域,聚类算法与降维方法的种类颇为丰富多样。其中,FlowSOM 堪称速度较快且效果优异的聚类算法之一,因而在相关研究中得以广泛应用;而在降维方法方面,UMAP 和 tSNE 则是较为常用的手段。
在实际操作过程中,能够借助 R 语言中的不同软件包来具体实现这些算法功能。值得注意的是,在正式启动分析流程之前,强烈建议预先设置种子数,这主要是因为绝大多数算法都具有一定的随机性。在不同的运行过程中,极有可能产生不一样的结果。通过设置种子数这一操作,能够有效避免出现不同结果的情况,进而切实确保分析结果的可重复性。不过,从另一个角度来看,率先对种子数进行改变,对于探索算法结果的稳健性也有着积极的帮助作用。
具体而言,首先运用降维方法来对数据集中的不同细胞群展开探索工作。除了种子数之外,各类参数(例如 UMAP 中的邻居数量)均会对 UMAP 或者 tSNE 的最终结果产生影响。基于此,建议在子采样数据集中针对这些参数以及其他相关参数尝试采用不同的取值,以此来探寻最为理想的参数设置组合,从而为获得更为精准、可靠的光谱流式数据分析成果奠定坚实基础。
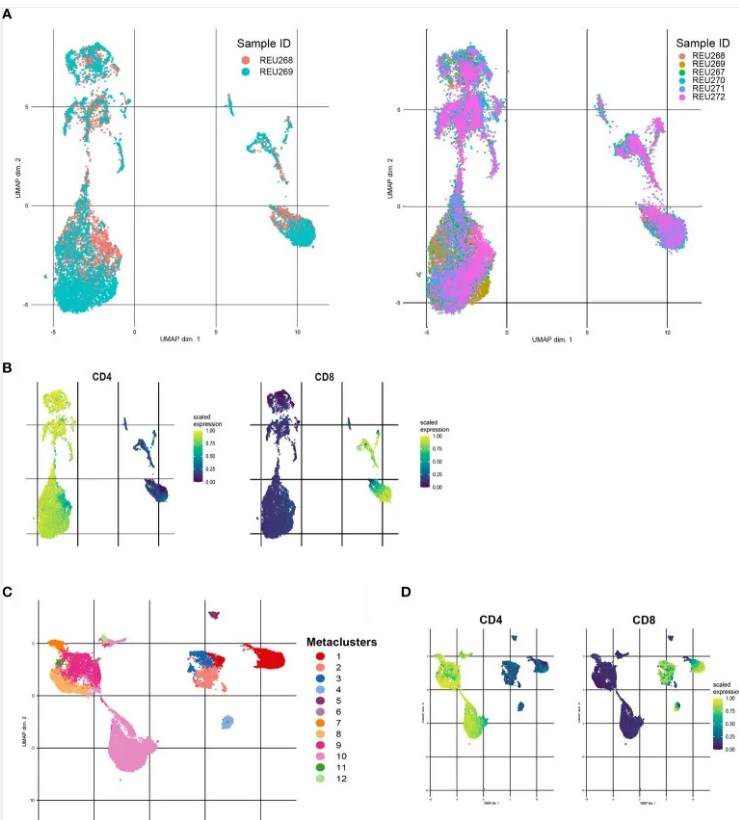
随后,运用诸如 FlowSOM 这类聚类方法,能够更进一步助力对数据进行解读,并且可以静态地测试不同组之间所存在的差异,比如健康对照组与患者组或者患者各亚组之间的差异情况。CATALYST 软件包具备相应功能,它既能利用 FlowSOM 开展聚类操作,又能借助 Consensus ClusterPlus 软件包对数据进行初次聚类,同时还可在 UMAP 或 tSNE 所构建的可视化环境中呈现元集群。而 diffcyt 包则有助于在不同组以及 / 或者元集群之间执行统计测试,以便从统计学角度更深入地分析数据特征及差异情况。
要确定出最为合适的聚类数往往颇具挑战性。增量面积图(delta area plot)以及弯头图(elbow plots)这两种数学方法可用于确定最佳聚类数。在实际运用聚类分析方法之前,率先采用无监督的 UMAP 或 tSNE 对数据加以探索,同样有助于锁定恰当的聚类数,为后续更精准的聚类分析做好铺垫。
除了借助 R 语言来实现聚类和降维操作之外,还可以选用 Cytosplore 这款软件。Cytosplore 属于交互式软件,使用时无需依赖 R 语言环境。它提供了多种不同的方法,像 SPADE 聚类算法、近似 t - SNE(这是标准 tSNE 的一种更快版本)以及分层 SNE(hSNE)等。并且,它能够生成展示每个集群以及每个组中位表达情况的热图,方便直观查看数据特征。此外,数据或者单独的簇还可以被导出,然后利用 Cytofast 包在 R 语言环境中做进一步的处理。
值得一提的是,还可以使用 Spectre R 包,该软件包为 R 语言环境提供了端到端的工作流程,它能够很好地适配光谱流式细胞术数据,同时还提供了各式各样的聚类和降维方法,为光谱流式数据分析提供了多样化且便捷高效的途径,助力科研人员从不同角度、运用不同方法对数据进行深度剖析,进而获取更有价值的研究成果。
上海优宁维生物科技股份有限公司
试剂 | 耗材 | 仪器 | 软件 | 定制 | 实验服务 | 供应链
免费热线:4008-168-068
咨询邮箱:info@univ-bio.com
订购商城:www.univ-bio.com
微信公众平台:优宁维抗体专家,欢迎关注!
小优博士(小程序):5大课堂, 让你的科研不再难!


相关新闻资讯